单细胞空间多组学揭示肿瘤相关成纤维细胞的保守空间亚型和细胞邻域-空间细胞亚群细分代码实现
背景介绍
CAF 是肿瘤微环境中的关键成分,能够通过细胞间相互作用影响免疫逃逸、药物耐受和转移。然而,CAF 的空间异质性与其生物功能尚未完全理解。癌症相关成纤维细胞(CAF)在肿瘤微环境(TME)中发挥着关键作用。2025年3月27日,Cancer Cell上在线发表了题为Conserved spatial subtypes and cellular neighborhoods of cancer-associated fibroblasts revealed by single-cell spatial multi-omics的最新研究成果,该研究通过整合来自10种癌症、7个空间转录组及蛋白组学平台的超过1400万个细胞的单细胞空间多组学数据中的肿瘤相关成纤维细胞(CAFs),成功识别出四种具有不同空间分布、细胞组成和功能特征的CAFs亚型。这些亚型在不同癌症类型中普遍存在,并且与肿瘤微环境特性、免疫细胞浸润及患者生存期密切相关。为理解CAFs在肿瘤中的作用以及开发针对CAFs的治疗策略提供了重要依据。
四种空间CAF亚型的鉴定
数据来源与样本概况
利用CosMx和MERSCOPE公开数据集,共24张覆盖NSCLC、乳腺、结直肠、前列腺、子宫、卵巢、肝脏、黑色素瘤等8种癌组织的大型切片,总计570余万个高质量细胞。
空间模式提取与亚型命名
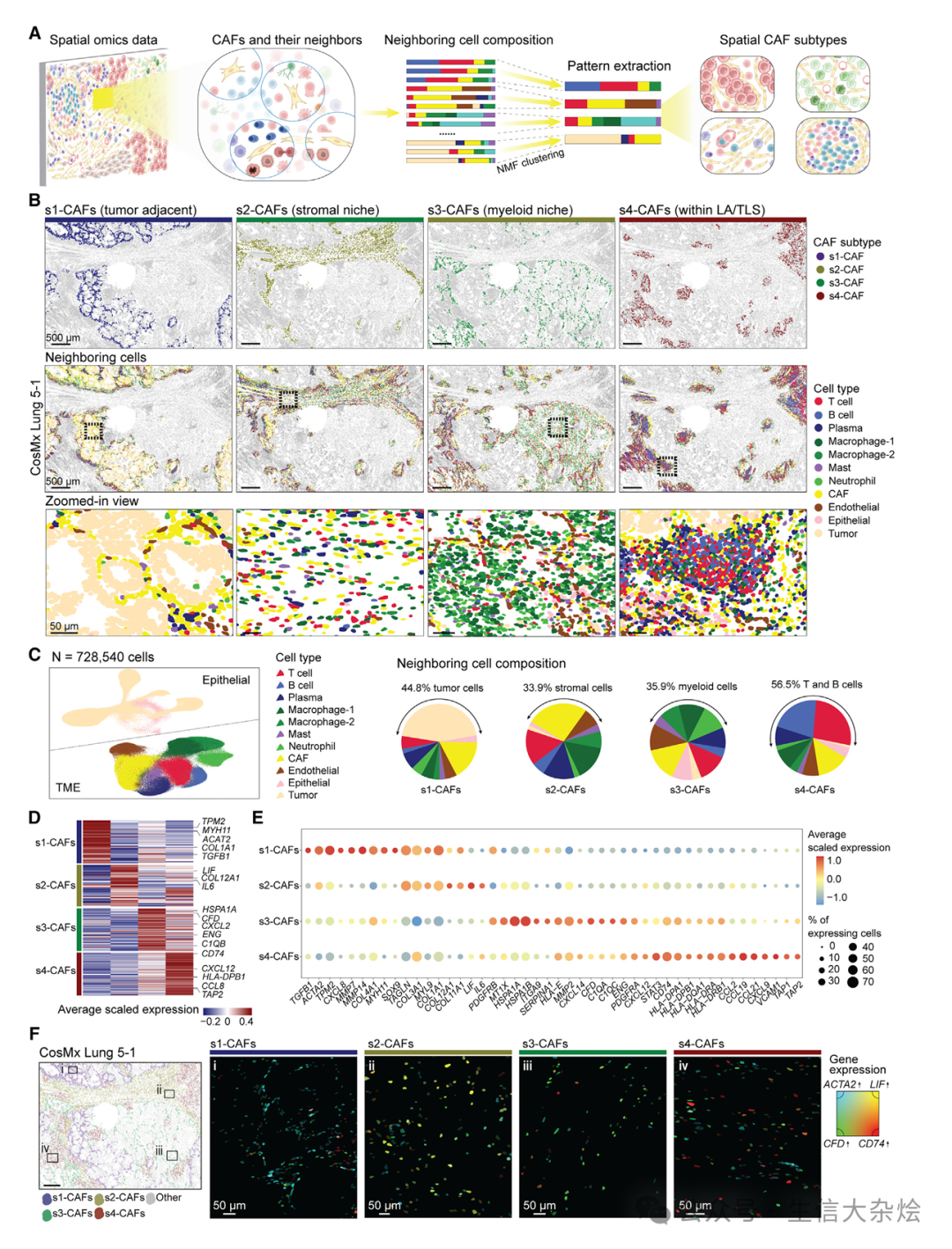
以80 µm半径定义CAF邻域,构建“邻域向量”。采用非负矩阵分解(NMF)提取空间模式,识别出四个空间CAF亚型:s1-CAF、s2-CAF、s3-CAF、s4-CAF。
各亚型的空间分布特征
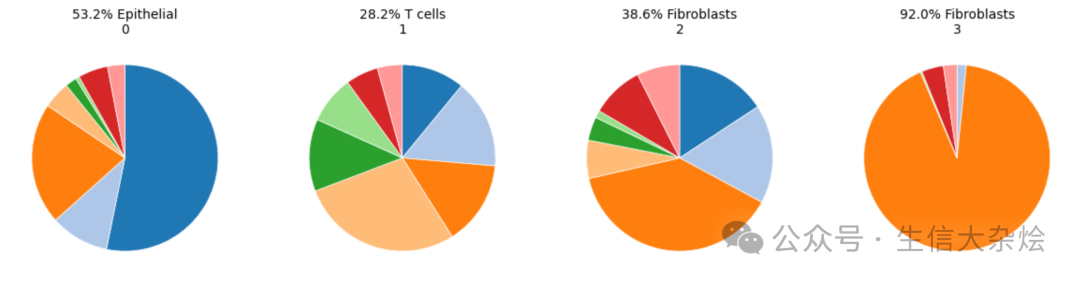
s1-CAF:紧邻癌细胞,44.8%邻域为癌细胞。
s2-CAF:位于基质区,33.9%邻域为基质细胞。
s3-CAF:靠近血管的髓系富集基质微环境,35.9%邻域为髓系细胞。
s4-CAF:与T、B细胞共定位于淋巴聚集体(LAs)或三级淋巴结构(TLSs),56.5%邻域为B/T细胞。
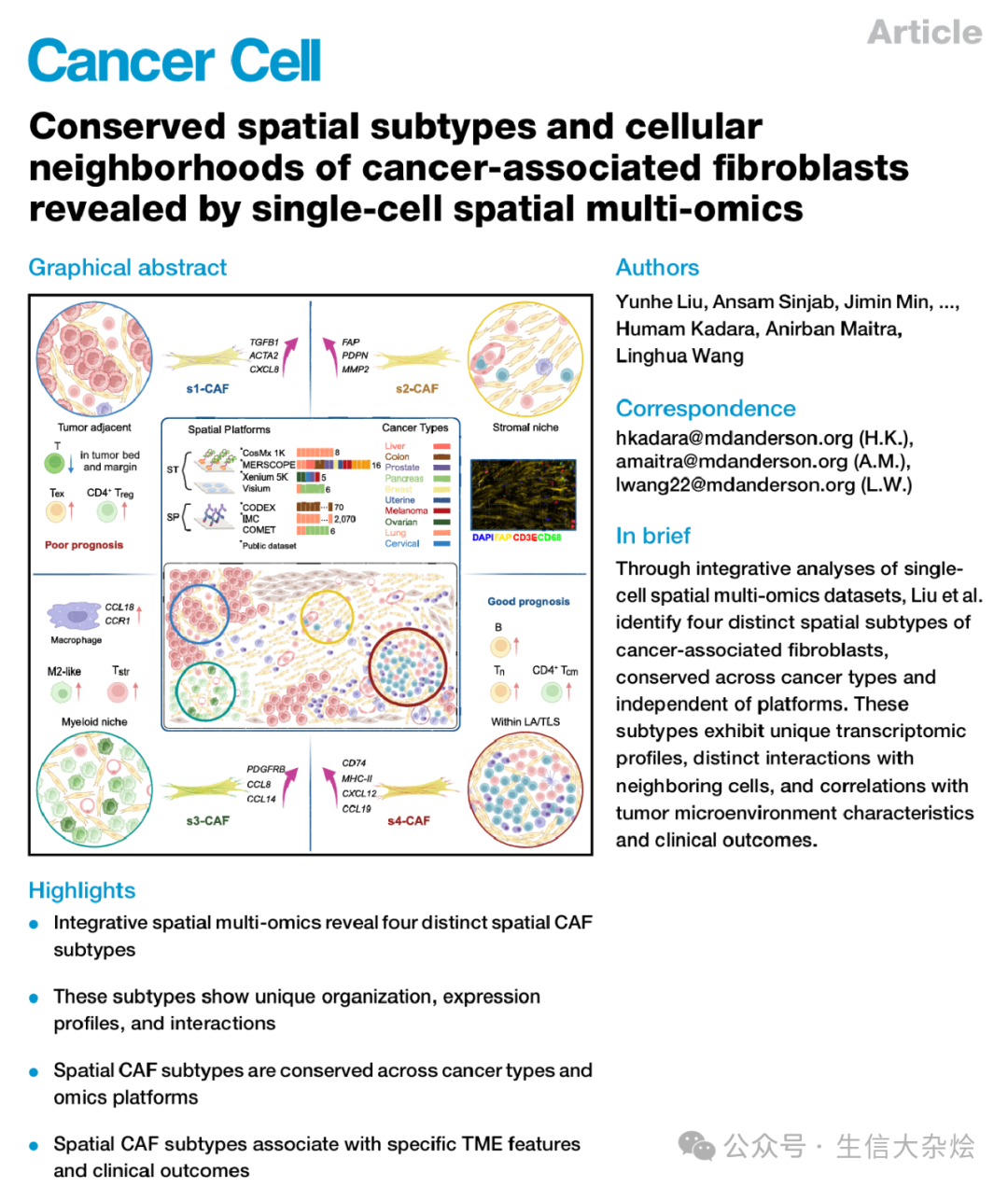
CAF空间亚型识别流程

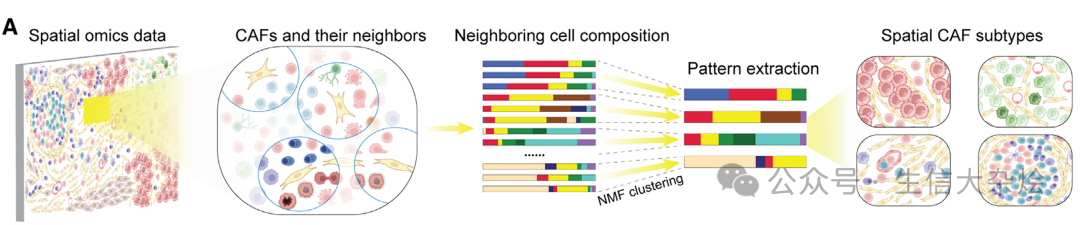
步骤一:定义邻域构成向量
以 CAF 为中心,设定一定半径(如 80μm),统计其邻域细胞类型比例,构建 CAF × cell type 的矩阵
步骤二:使用非负矩阵分解(NMF)

将邻域构成矩阵分解为:
CAF × factor 的权重矩阵
factor × cell type 的细胞生态位成分
步骤三:基于 CAF × factor 矩阵进行聚类
使用 Leiden 聚类算法识别 CAF 空间亚型(CAF subtype)
代码实现
import numpy as np
import pandas as pd
import scanpy as sc
from sklearn.neighbors import KDTree
from sklearn.decomposition import NMF
from sklearn.neighbors import NearestNeighbors
import matplotlib.pyplot as plt# 函数封装
def identify_spatial_subtypes(adata, celltype_name='Fibroblasts', celltype_col='celltype', sample_col='sample', radius=80, n_components=5, resolution=0.1):"""step1: 对每个celltype_name细胞计算其radius范围内邻域细胞类型构成,返回一个 cell × cell-type 比例的 DataFrame。"""all_vecs = []all_indices = []for sample in adata.obs[sample_col].unique():adata_sample = adata[adata.obs[sample_col] == sample, :]coords = adata_sample.obsm['spatial']tree = KDTree(coords)caf_mask = adata_sample.obs[celltype_col] == celltype_namecaf_indices = np.where(caf_mask)[0]caf_coords = coords[caf_mask]for i, idx in enumerate(caf_indices):center = coords[idx]ind = tree.query_radius(center.reshape(1, -1), r=radius)[0]neighbor_types = adata_sample.obs.iloc[ind][celltype_col].value_counts(normalize=True)vec = pd.Series(0, index=adata.obs[celltype_col].unique())vec[neighbor_types.index] = neighbor_types.valuesall_vecs.append(vec)all_indices.append(adata_sample.obs_names[idx])neighbor_matrix = pd.DataFrame(all_vecs, index=all_indices).fillna(0)# print(neighbor_matrix)"""step2: NMF分解"""model = NMF(n_components=n_components, init='nndsvda', random_state=0)W = model.fit_transform(neighbor_matrix.values) # cell × factorH = model.components_ # factor × cell-typeW_df = pd.DataFrame(W, index=neighbor_matrix.index, columns=[f'Factor_{i}' for i in range(n_components)])H_df = pd.DataFrame(H, columns=neighbor_matrix.columns, index=[f'Factor_{i}' for i in range(n_components)])# print("W_df", W_df)# print("H_df", H_df)"""step3: 识别空间亚型"""caf_adata = adata[adata.obs_names.isin(W_df.index)].copy()caf_adata.obsm['X_nmf'] = W_df.loc[caf_adata.obs_names].valuessc.pp.neighbors(caf_adata, n_neighbors=20, use_rep='X_nmf', metric='cosine')sc.tl.leiden(caf_adata, resolution=resolution)# 写回主 AnnDataadata.obs.loc[caf_adata.obs_names, celltype_name+'_sub'] = caf_adata.obs['leiden'].astype(str)"""step4: 统计绘图"""subtypes = adata.obs.loc[neighbor_matrix.index, celltype_name+'_sub']neighbor_matrix_with_subtype = neighbor_matrix.copy()neighbor_matrix_with_subtype['subtype'] = subtypesavg_neighbor_composition = neighbor_matrix_with_subtype.groupby('subtype').mean()# print(avg_neighbor_composition)n_subtypes = avg_neighbor_composition.shape[0]fig, axes = plt.subplots(1, n_subtypes, figsize=(n_subtypes * 3, 3))# 自动生成颜色all_cell_types = avg_neighbor_composition.columnscmap = plt.get_cmap('tab20')cell_type_colors = {cell: cmap(i) for i, cell in enumerate(all_cell_types)}subtype_names = {i: f"s{i+1}-CAFs" for i in range(n_subtypes)}for i, (subtype, row) in enumerate(avg_neighbor_composition.iterrows()):ax = axes[i]values = row.valueslabels = row.indexcolors = [cell_type_colors[ct] for ct in labels]wedges, texts = ax.pie(values, colors=colors, startangle=90, counterclock=False,wedgeprops={'linewidth': 0.5, 'edgecolor': 'white'})dominant_type = labels[values.argmax()]percent = values.max() * 100ax.set_title(f"{percent:.1f}% {dominant_type}\n{subtype_names.get(subtype, str(subtype))}", fontsize=10)plt.tight_layout()return adata
4种空间成纤维细胞亚群
空间CAF亚型的转录组特征
差异表达基因(DEG)分析
s1-CAF:高表达胶原基因(COL1A1、COL3A1)、ACTA2、MMP7、TGFB1等,符合myCAF特征。
s2-CAF:高表达LIF、IL6,胶原基因中等,符合iCAF特征。
s3-CAF:高表达PDGFRA/B、CFD、CD74、HLA-II等,兼具iCAF与apCAF部分特征。
s4-CAF:高表达STAT3、CXCL9/19/21、HLA-II、IDO1,接近apCAF。
CAF邻域的细胞互作网络
配体-受体互作推断
s1-CAF与癌细胞:通过胶原-整合素、THBS1-CD47、TGFB1-TGFBR1、FGF2-FGFR1等通路互作。
s2-CAF与内皮细胞及T细胞:通过胶原-整合素、CD55-ADGRE5等互作。
s3-CAF与髓系细胞:通过RARRES2-CMKLR1、GAS6-MERTK、DLL1-NOTCH3等通路。
s4-CAF与B/T细胞:通过CCL19/21-CCR7、VCAN-SELL、JAG1-NOTCH等通路。
CAF邻域内免疫细胞状态与分布
T细胞状态
s1邻域:CD4+Treg、CD8+Teff、CD8+Tex富集。
s2邻域:Tstr升高。
s3邻域:Tstr显著升高。
s4邻域:Tn、Tfh、Tcm富集。
巨噬细胞状态
s1邻域:高表达IL15RA、IL32。
s3邻域:高表达CCR1、CCL18、CCL3/3L3及M2标记CD163、CD209等。
