泛英国生物样本库全基因组关联分析

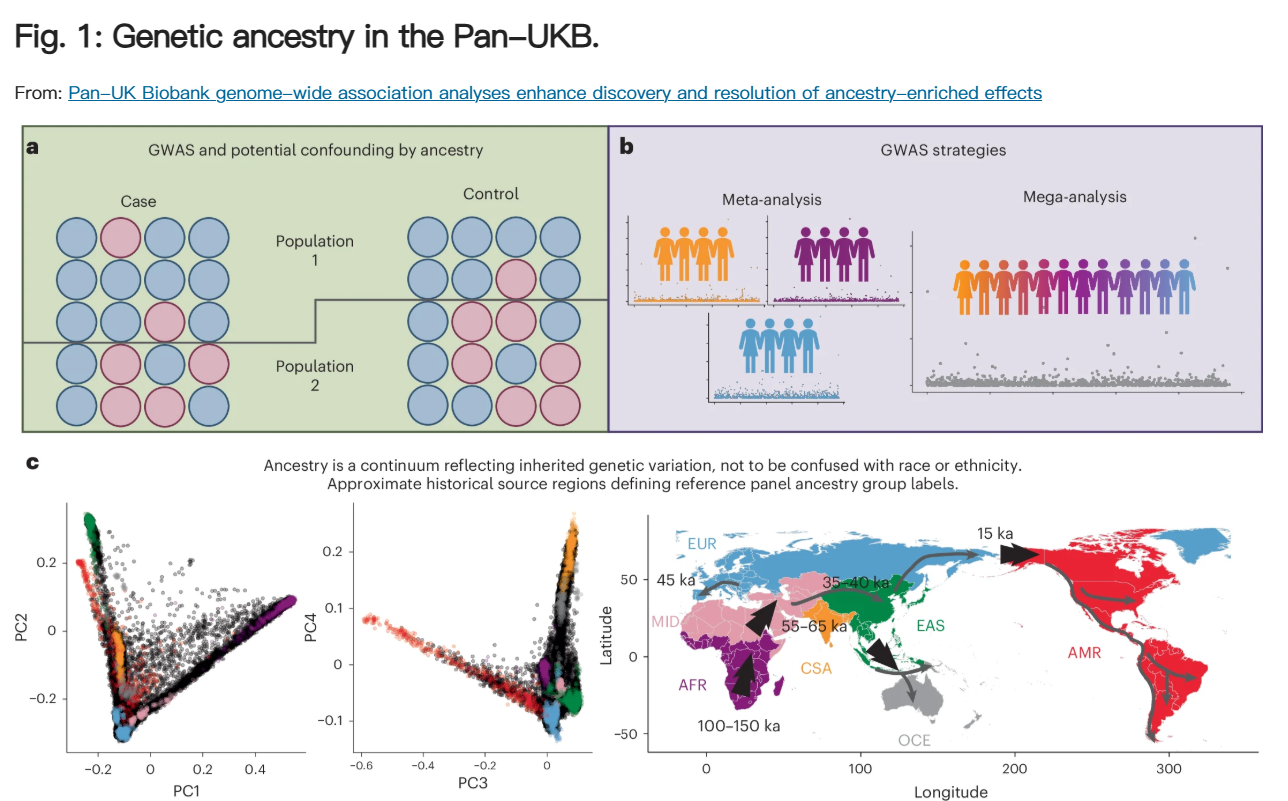
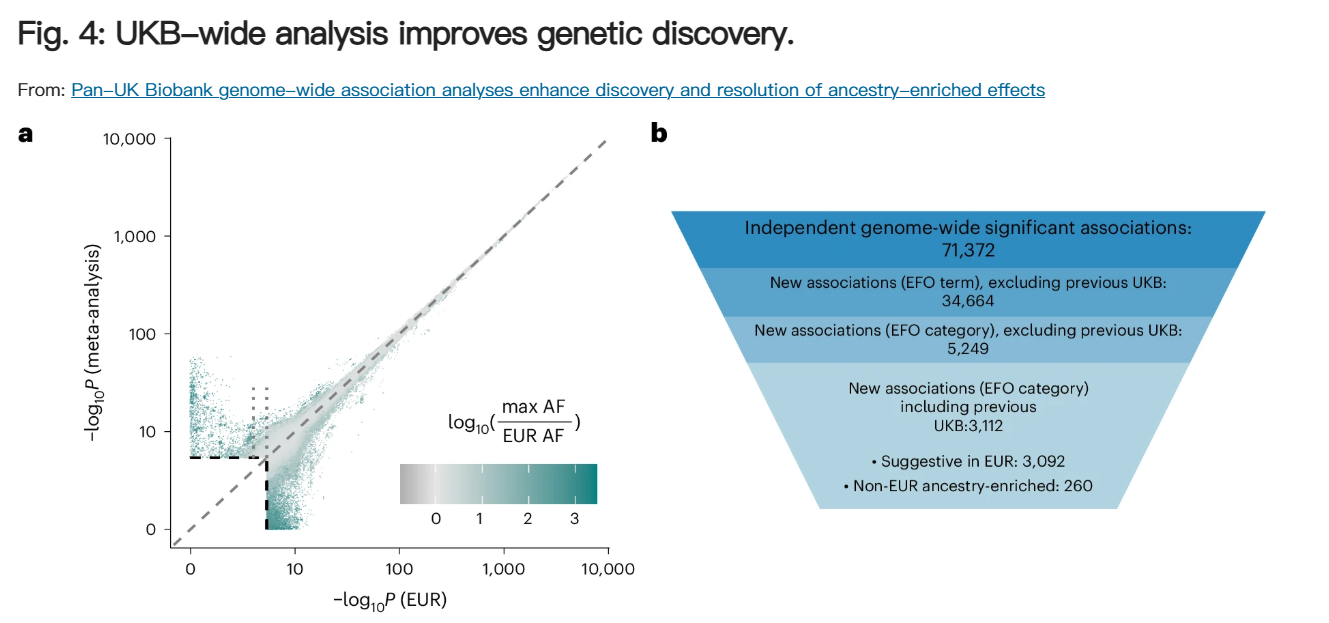
由于担心人口结构引入假阳性关联,以前UKB研究中来自不同遗传血统群体的人经常被排除在关联分析之外。该研究进行了跨遗传血统组的混合模型关联和荟萃分析,包括比之前工作更大的英国生物样本库比例,以生成 7,266 个性状的汇总统计数据。建立了一个以遗传结构为依据的质量控制和分析框架。总体而言,确定了 14,676 个显著位点 (P < 5 × 10−8)在荟萃分析中,这些因素仅在 EUR 遗传血统组中未发现,包括新的关联,例如 CAMK2D 和甘油三酯之间。研究还强调了与血统富集变异的关联,包括与几种生物标志物特征相关的 G6PD 中已知的多效性错义变异。公开发布这些结果以及描述解释结果的注意事项的常见问题,从而增强了解释不同人群的风险变异的可用资源。
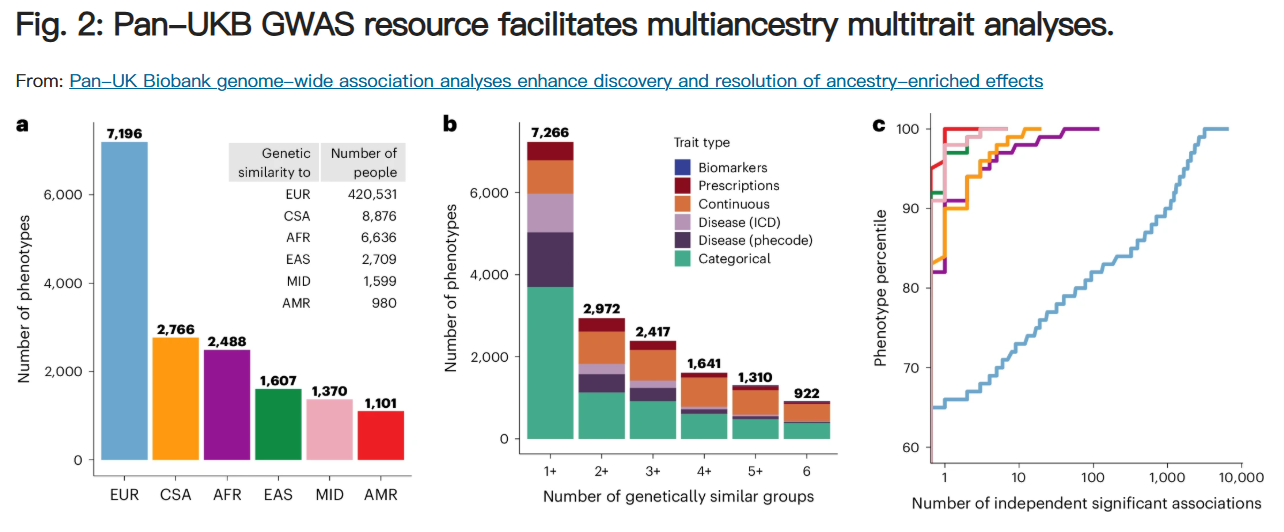
由于 EUR 的样本量要大得多,我们发现 EUR 中每个表型存在更多全基因组的显着关联:25% 的表型在 EUR 中具有 >18 个相关位点,而在所有非 EUR 人群中,只有不到 10% 的表型具有三个或更多相关位。
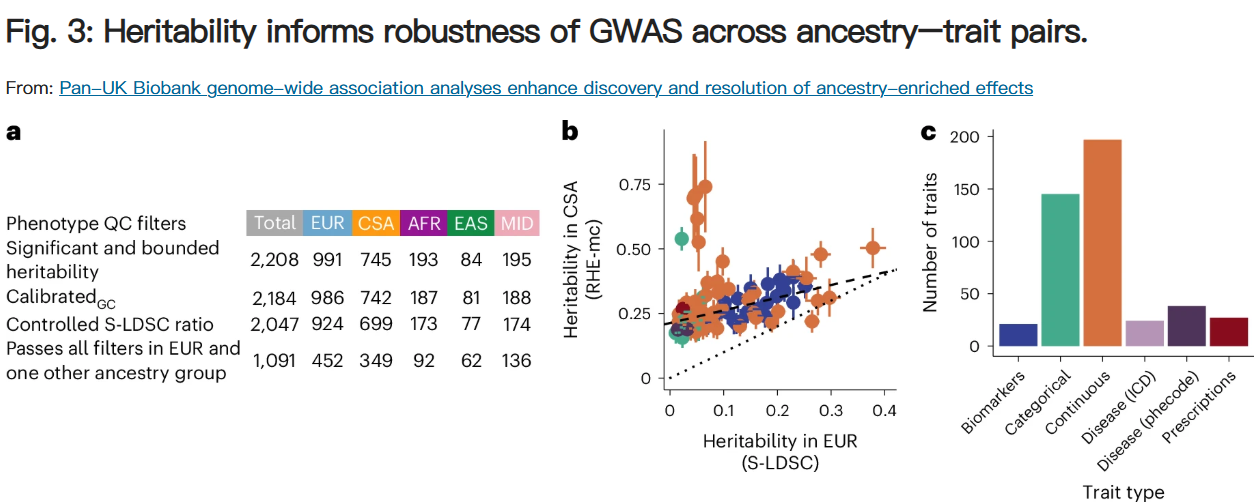
为了提高跨性状和祖先分析的可信度,设计了一种 QC 策略,系统地标记具有潜在问题 GWAS 结果的性状,同时保留两个或多个群体中通过 QC 的遗传性状的 GWAS,并确定了种群之间遗传力估计的相对幅度的一致性。
对于 452 种高质量表型,将多血统荟萃分析的 P 值与 EUR GWAS 的 P 值进行了比较,后者总体上高度相关。
可用资源
https://pan.ukbb.broadinstitute.org/
英国生物样本库的泛祖先遗传分析网站,贡献自马萨诸塞州总医院分析与转化遗传学部门 (ATGU) 以及麻省理工学院和哈佛大学布罗德研究所的一组研究人员。

https://aws.amazon.com/marketplace/pp/prodview-2efssfw2ezyq6#resources
亚马逊云上有同样的数据存储和分享。
研究意义
虽然以前的遗传学研究已经为许多表型的分子基础提供了深入的见解,但这些研究的参与者大多由祖先追溯到欧洲地区的人群组成。虽然所有人的遗传相似性多于差异,但某些遗传变异或变异组合在祖先可追溯到附近地区的群体中更为常见。因此,大多数先前的研究最适合了解在这些欧洲地区人群中更常见的遗传变异的作用。扩大遗传学研究以包括具有不同血统的个体将提高我们对每个人对这些表型的理解。例如,更多的多样性将帮助研究人员确定哪些遗传变异实际上是因果的,哪些只是简单地相关。它还将帮助研究人员发现新的生物学机制,因为某些遗传变异仅在某些人群中普遍到足以研究。发现这些生物学机制将有助于我们更好地了解每个人共享的重要表型的潜在生物学。此外,研究代表性不足的群体可能会使这些目前服务不足的人群本身获得精准医疗成为可能。
