【文献分享】Cell Decode:利用多尺度可解释深度学习进行细胞身份解码
文章目录
- 介绍
- 代码
- 参考
介绍
细胞是生命的基本结构和功能单位[1]。不同组织和器官的复杂功能都源于细胞的组成,研究各种细胞的组织和功能有助于理解生物体如何实现正常的生命功能。根据结构和功能对细胞类型进行定义已有数百年历史。通常,相同类型的细胞具有相似的特征和功能。对细胞进行分类和标注能够极大地有助于理解其组织和功能[2]。单细胞转录组技术在生物研究中的广泛应用极大地推进了细胞类型的研究[3,4,5,6]。然而,在多个尺度上不同细胞类型所表现出的各异特性给精确的细胞类型定义和标注带来了巨大挑战。
在单细胞转录组学数据中对细胞类型的识别通常需要经过多个步骤。这包括对转录组数据的预处理、降维以及无监督聚类。随后,会根据人工筛选出的差异表达标志基因来进行类别标注[7, 8]。基于传统方法进行的细胞类型识别既耗时又费力,而且标志基因的选择很大程度上依赖于研究人员的专业知识,这种知识是基于经验的,且容易受到偏差的影响。随着已标注的单细胞转录组学数据的积累为细胞类型识别提供了大量的参考数据集,一些具有代表性的深度学习模型,包括全连接神经网络、自编码器和转换器,被用于将参考数据集映射和迁移至新的数据集,以进行细胞类型识别[9,10,11,12]。尽管上述方法在不同的数据集上都取得了令人满意的性能,但它们的“黑箱”性质使得它们难以解释。模型学习过程的本质与人类的推理方式有着显著的不同,这使得很难理解深度学习模型是如何从单细胞数据中进行学习的[13]。然而,对于生物学研究而言,模型的透明度与其准确性同样重要。对模型运作机制的清晰理解对于解读其研究结果的生物学意义是必不可少的。
机器学习领域的一个重要趋势是发展可解释的深度学习方法(XAI)[14]。例如,在药物反应预测、肿瘤分类和生物标志物发现等方面将领域知识融入模型中[15,16,17,18,19]。然而,这些方法并未充分利用生物学领域的知识,特别是蛋白质之间的相互作用以及生物途径之间的相互依存关系。因此,构建一个用于细胞类型识别的多尺度可解释模型仍然是一个挑战。
在此,我们提出了一种可解释的深度学习模型——“细胞解码器”,它将多尺度的生物学知识嵌入到图神经网络中,能够解码不同的细胞身份特征。细胞解码器基于基因之间的相互作用、基因与通路之间的映射关系以及层级化的通路信息构建了一个分层的图结构。通过应用自动机器学习技术,该模型的表示能力得到了增强,从而能够实现精确且可靠的细胞类型识别以及多尺度数据整合。此外,我们还开发了一种多视角的属性解释方法,阐明了模型的学习和决策过程,并将其与生物学解释相联系。细胞解码器有助于理解区分不同细胞类型的相互作用、通路和生物学过程,为更深入地探索细胞身份和功能提供了重要启示。
Cells are the basic structural and functional units of life [1]. The complex functions of different tissues and organs are rooted in cellular composition, and studying the organization and function of various cells can help understand how organisms achieve normal life functions. Cell types have been defined according to their structures and functions for centuries. Usually, the same kind of cells exhibit similar characteristics and functions. Classifying and annotating cells can significantly aid in understanding their organization and functions [2]. The increasing application of single-cell transcriptomic technologies in biological research has greatly advanced the study of cell types [3,4,5,6]. However, the varying properties exhibited in different cell types at multiple scales present substantial challenges for precise cell-type definition and annotation.
The identification of cell types in single-cell transcriptomics data is usually reliant on a multi-step process. It includes the preprocessing of transcriptomics profiles, dimensionality reduction, and unsupervised clustering. Subsequently, category annotation is conducted based on manually curated differentially expressed marker genes [7, 8]. Cell identification based on the traditional approach is time-consuming and laborious, and the selection of marker genes heavily relies on researchers’ domain knowledge, which is empirical and easily biased. As the accumulation of annotated single-cell transcriptomics data provides a large number of reference datasets for cell type identification, some representative deep learning models, including fully connected neural networks, autoencoders, and transformers, are applied for mapping and migrating from reference datasets to new datasets for cell-type identification [9,10,11,12]. While the aforementioned methods have achieved commendable performances across different datasets, their “black box” nature renders them largely unexplainable. The essence of the model learning process and human reasoning is significantly different, making it difficult to understand how deep learning models learn [13] from single-cell data. However, for biological research, the transparency of the model is as important as its accuracy. A clear understanding of a model’s workings is indispensable for interpreting the biological significance of its findings.
A significant trend in machine learning is the development of explainable deep learning methods (XAI) [14]. For instance, the incorporation of domain knowledge into the model for drug response prediction, tumor typing, and biomarker discovery [15,16,17,18,19]. However, these methods do not fully leverage biological domain knowledge, particularly the interactions between proteins and the interdependencies among biological pathways. Therefore, constructing a multi-scale interpretable model for cell-type identification remains a challenge.
Here, we propose an interpretable deep learning model called Cell Decoder, which embeds multi-scale biological knowledge into the graph neural network, enabling the decoding of distinct cell identity features. Cell Decoder constructs a hierarchical graph structure based on the interactions between genes, the mapping relationships between genes and pathways, and the hierarchical pathway information. Through the application of automated machine learning techniques, the model’s representation power is enhanced, facilitating precise and robust cell-type identification and multi-scale data integration. Moreover, we have developed a multi-view attribution interpretation method, elucidating the model’s learning and decision-making processes and mapping them to biological explanations. Cell Decoder facilitates the understanding of the interactions, pathways, and biological processes that distinguish different cell types, providing significant implications for deeper exploration of cell identity and function.
代码

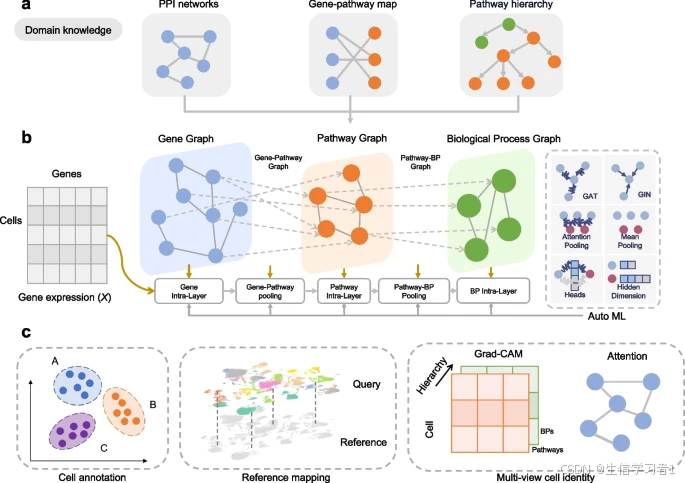
细胞解码器概述。细胞解码器充分利用了细胞类型识别中的生物学领域知识,包括蛋白质-蛋白质相互作用网络、基因途径图以及途径层次关系。这些网络以图(邻接矩阵)的形式呈现,作为细胞解码器的输入。b 此外,细胞解码器还将基因表达作为每个细胞的特征。细胞解码器采用内部尺度和外部尺度的消息传递层来整合不同尺度的信息,从而获得基因、途径和生物过程的表示。具体的层以及其他超参数和架构修改是使用自动机器学习技术自动设计的,以适应各种细胞类型识别场景。GAT:图注意力网络;GIN:图同构网络。c 细胞解码器的输出可用于细胞标注、参考映射,并包含事后可解释性模块,以提供可解释的多视图细胞身份。Grad-CAM:梯度加权类激活映射。
https://github.com/PHOENIXcenter/CellDecoder
参考
- Cell Decoder: decoding cell identity with multi-scale explainable deep learning
- https://github.com/PHOENIXcenter/CellDecoder
