两个RNA-蛋白以及蛋白间相互作用数据库
HuRPA:PRIM-seq技术生成的人类RPA参考网络
文章由华人学者团队上个月发表在自然生物技术,开发了RIM-seq(通过测序进行蛋白质-RNA 相互作用图谱),一种同时从头鉴定 RNA 结合蛋白及其相关 RNA 的方法。通过将 RNA 与蛋白质连接的 DNA 条形码相近连接来生成独特的嵌合 DNA 序列,随后通过测序对其进行解码。应用于两种人细胞系,构建人 RNA-蛋白质关联网络 (HuRPA),包括 >350,000 个关联,涉及 ~7,000 个 RNA 和 ~11,000 个蛋白质,包括 2,610 个蛋白质,每个蛋白质与至少 10 个不同的 RNA 相互作用。并通过实验验证了肿瘤发生相关的lincRNA LINC00339,即HuRPA中蛋白质结合数量最多的RNA,作为蛋白质相关RNA。进一步验证了染色质构象调节因子 SMC1A、SMC3 和 RAD21 以及代谢酶 PHGDH 的 RNA 关联能力。PRIM-seq 无需基因或蛋白质特异性试剂即可系统地发现 RNA 结合蛋白及其靶标并确定其优先级。

RNA-蛋白质相互作用对基因表达和细胞过程进行关键调节,但由于其结构多样性,其全面定位仍然具有挑战性。HuRPA官网是:https://sysbiocomp.ucsd.edu/prim/
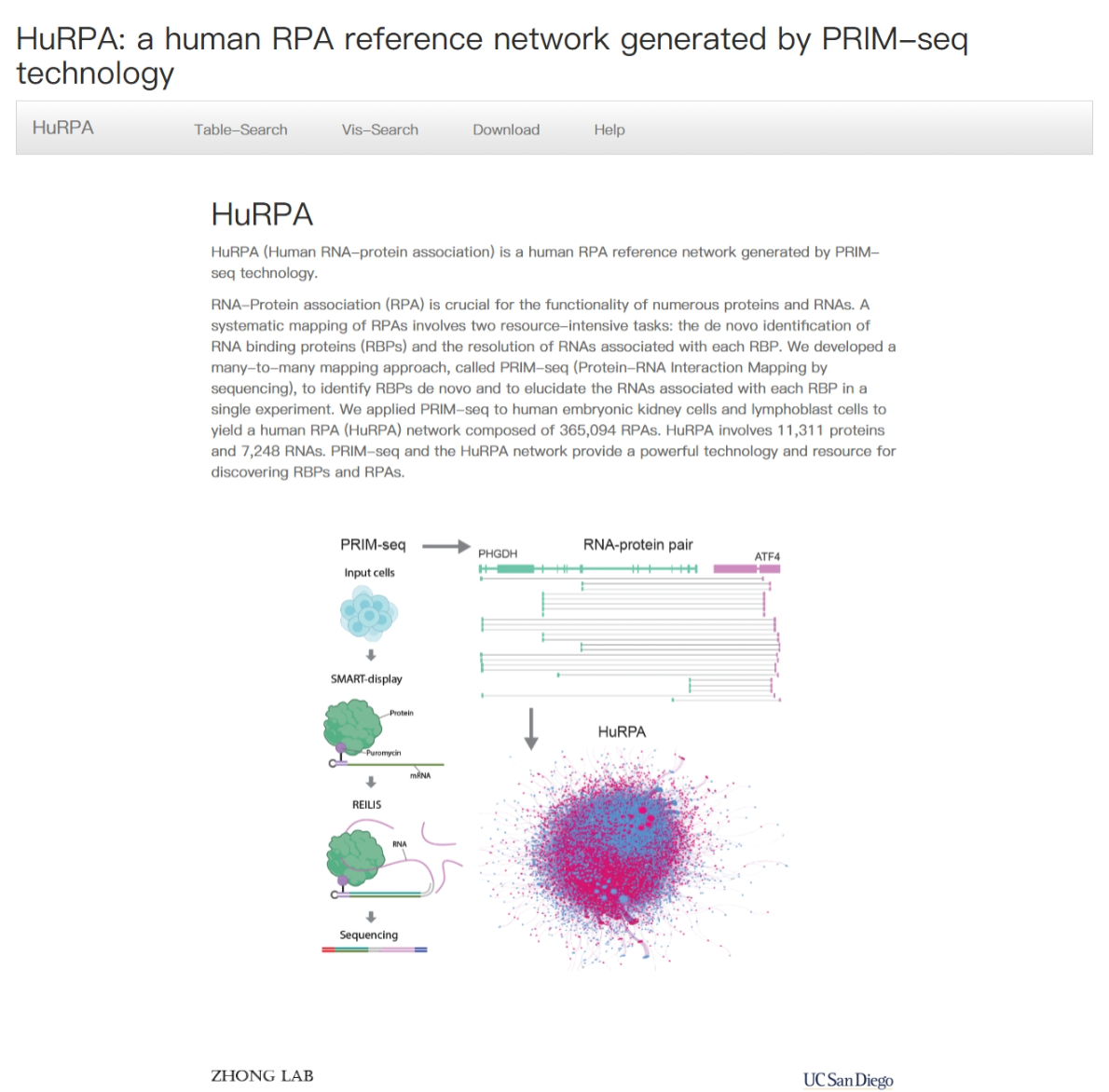
有两种数据查询方法:
- 表搜索:查询数据表中的基因。
- Vis-Search:通过网络可视化查询基因。下图是一个示例,查看10余个基因的网络。
Vis-Search 应用程序受查询子网络大小的限制。当 查询结果是具有超过 10,000 条边的网络,Vis-Search 方法 将被终止。请少输入查询基因或使用表格搜索方法。
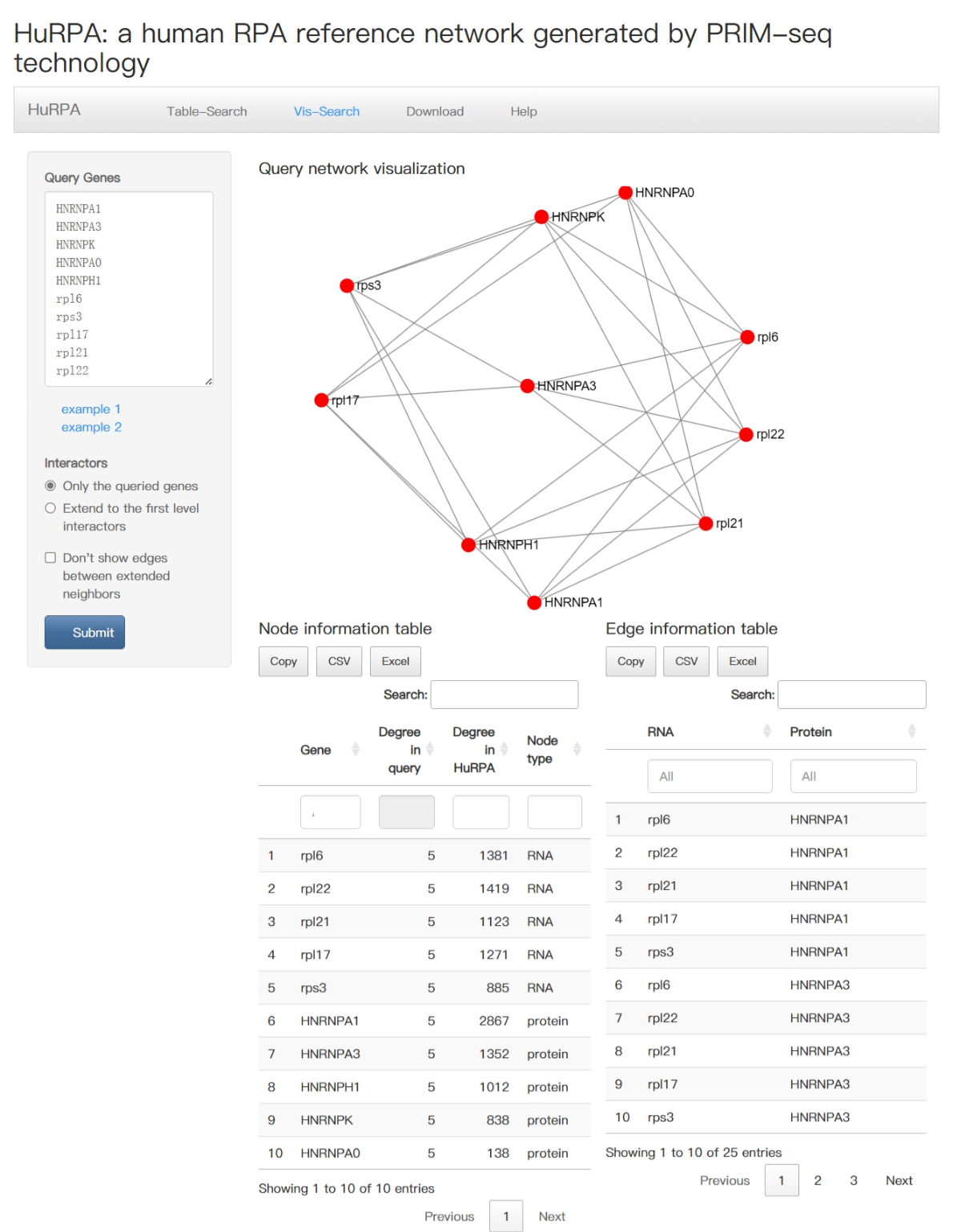
数据库可供下载,下载的PRIM v1.0 数据文件是 csv 格式的所有边的简单表格。有 6 列:
RNA:相互作用RNA的基因符号
蛋白质:相互作用蛋白质的基因符号
读取计数:PRIM-seq 库中映射的读取对数
BH 校正的 p 值:Benjamini-Hochberg 校正的 p 值源自从 PRIM-seq 库中调用 RPA 的卡方检验
Human PPI:预测人类蛋白质组中的蛋白质-蛋白质相互作用
蛋白质-蛋白质相互作用 (PPI) 对于生物学功能至关重要。基于协同进化分析和基于深度学习 (DL) 的蛋白质结构预测已经实现了细菌和酵母中的 PPI 综合鉴定,但这些方法对于更复杂的人类蛋白质组的成功有限。作者通过从 30 PB 的未组装基因组数据中收集的 7 倍深的多序列比对来增强共同进化信号,并开发一个新的 DL 网络,该网络在 2 亿个预测蛋白质结构的域间相互作用的增强数据集上进行训练。系统地筛选了 2 亿对人类蛋白质对,并预测了 17,849 次相互作用,预期精度为 90%,其中 3,631 次相互作用在之前的实验筛选中未被识别出来。这些预测相互作用的三维模型提供了许多关于蛋白质功能和人类疾病机制的假设。
在网站上,展示了在 PPI 筛选流程的最后阶段评估的 340 万个候选蛋白质-蛋白质相互作用 (PPI) 的结果。请注意,这些候选 PPI 中只有一小部分 (5-10%) 可能是正确的。有关作者论文中最有信心的 29,257 个预测,请查看最终预测选项卡。
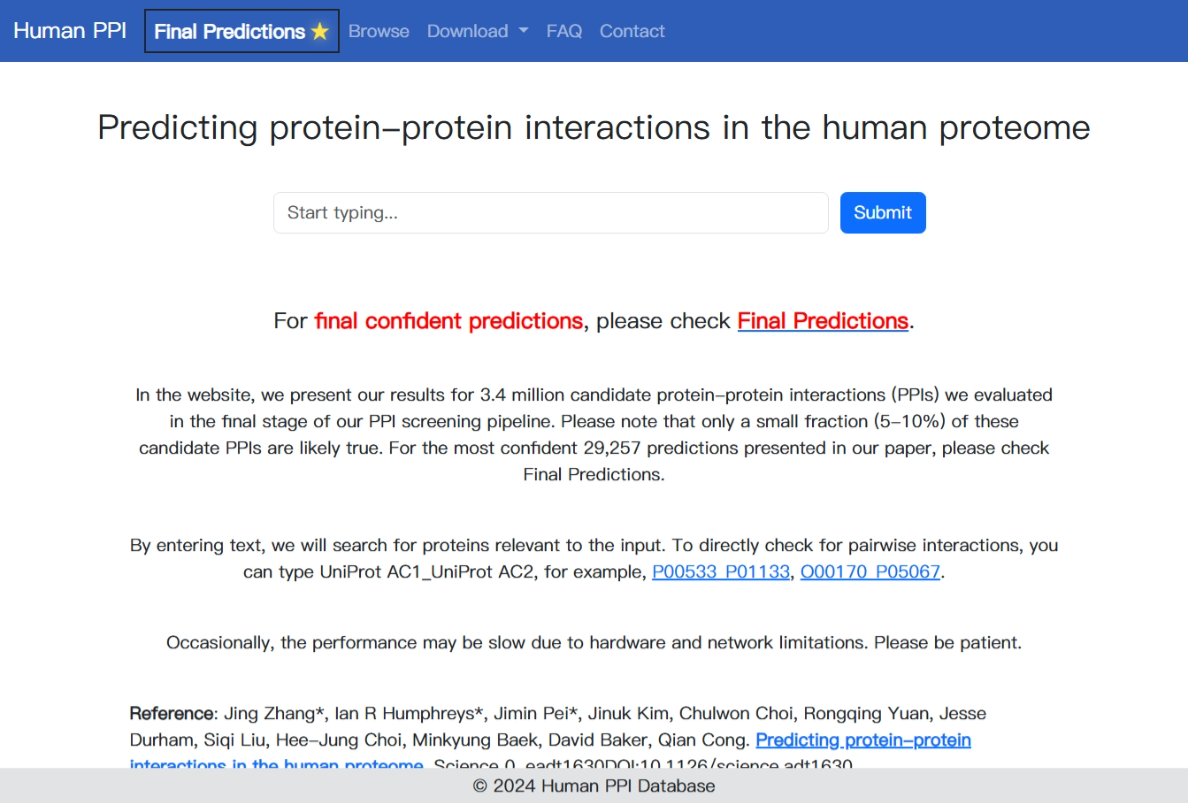
同样提供了数据下载选项,
参考:
- Qi, Z., Xue, S., Chen, J. et al. Genome-wide mapping of RNA-protein associations through sequencing. Nat Biotechnol (2025). https://doi.org/10.1038/s41587-025-02780-z
- https://www.science.org/doi/10.1126/science.adt1630-
- http://prodata.swmed.edu/humanPPI/
