scTenifoldKnk:“虚拟敲除基因”,查看转录组其他基因的变化幅度(升高or降低)
本期和大家分享一个强大的生信工具——scTenifoldKnk。它可以帮助我们对任意转录组数据中的某一或某一组基因进行虚拟敲除,从而估算出转录组中其他基因的变化情况:如上调或下调,改变幅度等。十分适合选题前的预实验,或者辅助验证/说明一些实验结果。
01 在python中安装
pip install scTenifoldpy02 使用示例
① 导入模块
from scTenifold.data import get_test_df
from scTenifold import scTenifoldKnk② 准备转录组数据
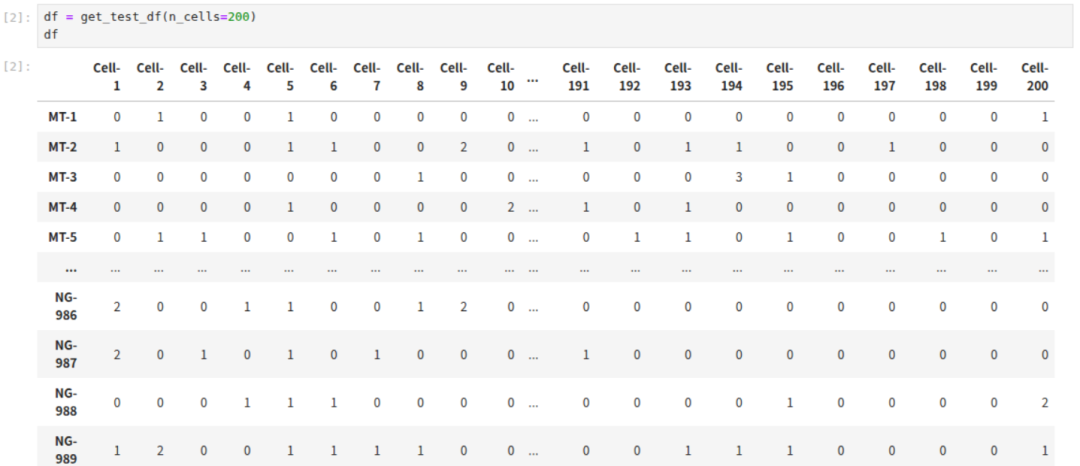
数据形式应该整理为(基因 x 细胞)的形式。这里我使用示例数据随机选取了200个细胞。数据形式如下:
③ 执行虚拟敲除(注意这步比较耗时,耐心等待)
sc = scTenifoldKnk(data=df,ko_method="default",ko_genes=["NG-568","NG-1"], qc_kws={"min_lib_size": 10, "min_percent": 0.001},)
result = sc.build()上段代码参数解读:
data:传入转录组数据;
ko_method: 设定敲除基因的方法, 这里不用改,使用“default”默认即可;
ko_genes:指定要虚拟敲除的基因名。这里是列表的形式,所以可以传入多个基因,每个基因用逗号隔开。本例我同时敲除了两个基因:NG-568和NG-1
min_lib_size: 指单个基因在所有细胞或样本中的最小总表达量。如果某个基因的总表达量小于这个阈值,会被过滤掉。这里 10 表示总表达量低于 10 的基因不参与网络构建。
min_percent:指基因在细胞中至少有多少比例是非零表达才保留。例如 0.001 表示至少在 0.1% 的细胞中表达,否则该基因会被过滤掉。这个参数用于去除几乎不表达的基因,避免噪声影响网络。
④ 查看结果
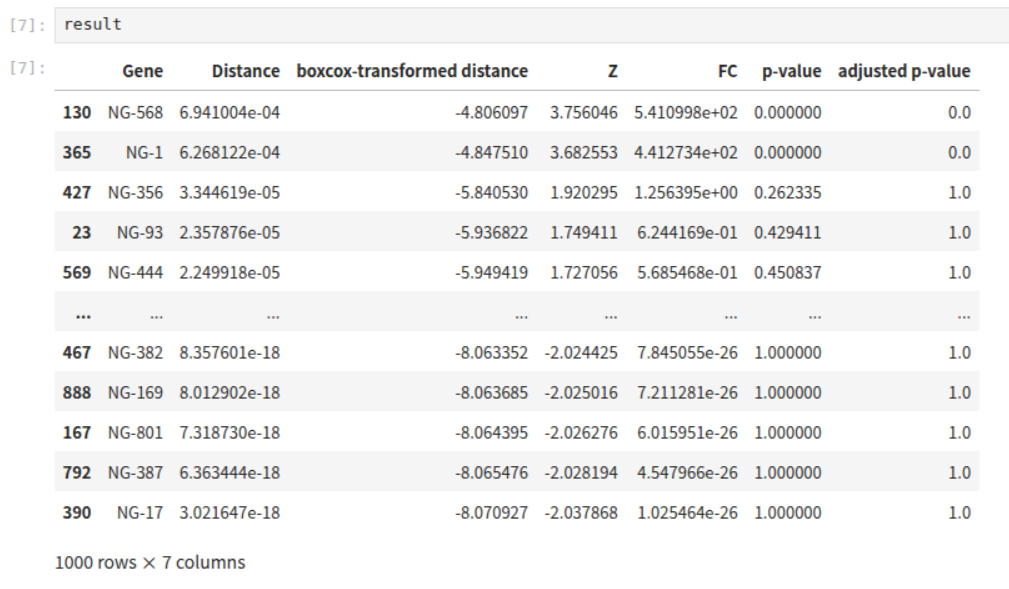
列名含义解释:
Gene:基因名;
Distance:这个基因在虚拟敲除(KO)网络与原始网络中的扰动程度(网络距离)。数值越大,说明该基因受KO影响越大;
boxcox-transformed distance:为了满足统计假设,对原始 Distance 做 Box-Cox 变换后的值,通常用于后续Z统计量计算,使数据更接近正态分布;
Z:标准化后的扰动量:衡量该基因的距离相对于其他基因的离群程度。Z 越大(正或负)表示扰动越明显;
FC:反映虚拟敲除后基因表达或扰动的变化倍数;
p-value: 原始P值;
adjusted p-value: 多重假设校正后的P值。
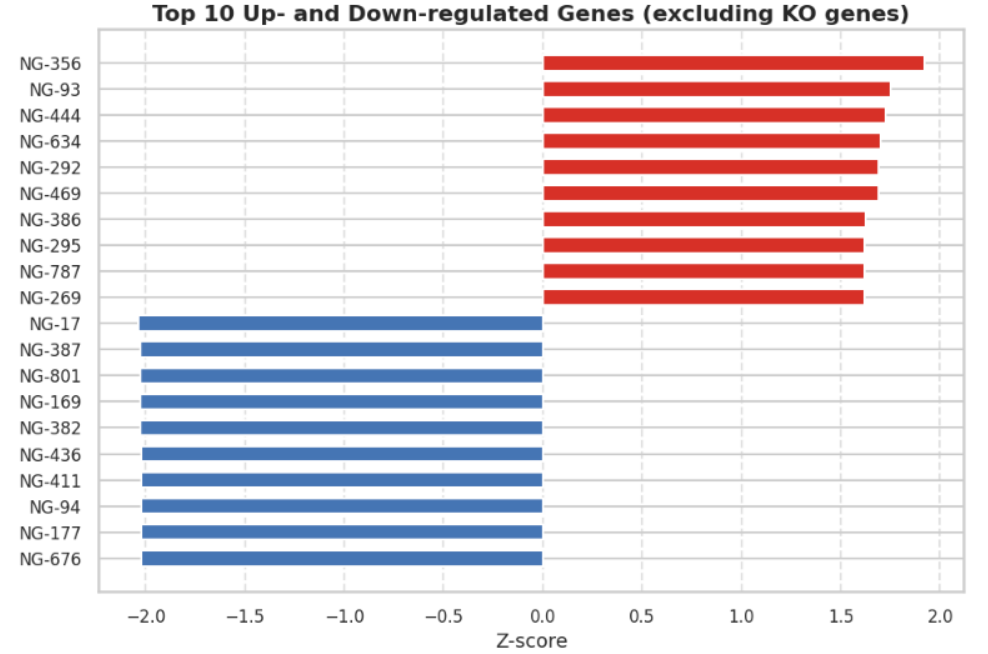
⑤ 绘图(绘制上调及下调幅度排名前10的基因)
import matplotlib.pyplot as plt
import seaborn as sns
import numpy as np
import pandas as pdsns.set(style="whitegrid", context="talk")ko_genes = ["NG-1", "NG-568"] #这里替换成你所敲除的基因名
filtered_result = result[~result["Gene"].isin(ko_genes)]z_pos_top = filtered_result[filtered_result["Z"] > 0].sort_values("Z", ascending=False).head(10)
z_neg_top = filtered_result[filtered_result["Z"] < 0].sort_values("Z").head(10)top_genes = pd.concat([z_pos_top, z_neg_top])
z_values = top_genes["Z"].values
genes = top_genes["Gene"].values
colors = ['#d73027' if z > 0 else '#4575b4' for z in z_values]plt.figure(figsize=(10,7))
plt.barh(genes, z_values, color=colors, height=0.6)
plt.xlabel("Z-score", fontsize=14)
plt.ylabel("")
plt.title("Top 10 Up- and Down-regulated Genes (excluding KO genes)", fontsize=16, fontweight='bold')
plt.gca().invert_yaxis()
plt.xticks(fontsize=12)
plt.yticks(fontsize=12)
plt.grid(axis='x', linestyle='--', alpha=0.6)
plt.tight_layout()
plt.show()