【论文阅读】Masked Conditional Variational Autoencoders for Chromosome Straightening
MC‑VAE 框架由 两步法组成:
Step 1:预处理(Patch Rearrangement)
提取染色体完整中轴线
沿中轴线依次裁取矩形patch
将这些patch按顺序重新排列,使patch中心点在一条垂直直线
Step 2:Masked Conditional Variational Autoencoder (MC‑VAE) 精修
输入:预处理后的初步直染色体(可能还有残留弯曲或长度问题)
条件(Condition):每个patch的曲率信息,作为条件输入
模型结构:
ConvNet CNN → 提取局部纹理
Transformer Encoder → 捕捉全局带型关系
Multiple Conditional Variational Processors → 在曲率条件下生成修正特征
Decoder(带高mask比例 + 高斯噪声掩码)
关键思想:
Mask策略(参考 MAE):训练时高比例mask掉输入的patch → 让模型必须根据剩余的上下文和条件重建缺失部分
高斯噪声mask适配此类显微图的背景特性(背景较大面积空白)
条件VAE学习到 banding pattern ↔ 曲率条件的映射,确保生成的拉直结果结构与细节保真。
创新点总结
预处理:Patch Rearrangement
针对高曲率样本 → 先几何方法消除部分曲率
保证输入到生成网络的是比较“接近直”的版本,减轻生成任务难度
Masked Conditional VAE
将曲率作为条件向VAE传入,指导生成
结合 CNN + Transformer 提取局部与全局特征
高比例mask训练 → 强迫模型学习带型与空间结构的深层表示
高斯噪声mask适配背景,避免模型学到错误特征
Preliminary Processing(预处理) → 先用几何 + patch 重排把低曲率消掉
MC‑VAE(生成式精修) → 在 mask + 条件的 VAE 框架中修正剩余弯曲、补全带型细节、去噪
A. Preliminary Processing 细节
目标:
消除低曲率区域
生成结构规范、patch 已经对齐到直线的初步图像,作为第二步模型的输入
步骤(对照论文 Fig. 2):
1)分割 & 中轴线提取
Otsu 阈值法:在直方图找“全局最小值”作为阈值(直方图两个峰分别是背景像素、染色体像素)。
为了稳定阈值 → 先 3×3 中值滤波平滑直方图
二值化 → 冲掉孔洞(flood fill)确保染色体区域连通
用 Zhang‑Suen 细化算法提取初步中轴线(骨架)
2)中轴线修复
中轴线可能 太短 或 有分支:
如果末端距染色体底部 > 6 像素 → 沿局部梯度方向迭代延伸
否则 → 按比例剪枝(去掉多余的分支)+ 局部梯度延伸
得到完整、单一的中轴线(Fig.2(c))
3)Patch 切分 + 直线重排
把染色体看作沿中轴线排列的矩形 patch 序列
等大小切 patch(如 8×16),patch 中心在中轴线
消除低曲率的方法:
将 patch 按原顺序重排到一条竖直直线上(中心等距分布)
输出一个“初步直”的版本(Fig.2(e))
B. Chromosome Straightening(MC‑VAE 精修)
核心任务:
抹平残留弯曲(高曲率处)
恢复带型细节
去掉噪声
保证结构(长度一致、边缘平滑)
1)Masking + Condition 条件输入
输入准备:
预处理输出的初步直染色体 → 切成 2n 个不重叠 patch(论文默认 H×W=128×32,patch=8×16 → 共32个patch)
按均匀分布随机 mask大比例(实验 70%)patch:
Mask patch 上覆盖高斯噪声(而不是空白)
背景是大量空白,如果直接空mask,模型可能被误导(以为mask区就是背景)→ 高斯噪声避免这个问题
为了保留中轴密度信息:
若 mask patch 跟中轴区域有重叠 → 在 mask 中保留中轴两侧2像素的纹理信息(这也是为了给带型定位提供额外先验)
Masking的设计目的:
去掉大量冗余信息 → 提高推理难度
强化模型重建能力 → 让模型必须学到全局+局部关系
条件 Ic(曲率信息):
用 Sobel 算子计算二值图像的水平梯度(论文用的是只计算水平方向,因为弯曲边缘水平方向变化大)
每个patch计算梯度总量:
大梯度 → 低曲率(基本直的)
小梯度 → 高曲率(弯曲明显)
用阈值 T=18 把每个 patch 分类成:
白(bent)、灰(straight)、黑(blank)
再按原顺序还原到完整“条件图” Ic,与 Im 对应
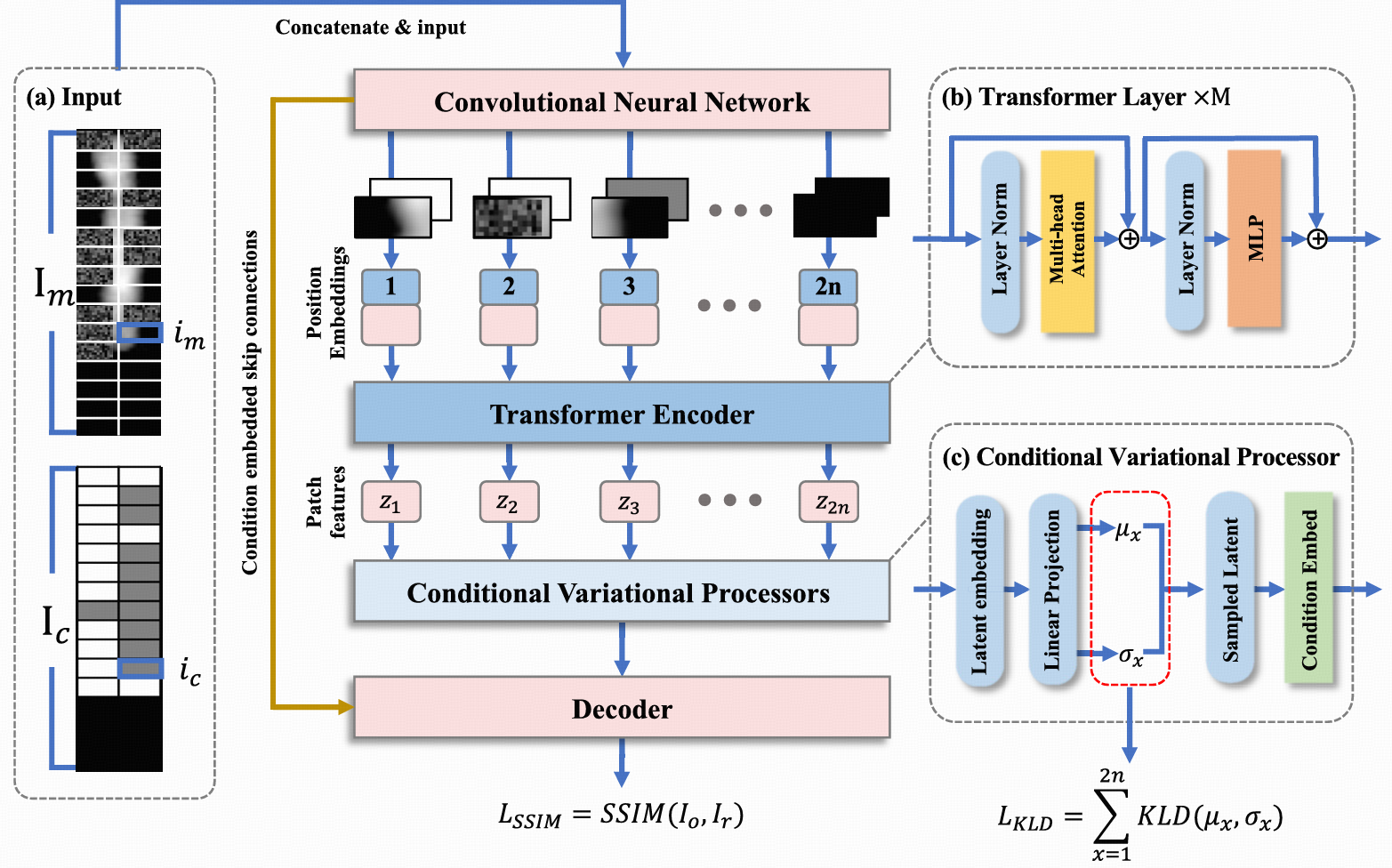
2)MC‑VAE 模型结构(Fig.3)
数据流:
输入:
Masked chromosome Im + 条件 Ic(曲率标签图)CNN 特征提取
三个 ResNet block + MaxPooling(根据输入尺寸调节)
输出为 2n 个 D 维 patch 向量
Transformer Encoder
补充位置编码 Epos
多层 Multi‑Head Self‑Attention + MLP
输出长程依赖编码 Z(每个patch的全局语义)
Conditional Variational Processors (CVP)
每个patch一个CVP模块 → 共享结构,不共享参数
根据输入 patch 表示 zx 估计后验均值 μx 和方差 σx
变分采样得到 latent hx
KLD 损失:KLD=21(μx2+σx2−logσx2−1)让后验分布逼近标准正态 N(0, I),避免 σx→0
输出: 预测 patch 的重建表示 + 对应条件向量
Decoder
三次上采样模块(UpSample + Conv + ReLU)
Skip-connection 把Ic卷积特征跨层送入 → 保证条件信息在不同空间尺度都被利用
输出重建图 Ir
Loss
结构相似度SSIM损失(luminance、contrast、structure 权重均=1)
KLD损失
总损失:Lall=LSSIM+LKLD
推理阶段:
条件图 Ic 中所有patch标灰(表示直的)
弯曲区域mask掉 → 让模型生成对应的直patch替换回去
实现细节
框架:PyTorch + 单 NVIDIA A100
Batch=36, Adam( lr=1e-3, weight_decay=1e-4)
Early stop:10 epochs无提升停止,max 50 epochs
Mask比例:70%,阈值 T=18
训练数据生成:随机弯曲 1-3 control points (factor 1.05-1.35)
Patch大小固定为 8×16
不同数据集会缩放图片比例,让最长染色体适配 128×32
三个公开数据集:
BioImLab(Q-band染色)
694 张 bent + 500 张 straight
Pki(G-band染色)
3554 张 bent + 3000 张 straight
ChromosomeNet(G-band metaphase,独立测试集)
1633 张 bent + 1500 张 straight
数据扩增策略:
从 real straight 染色体,用 ChrSNet [17] 的非刚性变形方法生成 5 张 synthetic bent
注意五折交叉验证时,同一真实图像及其合成版本放在同一fold,防止数据泄漏
同时在 real 和 synthetic 上评估,检验模型泛化
评估指标 (Evaluation Metrics)
论文针对 形态结构 和 带型模式(banding patterns) 两方面设计了一组指标:
形态结构:
长度 L score
与目标直染色体的像素长度接近程度 (公式 15)全局直线度 MA score
基于中轴线的整体斜率与局部斜率差异,越接近 100% 越直 (公式 16, n=6)边缘光滑度 Sobel score
Sobel 水平梯度总和,数值越低表示边缘更平滑 (公式 17)
带型模式:
LPIPS(感知相似度)
衡量生成直染色体与原图带型的一致性 (公式 参考LPIPS[31])DP score(密度曲线差异)
沿中轴线像素灰度值差异平方和,越低越一致 (公式 18)
模型对比 (Model Comparisons)
对比方法:
几何:MA[9],BP[11]
深度学习:ChrSNet[17]
几何+MC-VAE组合
主要结论:
在 BioImLab 和 Pki 的合成 + 真实数据上,MC-VAE+PPA 框架 全指标优于 各方法
L score 提升:相比 MA 方法,合成数据提升 1.385.79%,真实数据提升 3.054.18%
直线度:MA score 在真实数据上能到 97%/90%,全球局直、局部边缘都优(Sobel score 降低 3倍以上)
带型一致性:
LPIPS 高于 ChrSNet(尤其是在真实数据上,平均值更大且方差更小)
DP score 最低(最接近原曲染色体的密度曲线)
单用 MC‑VAE(不带预处理)也能比几何方法好,但低于 PPA + MC‑VAE → 证明预处理的价值
消融实验 (Ablation Studies) — Pki 数据集
基线:PPA 预处理结果
逐步加入组件的性能变化:
PPA + CNN + Transformer + 高斯噪声mask (GNRM) → 结构与带型指标提升,说明混合编码器提特征能力强
PPA + CNN + CVP + GNRM → 比用 Transformer 更能保持结构细节(MA、Sobel 更好)
blank mask(空白mask) → 会混淆模型,带型 LPIPS下降 → 有必要用高斯噪声mask
GNRM vs GNLRM(只mask 左右半行) → 全patch高斯噪声mask 效果更好(mask比例可达70%,去除更多冗余信息)
结合不同几何输入(MA/BP/PPA+MC-VAE) → PPA+MC-VAE最好
最终最佳配置:PPA + CNN + Transformer + CVP + GNRM
参数影响 (Effect of Parameters)
预处理修剪比例 (Prune ratio)
变化影响不大,0.1 最优
Mask比例
真实+合成数据,70% mask比例最优
避免信息冗余
与 MAE[19] 一致,高mask让模型学到更多判别特征
超过 70% → 信息不足,性能下降
Patch大小
8×16 最优
太大:特征维度过高,训练难度↑,性能下降
太小:信息不足,捕捉不到长程依赖
潜在应用 (Potential Application)
直化后提升下游分类性能
三个分类模型:VGG-16、ResNet-18、AlexNet
测试集:BioImLab + Pki
结论:
PPA+MC-VAE 直化 → 分类准确率、精确率、召回率和F1均提升
BP、ChrSNet 对分类提升不大,甚至接近原始数据
MA 方法性能略升(+12%),但低于我们方法(+34%)
MC‑VAE可配合 BP/MA 使用,也能带来增益
意义:
框架可与任意karyotyping系统配合,增强分类和异常检测
7️⃣ 最重要的 Takeaways
PPA 解决低曲率 + patch对齐 → 为生成网络减轻负担
MC‑VAE 高斯mask 强制模型学全局+局部带型关系
高mask(70%) + 合适的 patch size(8×16)是性能最佳点
PPA+MC‑VAE 在结构保持和带型一致性上双赢,还显著提升了分类性能,这在临床有直接价值
