蛋白质残基 - 残基距离计算:单蛋白工具与批量处理方案
在蛋白质结构分析、功能预测及疾病机制研究中,残基 - 残基距离是核心分析指标之一。本文整理了常用的单蛋白残基距离计算工具,并提供批量处理方案,帮助科研人员提升分析效率。
一、为何需计算蛋白质残基 - 残基距离?
蛋白质的稳定结构与功能依赖于氨基酸残基间的相互作用(氢键、疏水作用、盐桥等),而残基间的空间距离直接决定了这些相互作用的强度与类型,其计算价值主要体现在:
- 验证结构稳定性:通过距离判断氢键(0.25-0.35nm 范围)、疏水堆积等作用是否合理,辅助识别 PDB 文件建模偏差或构象不稳定区域;
- 定位功能位点:活性中心关键残基(如丝氨酸蛋白酶催化三联体)需通过特定距离排布实现功能,距离分析可辅助解析催化机制;
- 支撑结构预测与模拟:同源建模、从头预测中,残基距离约束能提升结构精度;分子动力学模拟中,追踪距离变化可分析构象动态;
- 疾病与突变研究:基因突变导致的残基距离异常可能引发蛋白功能紊乱,如镰刀型贫血症与血红蛋白 β 链残基突变后的疏水作用异常相关。
二、单蛋白质残基距离计算工具推荐(网页端)
若仅需分析单个蛋白质,以下网页工具可满足基础需求,无需本地部署,操作便捷:
| 工具名称 | 输入方式 | 核心输出 | 特色优势 | 访问地址 |
| PDBePISA | PDB ID / 上传结构文件 | 残基距离列表 + 相互作用界面 | 自动识别蛋白界面残基 | https://www.ebi.ac.uk/pdbe/pisa/ |
| ProteinContactAtlas | PDB ID | 8Å 接触图 + 可视化结果 | 支持按残基类型筛选结果 | https://www.mrc-lmb.cam.ac.uk/rajini/proteincontactatlas/ |
| COCOMAPS | 上传 PDB 文件 | 交互式残基距离热图 | 适配多链复合物结构分析 | https://www.molnac.unisa.it/BioTools/cocomaps/ |
| CAPRI Contact | PDB ID | 5/8/10Å 距离阈值列表 | 结合对接打分,辅助结合预测 | CAPRI HOME |
| RINalyzer | PDB ID / 本地文件 | 残基作用网络图 + 距离数据 | Cytoscape 插件,支持可视化分析 | https://www.rinalyzer.de/ |
| distance-matrix.ru | PDB ID | 整链 Cα 原子距离矩阵(可下载) | 支持自定义残基范围 | http://distance-matrix.ru/ |
三、批量计算痛点与解决方案
当需处理大量 PDB 结构(如几十甚至上百个 ID)时,逐个通过网页工具计算会消耗大量时间。针对这一痛点,可采用药理实验屋开发的批量下载工具,操作流程如下:
1. 前期准备
- 新建文本文件,按 “每行 1 个 PDB ID” 的格式整理需分析的 ID(示例:1a4g、2v3p、3h7k 等);
- 确保本地环境已安装 Python 基础依赖(无需复杂配置,基础环境即可)。
2. 工具使用步骤
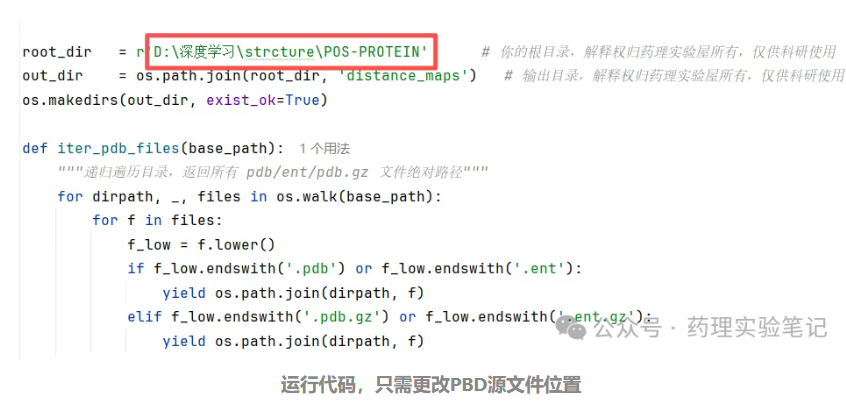
- 获取批量处理代码(见文末说明);
- 打开代码文件,修改 “PDB 源文件路径” 参数,指向第一步准备的文本文件;
- 运行代码,工具将自动读取 PDB ID 列表,批量请求计算并下载结果文件。
3. 工具优势
- 无需手动重复操作,大幅减少机械性工作时间;
- 结果文件按 PDB ID 分类存储,便于后续整理分析;
- 支持自定义残基范围,适配不同分析需求。