生信分析自学攻略 | R语言数据筛选和修改
在《生信小白自学攻略》系列的前几篇文章中,我们已经了解了 R 和 RStudio 的安装、RStudio 的深度探索,以及 R 语言的基本数据类型和数据结构。现在,是时候深入探讨如何运用 R 语言对数据进行精细化处理了。本篇推文将详细介绍如何在 R 中对数据进行排序、筛选、替换以及调用特定行和列等核心操作。
准备工作:导入你的数据
在进行任何数据处理之前,我们首先需要将文件中的数据导入到 R 中。常用的数据格式包括 .csv(逗号分隔值)、 .txt(制表符分隔值)、.xls 和 .xlsx 等格式。R语言有内置函数读取文件,也有扩展包函数读取相应文件。在这里,我只介绍常用的,掌握这些函数就可以熟练读取不同文件了。
read.table(): 这是一个通用的文本文件读取函数,你可以通过 sep 参数指定分隔符(如空格、制表符或逗号)。默认情况下,它能识别空格或制表符作为分隔符。
read_excel(): 读取 Excel 文件,可以指定工作表 (sheet)。
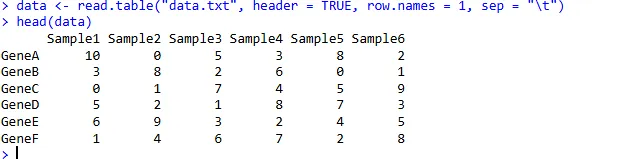
假设你有一个名为 data.txt 的基因表达数据文件,它包含了基因ID、样本信息和表达量等,我们可以这样导入:
# 制表符分隔的TXT文件
data <- read.table("data.txt", header = TRUE, row.names = 1, sep = "\t")# 如果是逗号分隔的csv文件
# data <- read.table("data.csv", header = TRUE, row.names = 1, sep = ",")# 查看数据的前几行,确保导入正确
head(data)
导入数据后,你可以使用 dim() 查看数据的维度(行数和列数),str() 查看数据结构,以及 summary() 获取数据的统计摘要。
R 语言数据处理的核心利器:dplyr 包
在 R 语言中进行数据处理,dplyr 包是不可或缺的工具。它是 tidyverse 系列包中的一员,提供了一套简洁、一致的函数,让数据操作变得直观高效。dplyr 的核心理念是使用一系列“动词”来描述数据操作,如 filter()(筛选)、select()(选择)、arrange()(排序)、mutate()(新增/修改列)和 summarise()(汇总)。
如果尚未安装 dplyr 包,请先安装 tidyverse(dplyr 是其一部分):
install.packages("tidyverse")
library(dplyr)
数据筛选 (filter())
数据筛选是根据特定条件选择数据子集的操作。dplyr 中的 filter() 函数可以帮助我们轻松实现这一点。
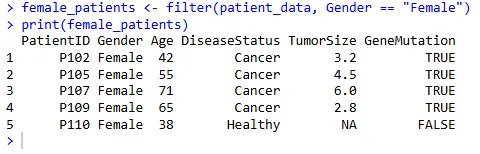
单条件筛选
选择所有性别为“Female”的患者:
female_patients <- filter(patient_data, Gender == "Female")
print(female_patients)
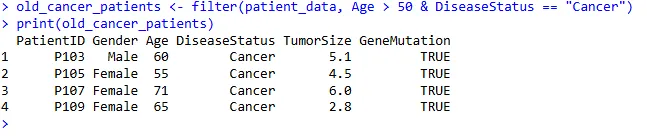
多条件筛选
选择所有年龄大于 50 岁且患有癌症的患者:
old_cancer_patients <- filter(patient_data, Age > 50 & DiseaseStatus == "Cancer")
print(old_cancer_patients)
注意:
==表示“等于”!=表示“不等于”>或>=表示“大于”或“大于等于”<或<=表示“小于”或“小于等于”&表示“和”(所有条件都满足)|表示“或”(满足任一条件)%in%表示“包含于”(例如:DiseaseStatus %in%c("Cancer", "Healthy"))is.na()用于筛选缺失值!is.na()用于筛选非缺失值
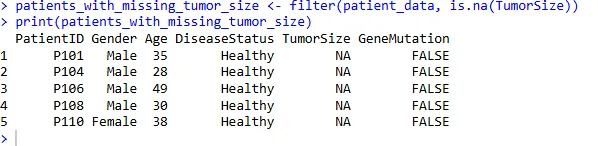
筛选缺失值
筛选 TumorSize 列中存在缺失值的行:
patients_with_missing_tumor_size <- filter(patient_data, is.na(TumorSize))
print(patients_with_missing_tumor_size)
数据排序 (arrange())
数据排序是根据一个或多个列的值,对数据框中的行进行升序或降序排列。dplyr 中的 arrange() 函数可以完成这项任务。
单列升序排序
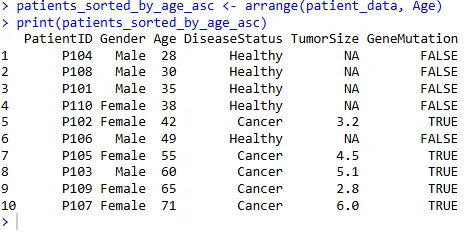
根据患者年龄 Age 进行升序排列:
patients_sorted_by_age_asc <- arrange(patient_data, Age)
print(patients_sorted_by_age_asc)
单列降序排序
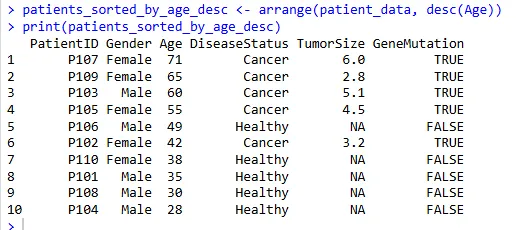
根据患者年龄 Age 进行降序排列,需要使用 desc() 函数:
patients_sorted_by_age_desc <- arrange(patient_data, desc(Age))
print(patients_sorted_by_age_desc)
多列排序
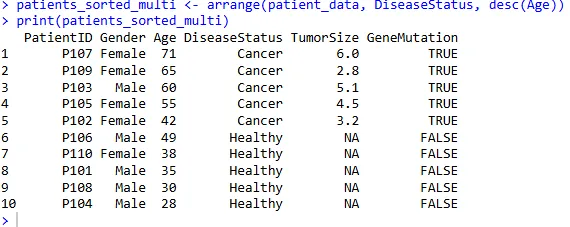
首先按疾病状态 DiseaseStatus 升序,然后按年龄 Age 降序排列:
patients_sorted_multi <- arrange(patient_data, DiseaseStatus, desc(Age))
print(patients_sorted_multi)
列的选取和操作 (select(), mutate(), rename())
对数据框的列进行操作是数据处理的另一个重要方面,包括选取、新增、修改和重命名列。
选取特定列 (select())
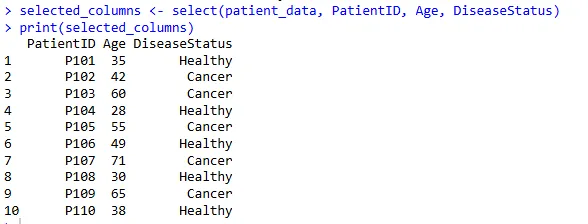
只保留 PatientID、Age 和 DiseaseStatus 三列:
selected_columns <- select(patient_data, PatientID, Age, DiseaseStatus)
print(selected_columns)
除了直接指定列名,你也可以使用一些辅助函数:
startswith("prefix"):选择以特定前缀开头的列ends_with("suffix"):选择以特定后缀结尾的列contains("string"):选择包含特定字符串的列matches("regex"):选择匹配正则表达式的列everything():选择所有列c()或::选取连续的列
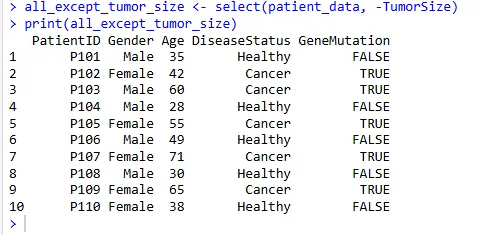
例如,选择除了 TumorSize 之外的所有列:
all_except_tumor_size <- select(patient_data, -TumorSize)
print(all_except_tumor_size)
新增或修改列 (mutate())
mutate() 函数用于创建新列,或修改现有列。
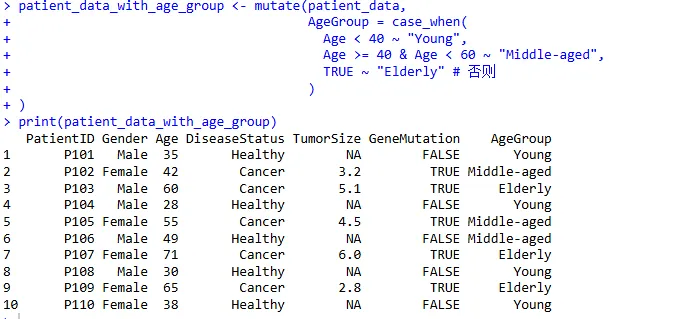
1、新增列:计算年龄组
根据年龄创建一个新的 AgeGroup 列:
patient_data_with_age_group <- mutate(patient_data,AgeGroup = case_when(Age < 40 ~ "Young",Age >= 40 & Age < 60 ~ "Middle-aged",TRUE ~ "Elderly" # 否则)
)
print(patient_data_with_age_group)
case_when() 是一个非常有用的函数,用于实现多条件判断赋值。
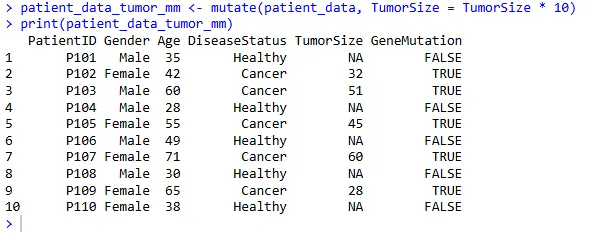
2、修改现有列:肿瘤大小单位转换
假设 TumorSize 是厘米,我们想将其转换为毫米并覆盖原列:
patient_data_tumor_mm <- mutate(patient_data, TumorSize = TumorSize * 10)
print(patient_data_tumor_mm)
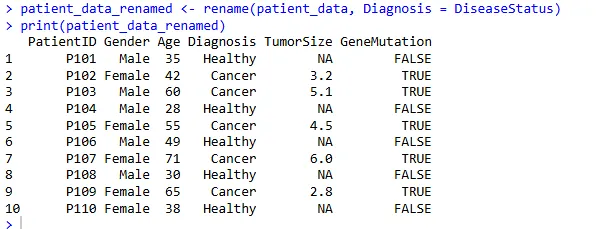
3、重命名列 (rename())
将 DiseaseStatus 列重命名为 Diagnosis:
patient_data_renamed <- rename(patient_data, Diagnosis = DiseaseStatus)
print(patient_data_renamed)
语法是 新列名 =旧列名。
数据的替换和清理 (replace_na(), 基础R替换)
在真实数据中,缺失值(NA)非常常见,对其进行合理处理至关重要。此外,有时我们也需要替换特定值。
缺失值替换 (replace_na())
tidyr 包(也是 tidyverse 的一部分)中的 replace_na() 函数可以方便地替换缺失值。
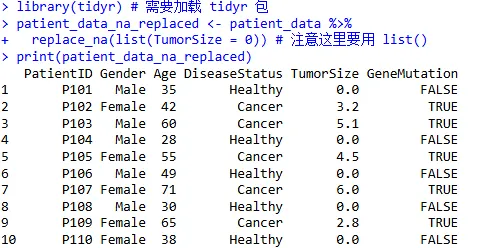
将 TumorSize 列中的缺失值(NA)替换为 0:
library(tidyr) # 需要加载 tidyr 包patient_data_na_replaced <- patient_data %>%replace_na(list(TumorSize = 0)) # 注意这里要用 list()
print(patient_data_na_replaced)
在这里,我们引入了 %>% 管道符,它来自 magrittr 包(tidyverse 自动加载)。管道符可以将前一个函数的输出作为后一个函数的第一个参数,大大提高了代码的可读性,让数据处理流程更加流畅。上面的代码可以理解为“将 patient_data 传递给 replace_na() 函数”。
替换特定值
有时我们需要根据条件替换某个列中的值。例如,将 DiseaseStatus 列中的“Healthy”替换为“Control”。
# 使用 ifelse() 函数
patient_data_status_replaced <- patient_data
patient_data_status_replaced$DiseaseStatus <- ifelse(patient_data_status_replaced$DiseaseStatus == "Healthy","Control",patient_data_status_replaced$DiseaseStatus
)
print(patient_data_status_replaced)
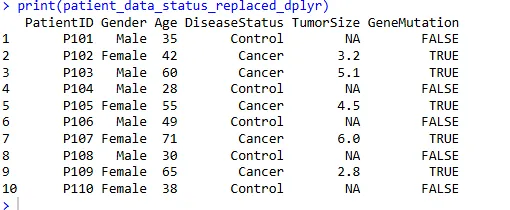
或者使用 dplyr 的 mutate() 和 case_when() 组合,它通常更灵活:
patient_data_status_replaced_dplyr <- patient_data %>%mutate(DiseaseStatus = case_when(DiseaseStatus == "Healthy" ~ "Control",TRUE ~ DiseaseStatus # 否则保持原样))
print(patient_data_status_replaced_dplyr)
小结
本篇教程详细介绍了 R 语言中数据导入以及对数据进行筛选、替换和调用特定行和列等核心操作。我们重点讲解了 dplyr 包的强大功能,并通过实际示例演示了 filter()、arrange()、select()、mutate() 和 rename() 等函数的用法。
本系列有关R和Rstudio的文章已经更新了四篇,大家可以尝试处理一下手头的数据,R语言的快速掌握离不开动手实践!下一篇我们将学习R语言函数与参数的介绍。
