K-Means 聚类
一·介绍
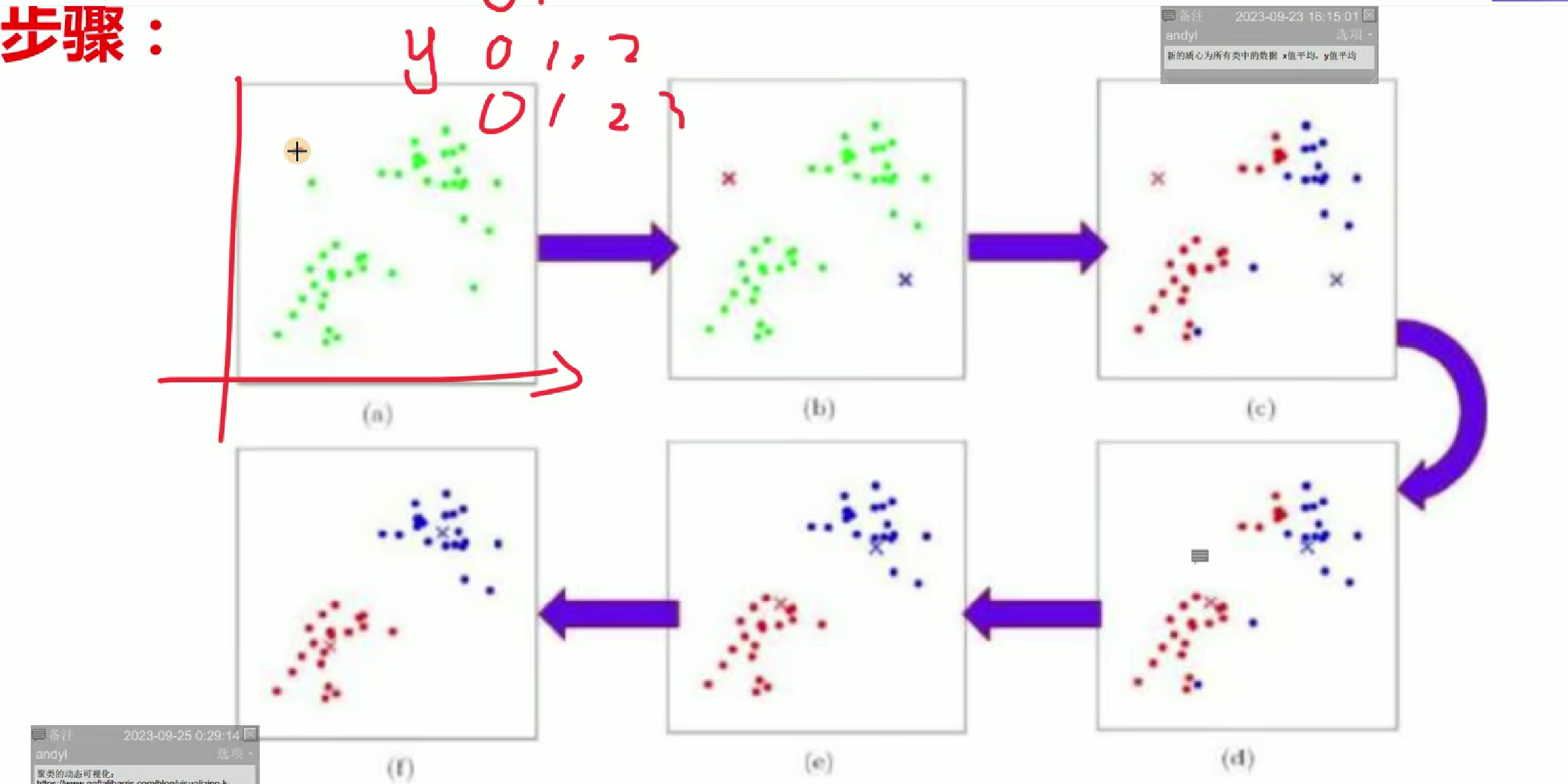
K-mean聚类是一种无监督学习算法,核心是将数据分为K个簇。通过随机选K个初始质心,计算样本与质心距离并归类,再更新各簇质心,重复迭代至质心稳定。它简单高效,适用于大规模数据,但需预先确定K值,结果易受初始质心影响,常用于客户分群、图像分割等场景。
图中选两个质心点,起始点
聚类效果评价方式
轮廓系数:
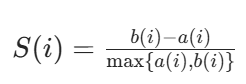
a (i):对于第 i 个元素 xi,计算 xi 与其同一个簇内所有其他元素距离的平均值,表示了簇内的凝聚程度。
b (i):选取 xi 外的一个簇,计算 xi 与该簇内所有点距离的平均距离,遍历其他所有簇,取所有平均值中最小的一个,表示簇间的分离度。
计算所有 x 的轮廓系数,求出平均值即为当前聚类的整体轮廓系数。
我们复习一下
分类的评估方法有:
分类的评估方法有:
分类问题主要用于预测离散的类别标签,常用评估方法围绕类别预测的准确性、完整性等展开,具体包括:
- 准确率(Accuracy):正确预测的样本数占总样本数的比例,公式为:
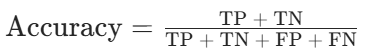
- (注:TP 为真正例,TN 为真负例,FP 为假正例,FN 为假负例)
- 精确率(Precision):预测为正例的样本中实际为正例的比例,反映预测结果的 “精度”,公式为:
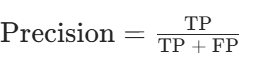
召回率(Recall):实际为正例的样本中被正确预测为正例的比例,反映对正例的 “覆盖能力”,公式为:
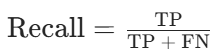
F1 分数(F1-Score):精确率和召回率的调和平均数,用于平衡两者的矛盾,公式为:
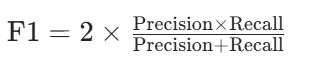
- 混淆矩阵(Confusion Matrix):以矩阵形式展示各类别预测结果与实际结果的对应关系,直观呈现错分情况(如二分类中的 TP、TN、FP、FN)。
- ROC 曲线与 AUC:
- ROC 曲线以假正例率(FPR)为横轴、真正例率(TPR)为纵轴,反映不同阈值下模型的分类性能。
- AUC(Area Under ROC Curve)为 ROC 曲线下的面积,取值范围 [0,1],值越大说明模型区分正负例的能力越强。
- 对数损失(Log Loss):衡量预测概率与实际标签的差距,适用于输出概率的分类模型,公式为:
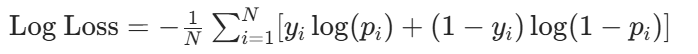
- (注:\(y_i\)为实际标签,\(p_i\)为预测为正例的概率)
回归的评估方法有:
回归问题用于预测连续数值,评估方法主要衡量预测值与实际值的误差大小,具体包括:
- 均方误差(MSE,Mean Squared Error):预测值与实际值差值的平方的平均值,对异常值敏感,公式为:
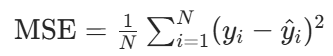
- (注:\(y_i\)为实际值,\(\hat{y}_i\)为预测值,N为样本数)
- 均方根误差(RMSE,Root Mean Squared Error):MSE 的平方根,与目标变量同量级,更易解释,公式为:
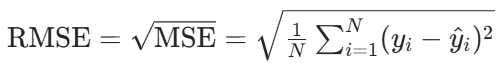
平均绝对误差(MAE,Mean Absolute Error):预测值与实际值差值的绝对值的平均值,对异常值不敏感,公式为:
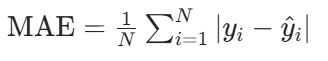
决定系数(\(R^2\),R-Squared):衡量模型对数据的解释程度,取值范围\((-\infty, 1]\),值越接近 1 说明模型拟合越好,公式为:
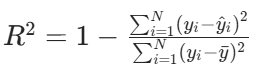
- (注:\(\bar{y}\)为实际值的平均值)
- 平均绝对百分比误差(MAPE,Mean Absolute Percentage Error):以相对误差衡量预测精度,适用于需要百分比解释的场景,公式为:
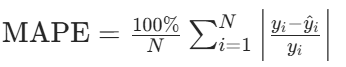
- 注:当\(y_i=0\)时不适用)
- 残差分析:通过观察残差(实际值 - 预测值)的分布、是否随机等,判断模型是否存在偏差或未捕捉的模式(如残差呈非线性分布可能说明模型假设不合理)。
不记得看我之前写的文章有讲

二·参数
class sklearn.cluster.KMeans(n_clusters=8, init='k-means++', n_init=10, max_iter=300, tol=0.0001, precompute_distances='auto', verbose=0, random_state=None, copy_x=True, n_jobs=None, algorithm='auto')[source]【参数:】
n_clusters: 类中心的个数,就是要聚成几类。【默认是8个】
init: 参初始化的方法,默认为'k-means++'
(1)'k-means++': 用一种特殊的方法选定初始质心从而能加速迭代过程的收敛.
(2) 'random': 随机从训练数据中选取初始质心。
(3) 如果传递的是一个 ndarray,则应该形如 (n_clusters, n_features) 并给出初始质心。
n_init: 整形,缺省值=10
用不同的质心初始化值运行算法的次数,最终解是在 inertia 意义下选出的最优结果。
max_iter:
执行一次 k-means 算法所进行的最大迭代数。
Tol: 与 inertia 结合来确定收敛条件。precompute_distances: 三个可选值,'auto',True 或者 False。
预计算距离,计算速度更快但占用更多内存。
(1) 'auto': 如果 样本数乘以聚类数大于 12million 的话则不预计算距离。
(2)True: 总是预先计算距离。
(3)False: 永远不预先计算距离。
verbose: 整形,默认值=0
random_state:随机状态
copy_x: 布尔型,默认值=True
当我们 precomputing distances 时,将数据中心化会得到更准确的结果。如果把此参数值设为 True,则原始数据不会被改变。如果是 False,则会直接在原始数据 上做修改并在函数返回值时将其还原。但是在计算过程中由于有对数据均值的加减运算,所以数据返回后,原始数据和计算前可能会有细小差别。
algorithm:'auto','full' or 'elkan'.默认为'auto'
full:采用经典的 EM 算法
elkan:通过使用三角不等式从而更有效,但不支持稀疏数据
auto:数据稀疏选择 full 模式,数据稠密选择 elkan 模式重点说明(加粗关键部分)
在 K-Means 聚类中,n_clusters(决定聚成几类,是聚类核心目标的直接体现 )、init(初始质心选择,影响算法收敛效率与最终结果,k-means++ 是常用且更优的初始化方式 )、max_iter(单次算法迭代上限,关系到算法是否能充分收敛 )、algorithm(算法实现模式,影响计算效率,elkan 在非稀疏数据下更高效 ) 是尤为关键的参数,直接左右聚类的效果、效率与稳定性 。其中 n_clusters 是 K-Means 聚类任务定义的基础,明确要聚成的类别数;init 里的 k-means++ 初始化对算法成功与否影响重大,合理初始化能大幅减少迭代次数、提升结果质量 ,它们是理解和用好 K-Means 算法的核心要点 。
三·代码
import pandas as pd
from sklearn.cluster import KMeans
from sklearn import metrics
import matplotlib.pyplot as plt# 读取文件
beer = pd.read_table("data.txt", sep=' ', encoding='utf8', engine='python')
# 传入变量(列名)
X = beer[["calories", "sodium", "alcohol", "cost"]]# 根据分成不同的簇,自动计算轮廓系数得分
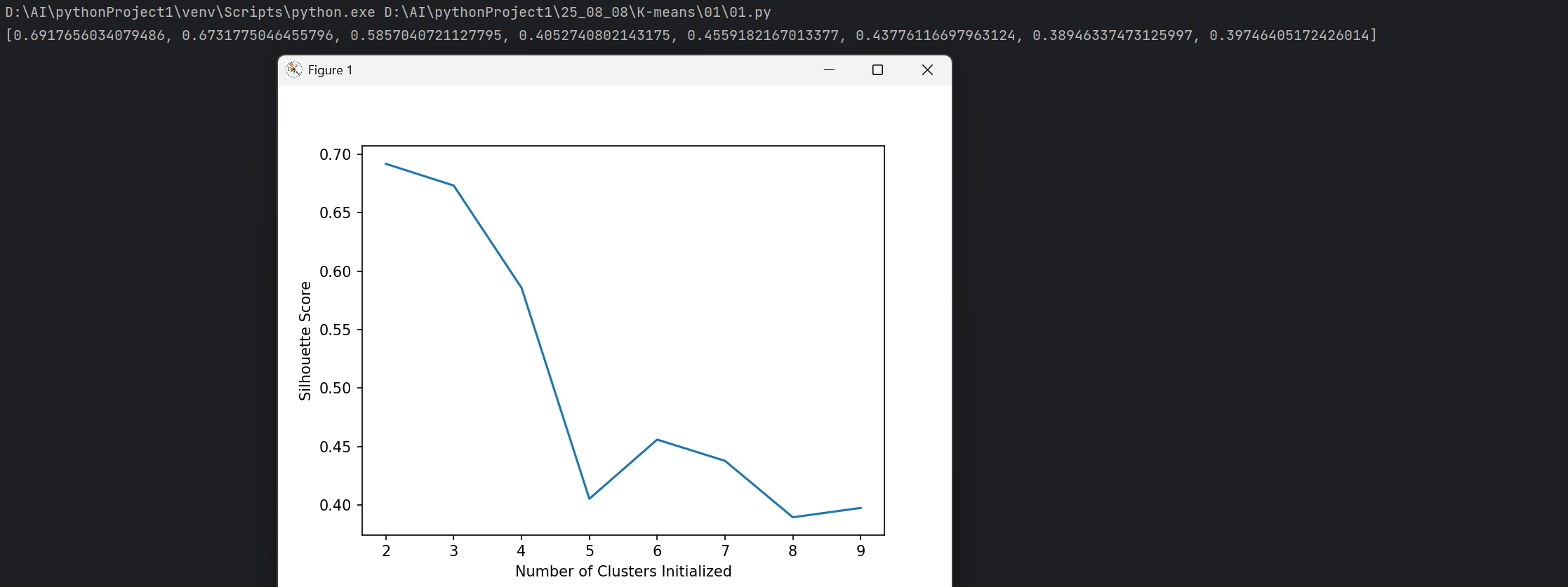
scores = []
for k in range(2, 10): # 寻找合适的K值labels = KMeans(n_clusters=k).fit(X).labels_ # 从左到右依次进行计算score = metrics.silhouette_score(X, labels) # 轮廓系数scores.append(score)print(scores)# 绘制得分结果
plt.plot(list(range(2, 10)), scores)
plt.xlabel("Number of Clusters Initialized")
plt.ylabel("Silhouette Score")
plt.show()# 聚类
km = KMeans(n_clusters=2).fit(X) # K值为2【分为2类】
beer['cluster'] = km.labels_# 对聚类结果进行评分
"""
采用轮廓系数评分
X:数据集 scaled_cluster: 聚类结果
score: 非标准化聚类结果的轮廓系数
"""
score = metrics.silhouette_score(X, beer.cluster)
print(score)https://www.naftaliharris.com/blog/visualizing-kmeans-clustering/
讲代码前先说一下这个网站,先打开
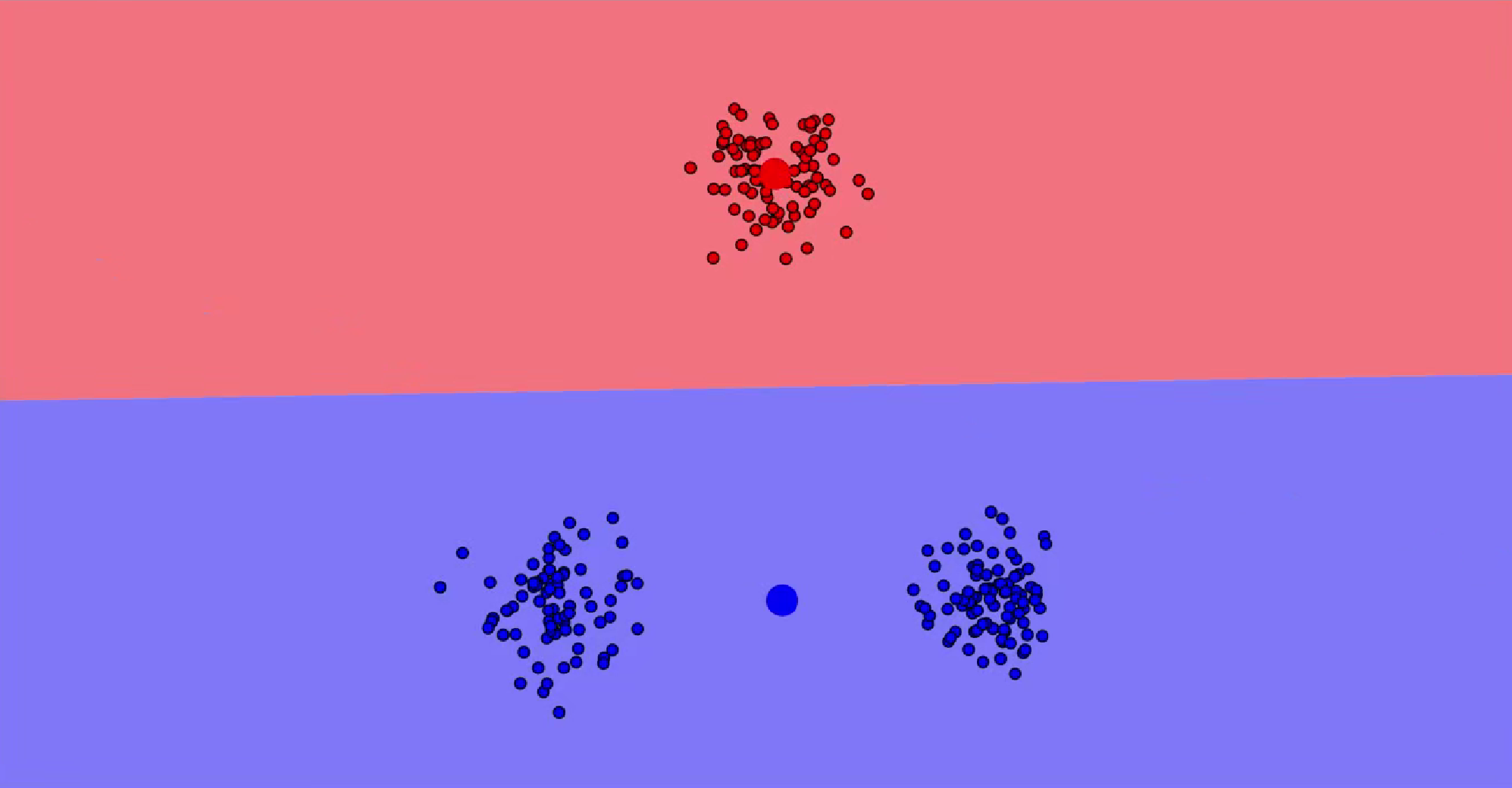
简单说明就是初始起点是会对分布有效果的
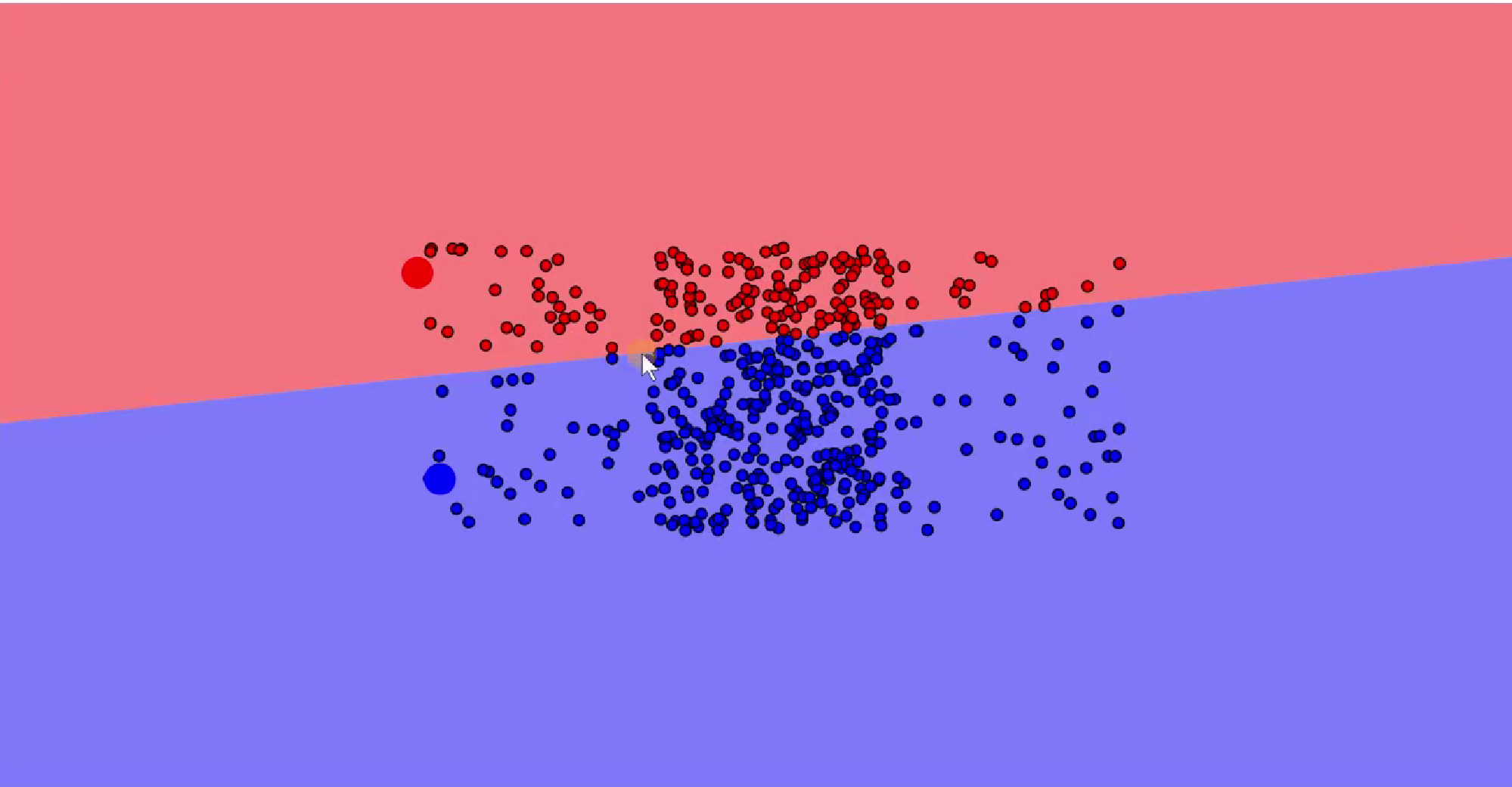
可以看到区别设置起始点不同聚类的结果也不同
现在讲代码
import pandas as pd
from sklearn.cluster import KMeans
from sklearn import metrics
import matplotlib.pyplot as plt# 读取文件
beer = pd.read_table("data.txt", sep=' ', encoding='utf8', engine='python')
# 传入变量(列名)
X = beer[["calories", "sodium", "alcohol", "cost"]]# 根据分成不同的簇,自动计算轮廓系数得分
scores = []
for k in range(2, 10): # 寻找合适的K值labels = KMeans(n_clusters=k).fit(X).labels_ # 从左到右依次进行计算score = metrics.silhouette_score(X, labels) # 轮廓系数scores.append(score)print(scores)# 绘制得分结果
plt.plot(list(range(2, 10)), scores)
plt.xlabel("Number of Clusters Initialized")
plt.ylabel("Silhouette Score")
plt.show()# 聚类
km = KMeans(n_clusters=2).fit(X) # K值为2【分为2类】
beer['cluster'] = km.labels_# 对聚类结果进行评分
"""
采用轮廓系数评分
X:数据集 scaled_cluster: 聚类结果
score: 非标准化聚类结果的轮廓系数
"""
score = metrics.silhouette_score(X, beer.cluster)
print(score)一·导入库
import pandas as pd
from sklearn.cluster import KMeans
from sklearn import metrics
import matplotlib.pyplot as plt二· 读取文件和传入变量(列名)
# 读取文件
beer = pd.read_table("data.txt", sep=' ', encoding='utf8', engine='python')
# 传入变量(列名)
X = beer[["calories", "sodium", "alcohol", "cost"]]
三·根据分成不同的簇,自动计算轮廓系数得分
scores = []
for k in range(2, 10): # 寻找合适的K值labels = KMeans(n_clusters=k).fit(X).labels_ # 从左到右依次进行计算score = metrics.silhouette_score(X, labels) # 轮廓系数scores.append(score)
整个代码最关键的就是
labels = KMeans(n_clusters=k).fit(X).labels_ # 从左到右依次进行计算score = metrics.silhouette_score(X, labels) # 轮廓系数km用法
# 1. 初始化 KMeans 模型,设置聚类数量为 k
km = KMeans(n_clusters=k)
# 2. 使用数据 x 训练模型,让模型学习数据特征来进行聚类
km.fit(x)
# 3. 获取聚类后每个样本的标签,即每个样本被划分到哪个簇(聚类结果标识)
km.labels_ 四·绘制+聚类
# 绘制得分结果
plt.plot(list(range(2, 10)), scores)
plt.xlabel("Number of Clusters Initialized")
plt.ylabel("Silhouette Score")
plt.show()# 聚类
km = KMeans(n_clusters=2).fit(X) # K值为2【分为2类】
beer['cluster'] = km.labels_五·评分
score = metrics.silhouette_score(X, beer.cluster)
print(score)最终呈现