基因编辑预测工具:inDelphi与Pythia

CRISPR基因编辑技术的快速发展,让准确预测编辑结果变得对科研人员来说至关重要。今天我们要介绍刚刚发表的两个重要的基因编辑预测工具:inDelphi和Pythia,它们为研究人员提供了强大的计算方法来预测和优化CRISPR/Cas9系统的编辑结果。
inDelphi
https://indelphi.giffordlab.mit.edu/about

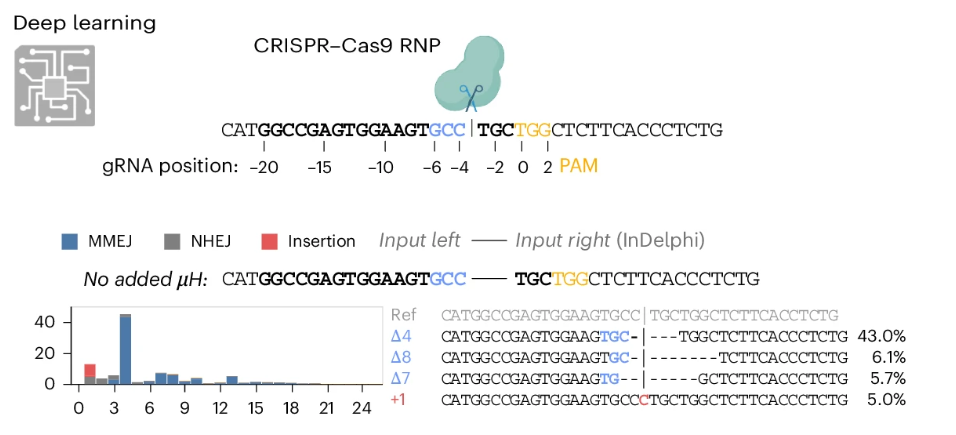
inDelphi是一个专门用于预测DNA修复结果的计算工具。它由MIT Gifford实验室开发。这个工具的核心功能是预测由CRISPR/SpCas9诱导的DNA双链断裂后的非同源末端连接修复结果。inDelphi能够预测1-bp插入和1-60bp缺失基因型的相对频率,这些结果占到了观察到的编辑结果的80-95%,特别适用于预测靶向基因编码区域的gRNA的移码频率。
这个工具的科学基础相当扎实。inDelphi基于大量的实验数据训练,包括在mESC、U2OS、HCT116、K562和HEK293细胞类型中生成的数据。模型主要关注三种主要的修复类型:1-bp插入、微同源性缺失和非微同源性缺失。其中1-bp插入会复制-4位置的核苷酸,其频率与细胞类型、DNA基序和gRNA方向相关;微同源性缺失通过微同源性介导的末端连接机制产生;而非微同源性缺失则通过Lig4介导的末端连接产生。
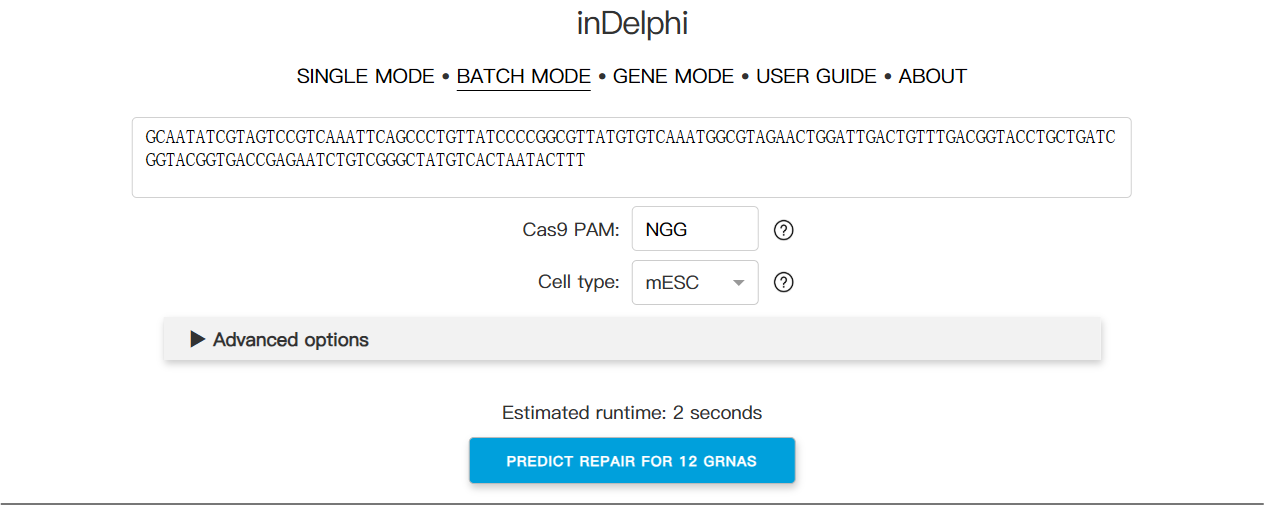
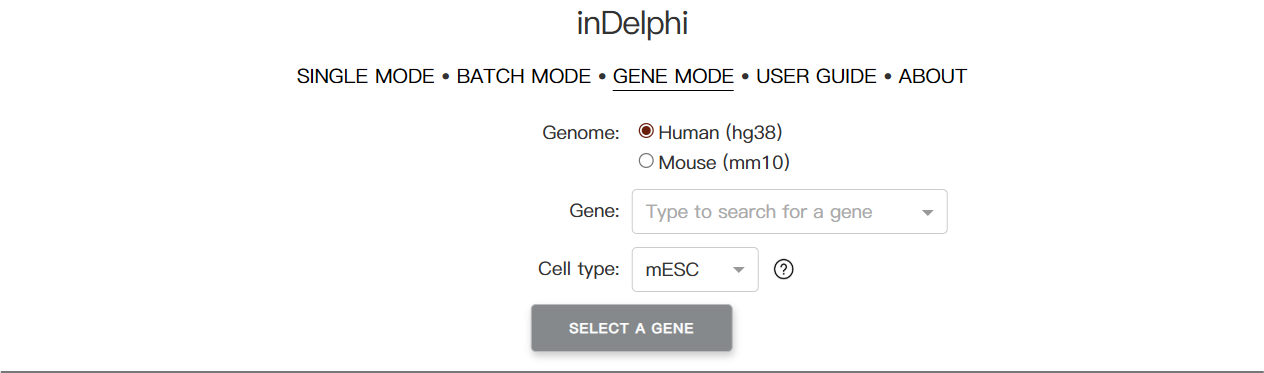
inDelphi在线交互式实现支持三种主要工作模式。单模式让用户输入序列上下文和切割位置,获得插入缺失频率预测和汇总统计;批处理模式可以输入序列上下文和PAM,获得序列上下文中与PAM兼容的所有gRNA的预测汇总统计;基因模式则允许用户输入hg38或mm10编码基因,获得靶向该编码基因的所有SpCas9 gRNA的预测汇总统计。不过需要注意的是,inDelphi主要针对SpCas9进行训练,在哺乳动物细胞类型中表现最佳,但不适用于细菌、植物和非哺乳动物真核生物。
Pythia
https://pythia.pythonanywhere.com/
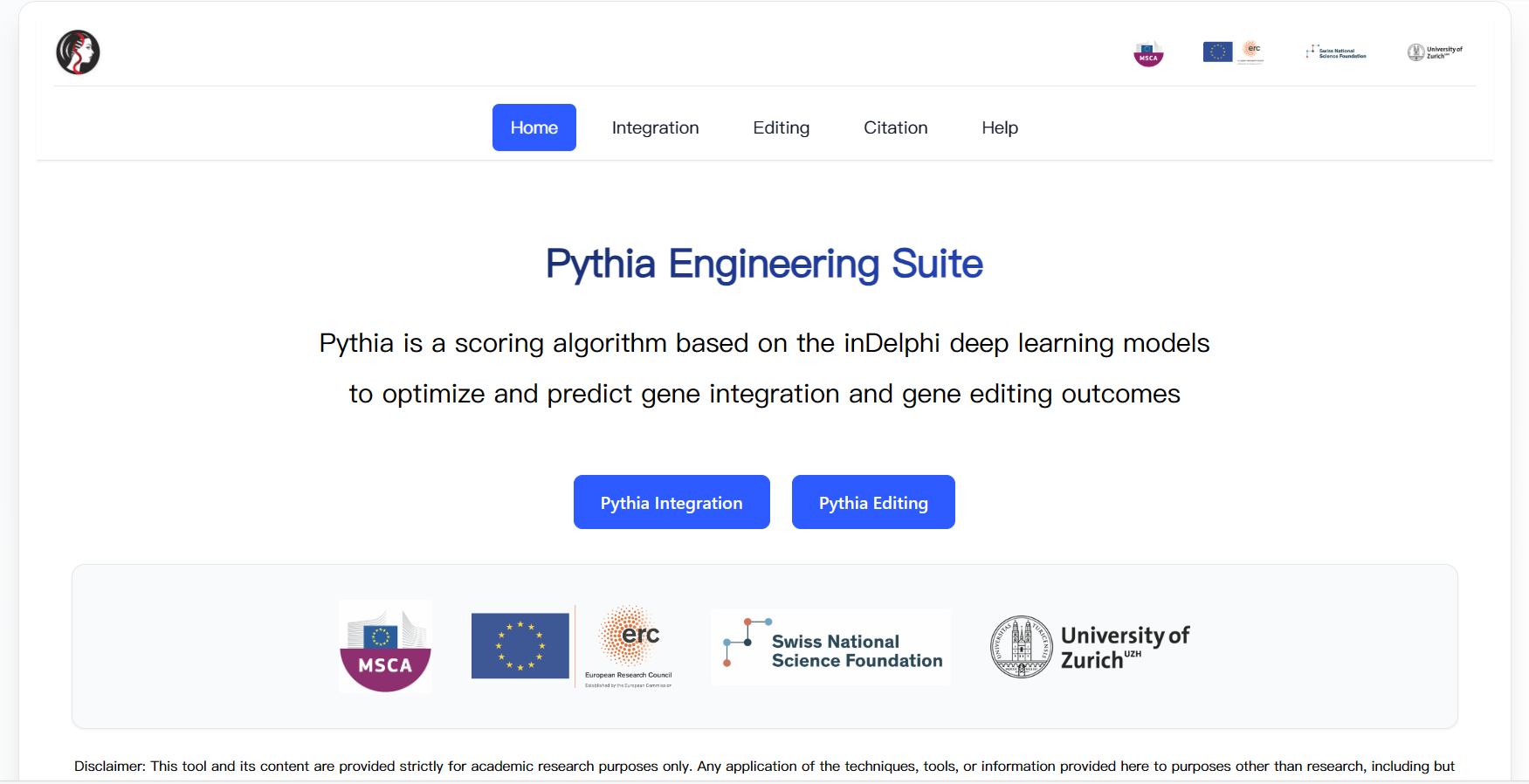
Pythia则是基于inDelphi深度学习模型的评分算法,专门用于优化和预测基因整合和基因编辑结果。作为inDelphi的延伸应用,Pythia提供了更加用户友好的界面和专门化的功能模块。它提供两个核心工具:Pythia Integration用于设计和优化基因整合策略,而Pythia Editing则用于预测和优化基因编辑结果。
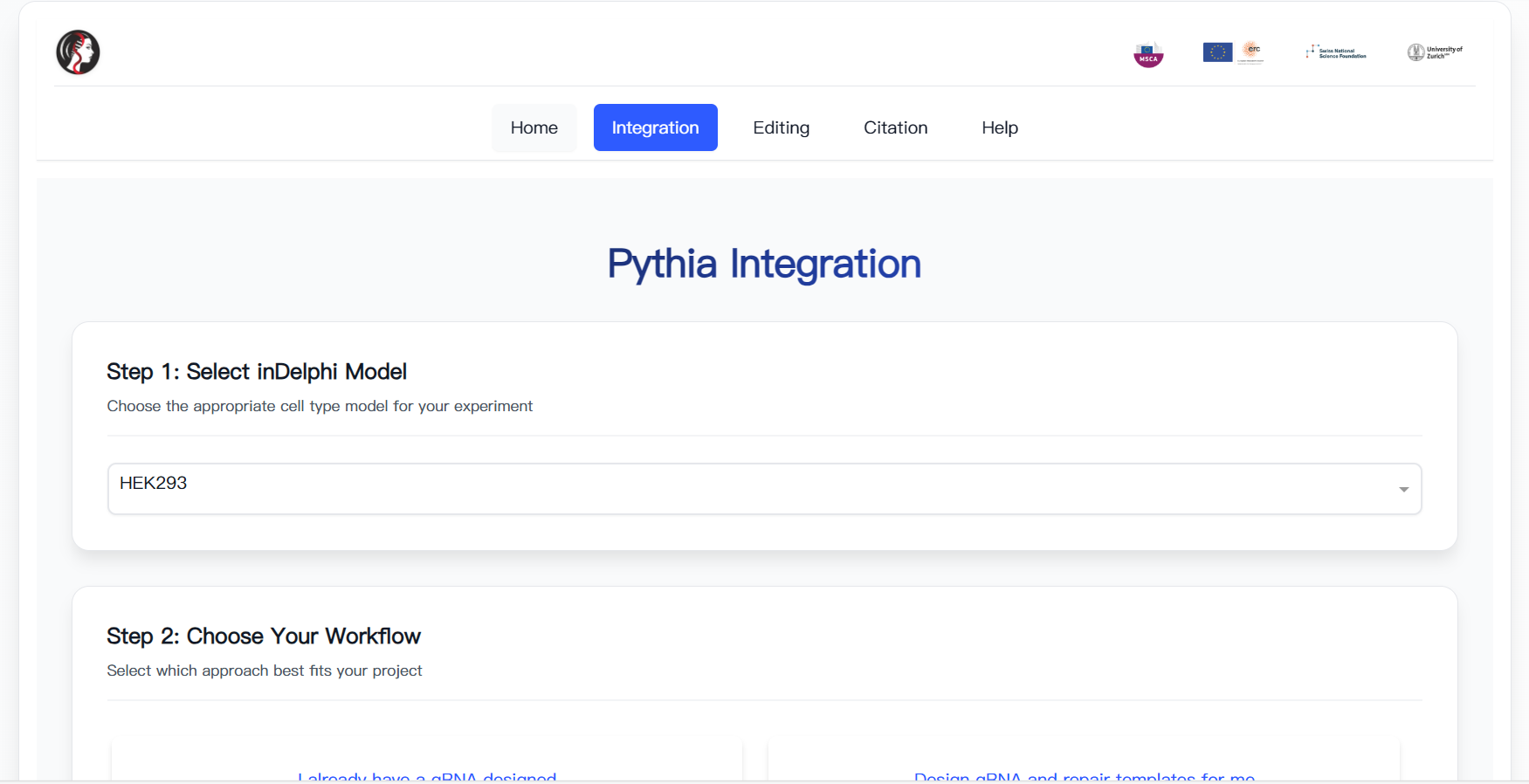
使用Pythia的流程相对简单直观。用户首先从导航菜单选择适当的工具,然后从下拉菜单选择细胞系模型,接着按要求格式输入遗传序列,点击提交运行分析,最后查看结果并根据需要下载。不过使用时需要注意,序列中只能使用ACTG字符,结果仅供学术研究使用,处理时间可能因序列复杂性而异。
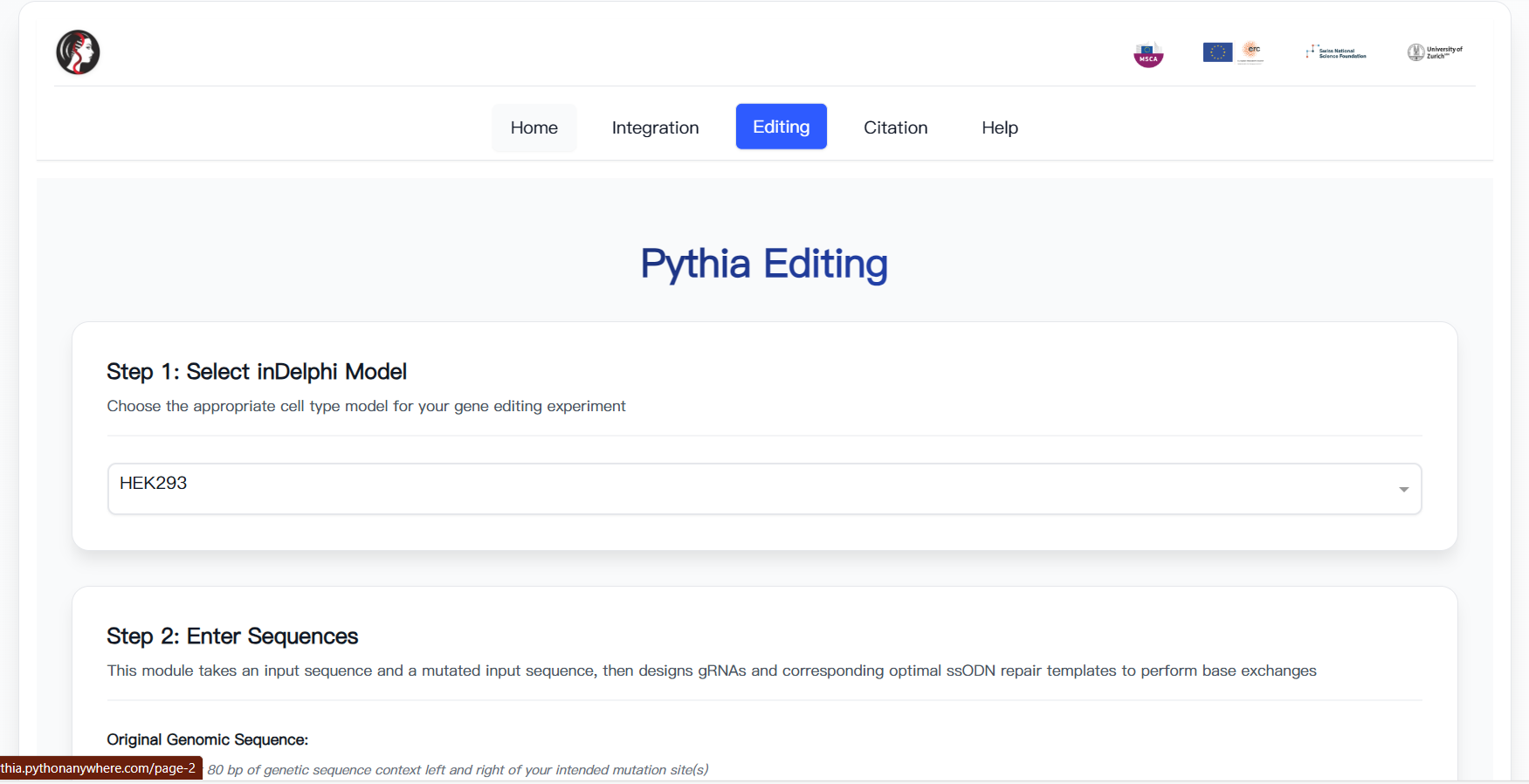
这两个工具之间存在着密切的协同关系。Pythia作为inDelphi的延伸应用,继承了inDelphi的核心算法和预测能力,同时提供了更加专门化和用户友好的界面。inDelphi提供了底层的科学原理和详细的预测模型,而Pythia则将这些复杂的科学原理转化为易于使用的工具。在应用场景上,inDelphi更适合需要深入了解DNA修复机制和进行详细预测分析的研究人员,而Pythia则更适合需要快速获得优化建议和预测结果的用户。
这两个工具共同为基因编辑研究提供了重要的计算支持。它们通过预测编辑结果,帮助研究人员选择最优的gRNA,从而提高实验设计效率,减少试错过程,节省实验资源。同时,它们提供的科学依据也增强了实验结果的可重复性,为治疗性基因编辑提供了重要的理论支持。
inDelphi和Pythia代表了基因编辑预测工具的重要发展方向,它们通过深度学习和大数据分析,为研究人员提供了强大的预测能力。inDelphi作为基础研究工具,提供了深入的科学洞察;而Pythia作为应用工具,将这些科学发现转化为实用的解决方案。随着基因编辑技术的不断发展和应用领域的扩大,这类预测工具将在基础研究、药物开发、基因治疗等领域发挥越来越重要的作用。未来,我们可以期待这些工具在准确性、适用范围和用户友好性方面继续改进,为生命科学研究提供更加强大的支持。
