从拟南芥到线虫:我的生物信息学多组学实操笔记
开篇:为什么选拟南芥 + 线虫练手?
作为生物信息学新手,选择模式生物练习能少走 90% 的弯路!拟南芥(Arabidopsis thaliana)和秀丽隐杆线虫(Caenorhabditis elegans)的核心优势:
- 基因组小(拟南芥~125Mb,线虫~100Mb),分析速度快,降低算力门槛
- 注释信息极完善(TAIR 数据库 / Ensembl Metazoa),结果验证方便
- 公开数据丰富(GEO/SRA 数据库海量测序数据),无需自己测序即可练手
- 覆盖植物 + 动物两类体系,流程通用性强,后续迁移到其他物种更轻松
接下来,我会按「测序原理→单一组学流程→多组学整合」的逻辑,分享实操中的关键步骤、工具选择和避坑指南~
一、先搞懂:测序原理是所有分析的基础
不管哪类组学,核心都是「将生物分子信号转化为测序数据」,新手不用深挖仪器细节,重点掌握这 2 点:
1. 主流测序平台(对应分析工具选择)
| 平台 | 优势 | 适用组学 | 拟南芥 / 线虫练习数据推荐 |
| Illumina | 短读长(100-150bp)、高通量、低成本 | 基因组重测序、转录组、表观组 | 优先选(数据量足、工具成熟) |
| PacBio/ONT | 长读长(10kb+)、无 GC 偏好 | 基因组组装、结构变异分析 | 进阶练习(补全短读长缺口) |
2. 核心概念(避免分析时踩坑)
- 双端测序(Paired-end):同一 DNA 片段两端测序,拼接 / 比对更准确(几乎所有组学都优先选双端数据)
- 测序深度(Depth):拟南芥 / 线虫练习时,基因组≥30×、转录组≥20M reads 即可(深度不够会导致结果假阴性)
- 质量值(Q30):≥85% 为合格数据(用 FastQC 检测,低于则需过滤)
二、单一组学实操流程(拟南芥 vs 线虫关键差异)
每个流程按「数据质控→核心分析→结果可视化」拆解,工具以「开源免费 + 新手友好」为原则推荐~
1. 基因组学分析(目标:组装 / 重测序变异检测)
核心流程:
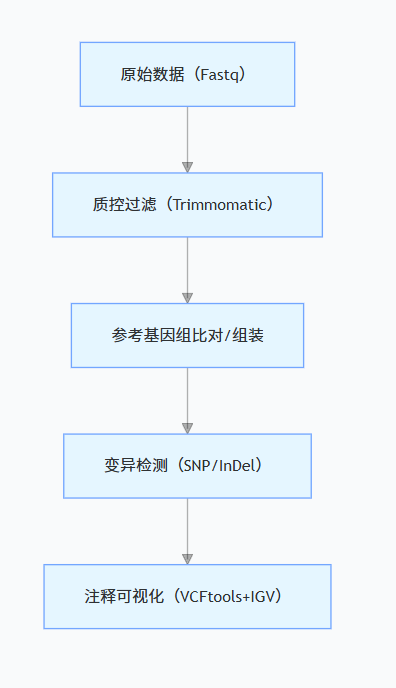
拟南芥 vs 线虫实操差异:
| 步骤 | 拟南芥(TAIR10 参考基因组) | 线虫(WS277 参考基因组) |
| 参考基因组获取 | TAIR 数据库直接下载(含注释文件) | Ensembl Metazoa 下载(需同步下载 GTF 文件) |
| 组装工具 | 短读长:SPAdes;长读长:Canu | 同拟南芥(基因组结构简单,组装难度低) |
| 变异注释 | SnpEff(加载 TAIR10 数据库) | SnpEff(加载 WBcel235 数据库) |
新手避坑:线虫有 2 个染色体版本(WS277/WBcel235),分析全程需保持版本一致!
2. 转录组学分析(目标:差异表达基因筛选)
核心流程(mRNA-seq):
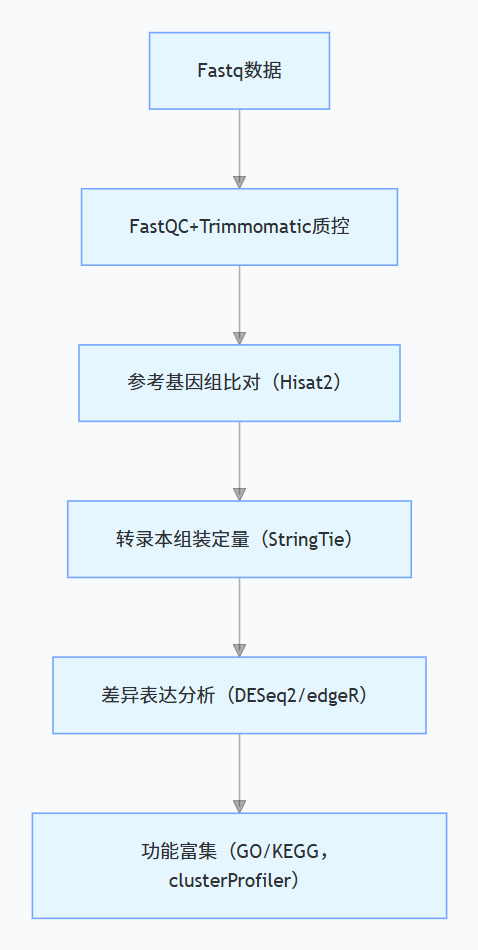
拟南芥 vs 线虫关键差异:
- 功能富集数据库:拟南芥用 TAIR GO/KEGG,线虫用 WormBase GO+Reactome 通路
- 特殊注意:线虫存在大量交替剪切本,定量时需用--eB参数保留可变剪切信息
拓展:单细胞转录组(10x Genomics)
核心工具:Cell Ranger(数据拆分→比对→定量)→ Seurat(细胞分群→差异基因→可视化)
- 拟南芥练习数据:GEO accession GSE152049(根组织单细胞数据)
- 线虫练习数据:GEO accession GSE138834(胚胎发育单细胞数据)
- 关键技巧:单细胞数据过滤标准(线粒体基因比例<10%,基因数 200-5000),拟南芥细胞异质性比线虫低,分群时可降低分辨率参数
3. 表观组学分析(以 ChIP-seq/ATAC-seq 为例)
核心流程:
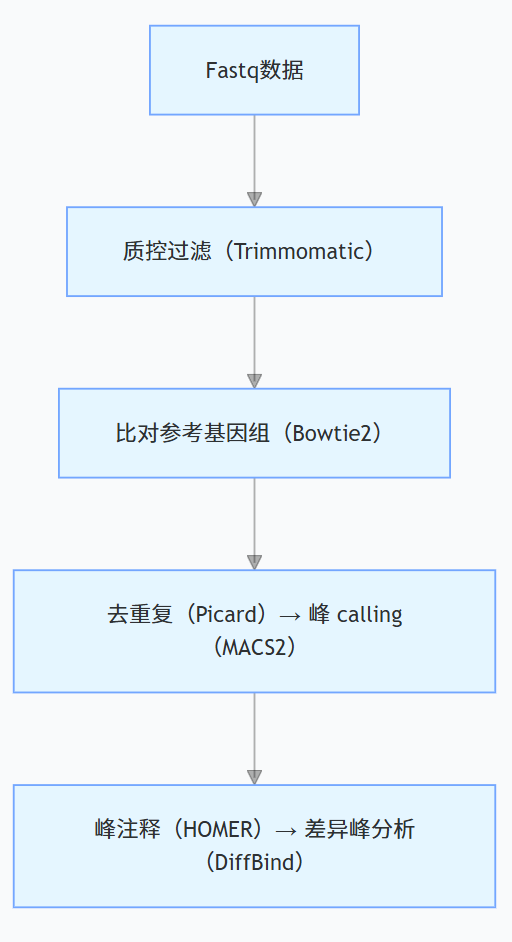
拟南芥 vs 线虫差异:
- ChIP-seq 抗体选择:拟南芥常用 H3K4me3(激活标记),线虫常用 H3K27me3(抑制标记)
- ATAC-seq:拟南芥细胞核提取难度高,公开数据较少,优先选线虫练习(GEO GSE162637)
4. 蛋白组学 / 代谢组学分析(计算机分析核心:数据解析)
蛋白组学(LC-MS/MS 数据):
- 核心工具:MaxQuant(数据检索)→ Perseus(质控 + 差异蛋白筛选)→ STRING(蛋白互作网络)
- 数据库选择:拟南芥用 UniProt Arabidopsis thaliana,线虫用 UniProt Caenorhabditis elegans
- 关键:导入数据时需匹配「物种特异性数据库」,否则鉴定率会大幅下降
代谢组学(LC-MS/GC-MS 数据):
- 核心工具:XCMS(峰提取→峰对齐)→ CAMERA(代谢物注释)→ MetaboAnalyst(差异代谢物 + 通路分析)
- 练习技巧:用拟南芥胁迫响应代谢组数据(GEO GSE183947),线虫代谢组数据较少,可先用模拟数据练习流程
5. 宏基因组学分析(目标:群落组成 + 功能注释)
核心流程:
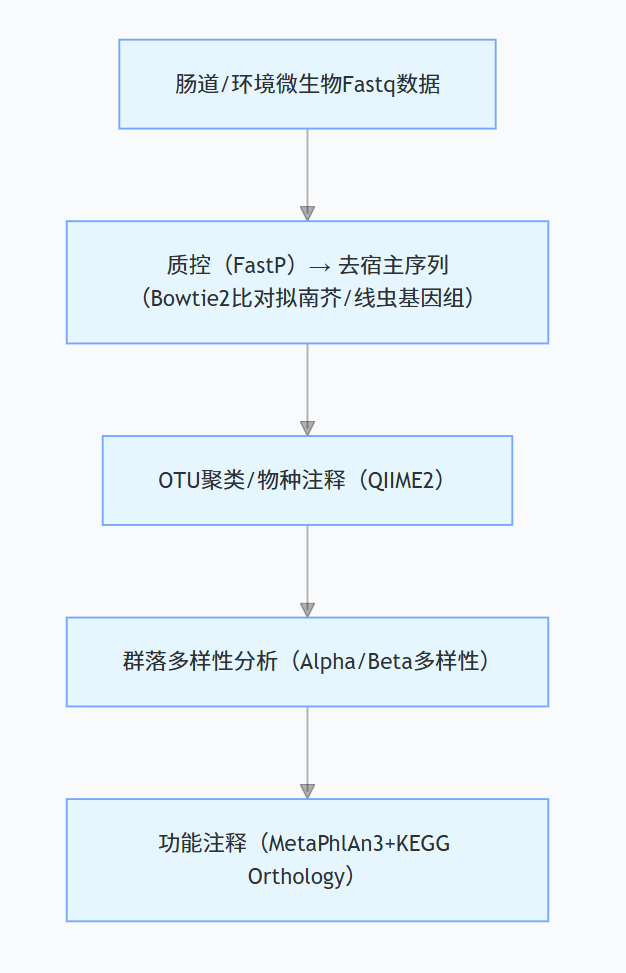
练习案例:
- 拟南芥根际微生物组(GEO GSE173934):重点分析植物 - 微生物互作相关功能基因
- 线虫肠道微生物组(GEO GSE127830):关注微生物对 nematode 发育的影响
三、多组学整合分析:从「单一维度」到「全景解析」
单一组学只能看到局部,整合分析才是生物信息学的核心价值!以「拟南芥盐胁迫响应」和「线虫衰老机制」为例,分享 2 种实用整合思路:
1. 横向整合(同一物种,多组学联动)
案例:拟南芥盐胁迫响应
- 数据组合:转录组(差异基因)+ 代谢组(差异代谢物)+ 表观组(ChIP-seq H3K4me3 峰)
- 整合逻辑:
- 用 WGCNA 构建转录组 - 代谢组共表达网络,筛选核心模块
- 验证核心模块基因是否与 H3K4me3 差异峰重叠(表观调控→转录→代谢的调控链)
- 用 OmicsNet 可视化多组学关联网络
2. 纵向整合(跨物种,核心通路对比)
案例:拟南芥 vs 线虫的氧化应激响应
- 数据组合:两者的转录组 + 蛋白组差异结果
- 整合逻辑:
- 分别富集两者的氧化应激相关通路(GO:0006979)
- 用 Venny 绘制差异基因 / 蛋白交集图,筛选保守通路(如 MAPK 信号通路)
- 用 Cytoscape 构建跨物种调控网络,对比植物和动物的应激响应差异
核心工具推荐:
- 入门级:OmicsNet(网页工具,无需编程)、clusterProfiler(R 包,功能富集整合)
- 进阶级:iDEP(多组学一站式分析)、WGCNA(共表达网络分析)
四、新手实操心得 & 资源推荐
1. 必备工具安装(少走弯路)
- 环境管理:Miniconda(统一管理软件包,避免版本冲突)
- 可视化:R(ggplot2/ggpubr)、Python(matplotlib/seaborn)、IGV(基因组可视化)、Circos(多组学环形图)
2. 关键资源网站
- 参考基因组 / 注释文件:TAIR(拟南芥)、WormBase(线虫)、Ensembl Metazoa
- 公开数据:GEO(https://www.ncbi.nlm.nih.gov/geo/)、SRA(https://www.ncbi.nlm.nih.gov/sra/)
- 工具教程:Bioconductor(R 包教程)、Qiime2 官方文档、Seurat 官方 vignettes
3. 避坑指南
- 所有分析前先「统一版本」:参考基因组版本、注释文件版本、工具版本必须一致
- 数据质控是基础:FastQC 检测不合格的 reads 一定要过滤,否则会导致后续分析全错
- 拟南芥 / 线虫的特异性:拟南芥有叶绿体基因组,分析时需排除;线虫有性别差异(雌雄同体 / 雄性),数据分组时需注意
结尾:从练习到实战的小建议
先专注 1-2 个组学练熟(比如先吃透转录组),再拓展到多组学;用拟南芥 / 线虫练完流程后,可尝试迁移到自己研究的物种(比如作物 / 人类疾病模型)。生物信息学的核心是「解决生物学问题」,工具只是手段,一定要结合具体生物学背景解读结果~
