一文读懂分子结合位点的预测:为双荧光素酶实验铺路
双荧光素酶实验可以验证转录因子和启动子、miRNA和mRNA、miRNA和lncRNA、miRNA和circRNA的结合验证。然而,验证它们之间的结合需要知道确切的结合位点,如何才能找到准确可靠的结合位点是实验成功的关键。今天,小医给您介绍一下四大权威数据库,找到那条最关键的线索——潜在的分子结合位点。
01 预测转录因子与启动子的结合——JASPAR数据库
1.访问数据库,在浏览器中输入网址:https://jaspar.elixir.no,进入到JASPAR首页。
2.在网站顶部的Search输入框中输入转录因子名字,以人源的转录因子p65为例,然后点击search。
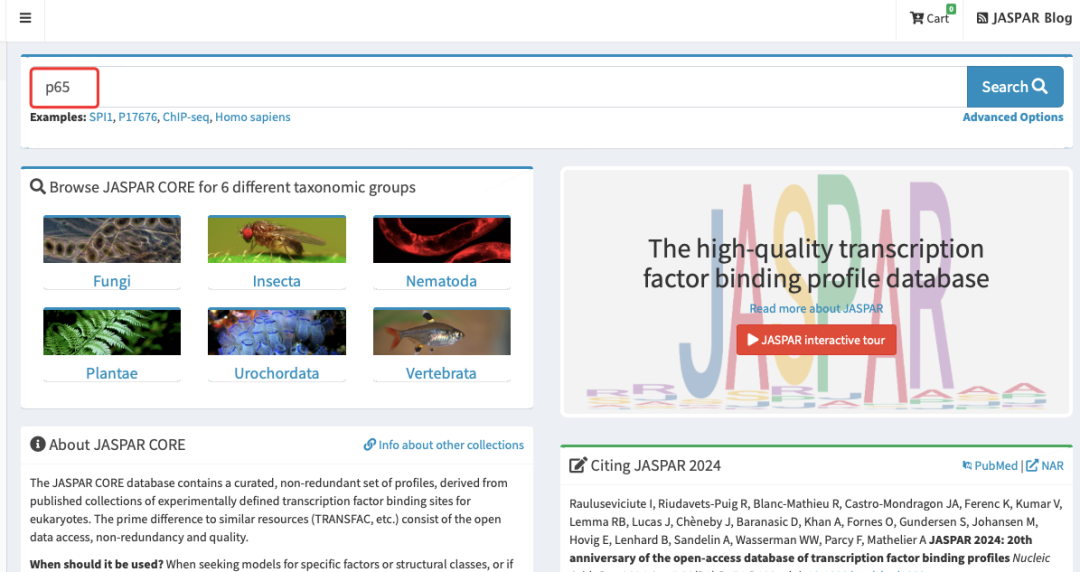
3.选择人源“Homo sapiens”,然后再点击右侧的Scan。
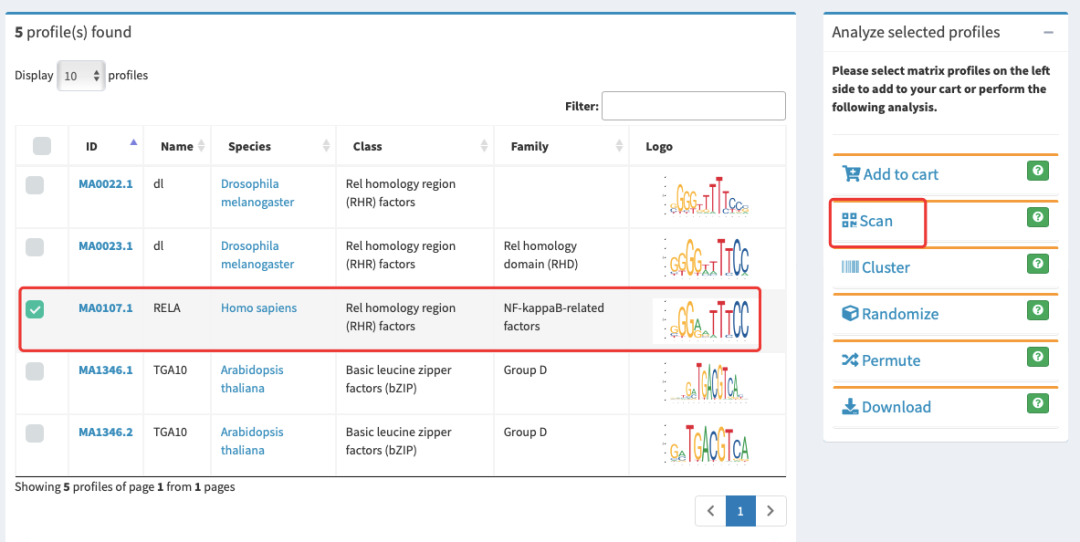
4.点击Scan后会展开一个序列的输入框,要求输入FASTA格式的启动子序列。推荐使用NCBI数据库。通常,我们选取转录起始位点上游约2000 bp的序列,启动子序列查找可查看我司公众号以往文章“NCBI保姆级使用教程(包含如何查找CDS、启动子、5'UTR和3'UTR、lncRNA序列)”,将这段序列复制下来,将您复制的启动子序列(FASTA格式)粘贴进去。得分阈值是一个重要的过滤器,建议保持默认的“Relative Profile Score Threshold: 80%”。这个值越高,预测结果越严格,假阳性越少,但可能会漏掉一些真实位点,点击Scan。
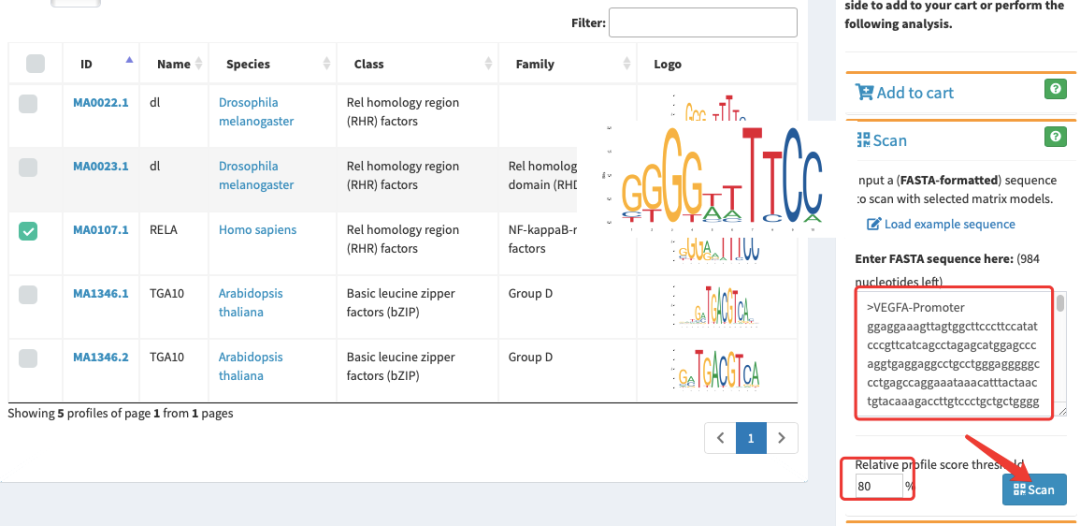
5.运行与分析结果:TF Name:转录因子名称;Score:匹配分数,越高表示结合可能性越大;Relative Score:相对分数(与最高可能得分的比值),我们刚才设置的80%阈值就是针对这个分数;Start / End:结合位点在您输入的序列中的起止位置。
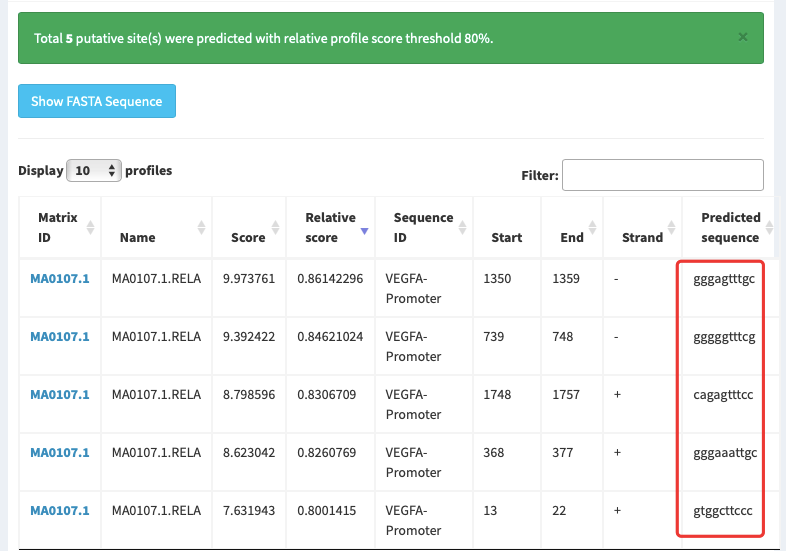
实战提示:将得分最高的几个转录因子作为候选,然后查阅文献,看是否有相关研究支持,最终确定双荧光素酶实验要验证的转录因子。
02 预测miRNA与mRNA的结合——miRDB数据库
1.访问miRDB数据库,数据库目前可以预测人、大小鼠、狗、鸡的物种,在浏览器中输入网址:http://mirdb.org/,进入首页。
2.两种预测模式:
模式一:由miRNA找靶基因,在首页“Search by miRNA name”框中,输入miRNA名称(如人源miR-182-5p),物种选择“Human”,最后点击“Go”;
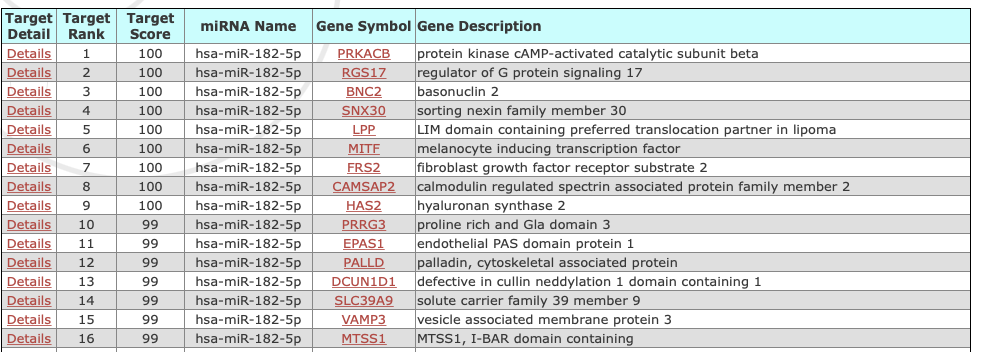
结果列表会显示所有预测的靶基因,按靶标评分降序排列。评分越高(最高100分),预测可靠性越高。通常认为评分>80的靶标质量很好。
模式二:由基因找miRNA,在首页“Search by Gene target”框中,输入基因的官方符号(如“PTEN”),选择物种”human”,点击“Go”。
结果列表会显示所有可能靶向该基因的miRNA,同样按评分排序。
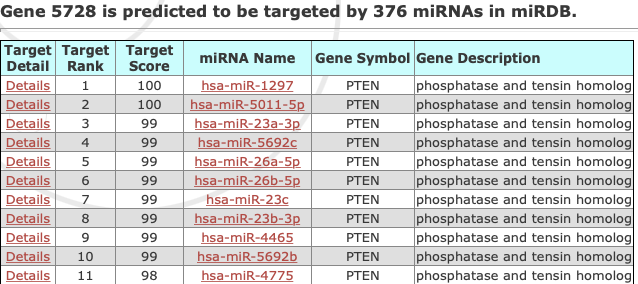
3.点击任意一个靶标对的”Details”,可以看到详细的预测信息:
Target Score:总评分;
Seed Match:种子区的匹配类型,这是miRNA结合的关键;
Target Site Context: 结合位点在mRNA 3‘UTR上的具体位置;

实战提示:miRDB的预测结果非常直观,是设计miRNA-mRNA双荧光素酶实验的首选起点。优先选择靶标评分高且种子区匹配完美的对子进行实验。
03 预测miRNA与lncRNA的结合——ENCORI (starBase)数据库
1.访问数据库,在浏览器中输入网址:https://rnasysu.com/encori/。
2.选择查询模块,在首页的“miRNA-Target”模块中,选择”miRNA-lncRNA”。

3.进入新的页面后,可以通过以下2种方式进行预测:
方式一(推荐):在“miRNA”框中输入您关注的miRNA(如“hsa-miR-100-5p”),页面就会更新出预测到有结合位点的lncRNA。
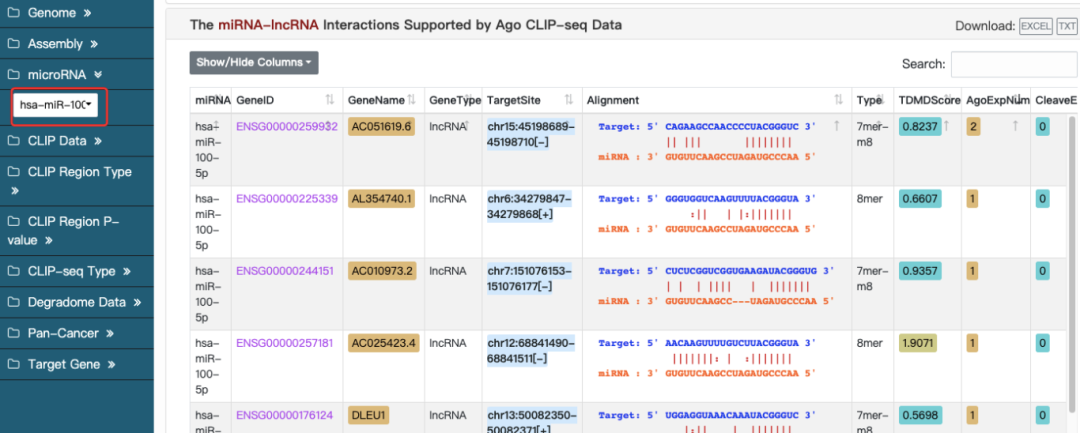
方式二:如果您想筛选目标lncRNA的所有结合miRNA,只需在Target Gene输入lncRNA名称如”MALAT1”,点Submit即可。
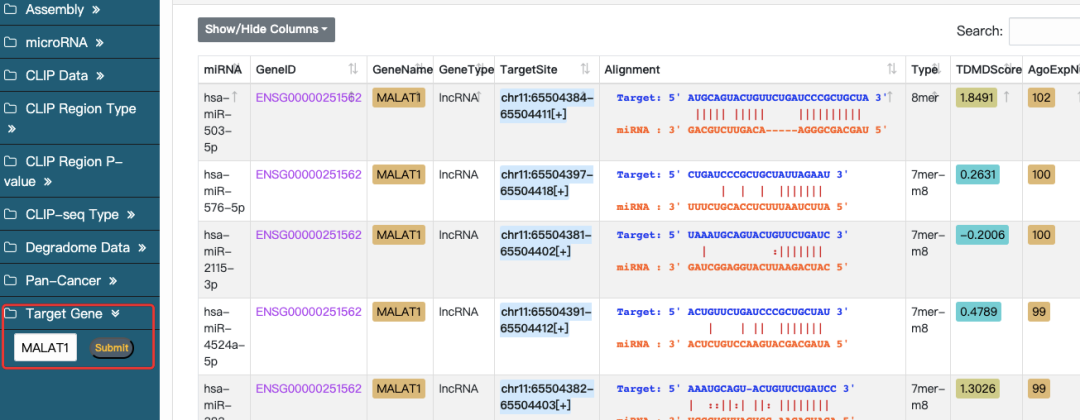
4.ENCORI的强大之处在于它整合了CLIP-seq实验数据,结果非常可靠。重点关注Pan-Cancer(CLIP Data)这个标签页下的结果。如果存在结合,您会看到来自不同CLIP数据库(如starBase, clipdb等)的支持证据,这为您选择后续的实验细胞系提供了重要参考。
04 预测miRNA与circRNA的结合——CircInteractome数据库
1.访问数据库,在浏览器中输入网址:https://circinteractome.irp.nia.nih.gov。

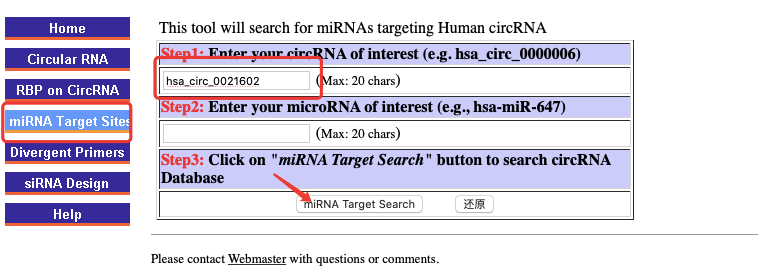
2.点击"miRNA Target Sites”,进入到新的页面后,输入目标circRNA如“hsa_circ_0021602”,然后点击miRNA Target Search。
4.解读结果:表格中会显示miRNA的名称、结合位点的起止位置、以及种子区的匹配序列。您可以直接使用这些信息来设计包含该位点的报告基因质粒。
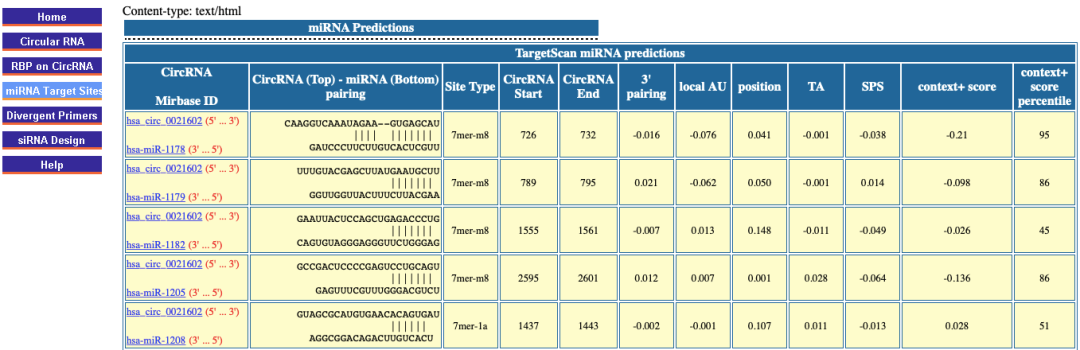
实战提示:CircInteractome是circRNA研究的利器,它不仅能预测miRNA结合,还能帮助设计用于验证circRNA特性的引物(如divergent primers)。对于双荧光素酶实验,直接使用它预测的位点序列即可。
05 总结:从预测到实验的完美动线
1.提出科学问题:例如lncRNA H19是否通过吸附miR-101来发挥作用?
2.数据库预测:使用ENCORI数据库,输入H19和miR-101,查看是否有预测和支持数据。
3.设计报告基因
野生型:根据数据库预测的结合位点序列,合成包含该位点的H19片段,插入报告质粒;
突变型:将结合位点中的关键种子区进行点突变,构建突变体质粒作为对照。
4.执行双荧光素酶实验:共转染报告质粒与miRNA,检测荧光活性变化。
通过以上清晰的步骤,您就可以独立完成从生物信息学预测到分子生物学实验假设构建的全过程。这些数据库是您探索基因调控网络的强大罗盘,祝您在科研的海洋中顺利航行!
