Genome Med|RAG-HPO做表型注释:学习一下大语言模型怎么作为发文思路
今天给大家介绍一篇做工具的文章,教大家怎么去模仿这种思路产出自己的文章,结合大语言模型去写工具也是可以出文章的,不论是集成化的docker本地部署,或者缩减点做个shiny,甚至是开放源代码都是可以的。

- 标题:利用检索增强生成通过大型语言模型改进自动深度表型分析
- DOI:10.1186/s13073-025-01521-w
摘要
RAG-HPO,一种基于 检索增强生成 (RAG) 的表型提取工具,利用 LLaMa-3.1 70B 模型结合动态向量数据库对临床文本进行 HPO 术语 分配。在 112 份病例报告(1792 个人工标注术语)的基准测试中,RAG-HPO 显著优于 Doc2HPO、ClinPhen 和 FastHPOCR,达到 0.81 精度、0.76 召回率与 0.78 F1(p<0.00001),并有效减少幻觉和无关匹配。假阳性多数为目标术语的上位词,仍具一定临床相关性。RAG-HPO 的 高准确性与适应性 不仅提升了表型提取的可靠性,还加速了 罕见病遗传机制的发现与临床基因组学应用,为表型驱动的基因诊断提供了新的解决方案。
部分结果
在基于病例队列的对比分析中,RAG-HPO 在多种 LLM 后端配置下均展现出优于传统工具的性能(表 4)。其中,LLaMA-3.1 70B + RAG-HPO 取得了最高的平均精度 (0.81)、召回率 (0.76) 和 F1 分数 (0.78),显著超过 FASTHPOCR、Doc2HPO 与 ClinPhen 等基于规则的表型识别工具。虽然不同 LLM 的规模和架构对性能有所影响,但整体趋势显示 RAG 框架有效缓解了误配和漏配问题,尤其在减少假阴性和提升上下文相关性方面具有优势。
| 模型 / 工具 | True positive | False positive | False negative | 平均精度 (Precision) | 平均召回率 (Recall) | 平均 F1 |
|---|---|---|---|---|---|---|
| LLaMA-3.1 70B + RAG-HPO | 1333 | 315 | 461 | 0.81 | 0.76 | 0.78 |
| LLaMA-3 70B + RAG-HPO | 1083 | 486 | 711 | 0.71 | 0.61 | 0.64 |
| LLaMA-4 Scout + RAG-HPO | 1257 | 634 | 537 | 0.65 | 0.69 | 0.66 |
| LLaMA-3 8B + RAG-HPO | 852 | 727 | 942 | 0.53 | 0.46 | 0.48 |
| Misra 24B + RAG-HPO | 581 | 174 | 1213 | 0.73 | 0.31 | 0.42 |
| Deepseek-R1 + RAG-HPO | 682 | 1944 | 1112 | 0.15 | 0.35 | 0.20 |
| FASTHPOCR | 816 | 726 | 978 | 0.53 | 0.45 | 0.49 |
| Doc2HPO | 634 | 260 | 1159 | 0.71 | 0.35 | 0.47 |
| ClinPhen | 635 | 370 | 1159 | 0.63 | 0.35 | 0.45 |
对比了各工具输出与人工注释参考集的一致性,结果显示 RAG-HPO + LLaMA-3.1 70B 能够在所有配置中恢复最多的真值术语,并保持最低的误报率,实现了最佳的整体误差平衡。相比之下,FastHPOCR 尽管检索到的正确术语数最多,但伴随极高的假阳性比例;而 ClinPhen 与 Doc2HPO 虽然误报率较低,却仅能捕获黄金标准中约三分之一的表型,存在明显漏检。总体而言,传统工具在准确性和覆盖率上均显著落后于 RAG-HPO,后者在减少错误分配的同时提升了表型恢复能力,凸显其在自动化 HPO 任务中的优势。
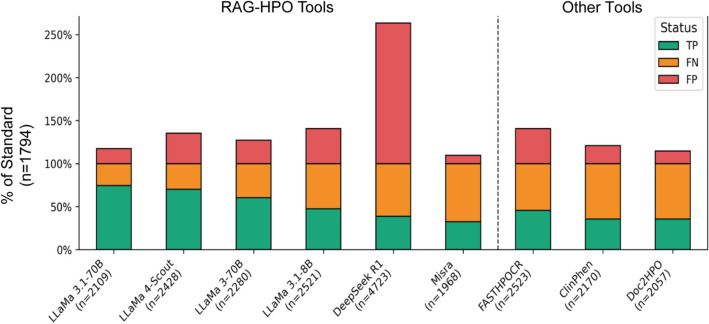
按器官系统与节点深度的性能评估
通过 二项式广义估计方程 (GEE) 并在患者水平聚类的条件下建模,我们发现 器官系统对 HPO 分配准确率的影响有限(图 6A),仅在心血管、泌尿生殖和头颈部表型中表现出略高的正确分配概率(比值比约 8–11),其余系统大多接近 1。随着 节点深度增加,所有工具的性能均呈单调下降趋势(图 6B),预测命中率从浅层节点(≤3)向深层节点(≥10)逐渐降低。最显著的差异来自 工具选择(图 6C):几乎所有 RAG-HPO 配置均显著优于 FastHPOCR、ClinPhen 与 Doc2HPO,其中 RAG-HPO + LLaMA-3.1 70B 在全深度范围内始终保持最高的预测命中概率,凸显其在复杂与特异性表型识别中的优势。
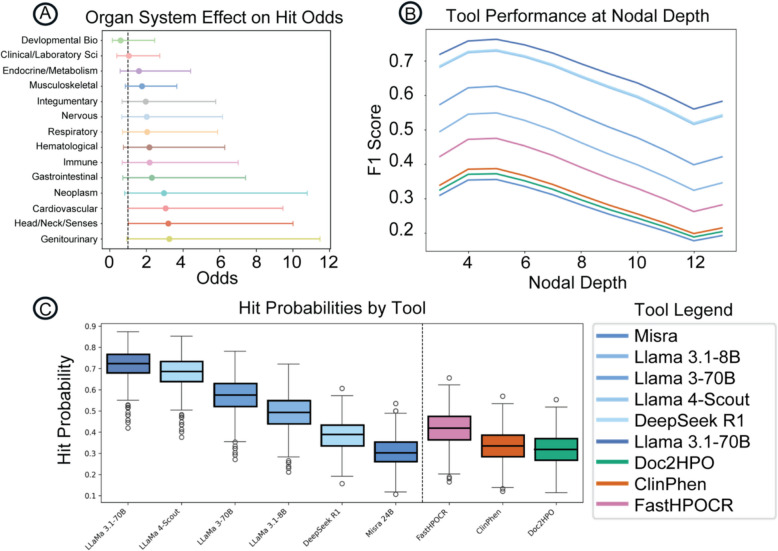
假阳性结果的分类与意义
在 HPO 分析中,我们将假阳性术语进一步细分为 直接祖先、间接相关(远亲)、完全无关以及幻觉 四类,以揭示其形成原因和潜在价值(图 7A)。结果显示,RAG-HPO + LLaMA-3.1 70B 共产生 315 个假阳性,其中 绝大多数为目标术语的祖先或远亲(95%),仅 14 个(4%)为不相关术语,且真正的幻觉仅 1 个(<1%)(图 7B)。其他 RAG 配置呈现类似模式,但与 LLaMA-3 70B 或 LLaMA-4 Scout 等较低对齐模型组合时,不相关术语比例更高。相比之下,基于规则的工具(FASTHPOCR、ClinPhen、Doc2HPO)虽然没有幻觉,但远亲术语依然占 62–79%。值得注意的是,RAG-HPO 的不相关术语并非源于 LLM 的自由生成,而主要由语义相似性检索结果不佳所致,因 LLM 必须在返回列表中强制选择术语,偶尔会导致语义不符的分配。整体来看,大多数假阳性依然提供了临床相关性,强调了根据下游应用需求灵活选择“最具体”还是“较广泛但相关”术语的重要性。
解读开源文件
Github:https://github.com/PoseyPod/RAG-HPO
我简化一下,主要分成两大部份:
1.HPO 向量数据库构建
# hpo_vector_builder.py
"""
步骤:下载/刷新 → 提取术语信息 → 生成向量 → 保存
"""
import requests, time, re, json
import pandas as pd, numpy as np
from pathlib import Path
import pronto
from sentence_transformers import SentenceTransformer# 自动下载 HPO
def initialize_hpo(obo_url="https://purl.obolibrary.org/obo/hp.obo", obo_path="hp.obo"):obo_file = Path(obo_path)if not obo_file.exists():print("Downloading HPO...")resp = requests.get(obo_url); resp.raise_for_status()obo_file.write_text(resp.text, encoding="utf-8")return pronto.Ontology(obo_file)# 构建 DataFrame(只保留主要字段)
def build_dataframe(ont):records = []for term in ont.terms():records.append({"hp_id": term.id,"label": term.name,"definition": term.definition or "","synonyms": [s.description for s in term.synonyms]})return pd.DataFrame(records)# 向量化 + 保存
def vectorize_and_save(df, out_meta="hpo_meta.json", out_vec="hpo_emb.npz"):model = SentenceTransformer("pritamdeka/SapBERT-mnli-snli-scinli-scitail-mednli-stsb")embeddings = model.encode(df["label"].tolist(), convert_to_numpy=True)np.savez_compressed(out_vec, emb=embeddings.astype(np.float16))df.to_json(out_meta, orient="records")print(f"Saved {len(df)} terms → {out_meta}, {out_vec}")if __name__ == "__main__":ont = initialize_hpo()df = build_dataframe(ont)vectorize_and_save(df)
2.随机打印 HPO 术语 测试
# hpo_random_term.py
"""
简化版:随机抽取一个 HPO 术语并打印其基本信息
"""
import random, requests, time
from pathlib import Path
import prontodef initialize_hpo(obo_url="https://purl.obolibrary.org/obo/hp.obo", obo_path="hp.obo"):if not Path(obo_path).exists():print("Downloading HPO...")resp = requests.get(obo_url); resp.raise_for_status()Path(obo_path).write_text(resp.text, encoding="utf-8")return pronto.Ontology(obo_path)def print_random_term(ont):term = random.choice(list(ont.terms()))print("\n=== Random HPO Term ===")print("ID:", term.id)print("Name:", term.name)print("Definition:", term.definition)print("Synonyms:", [s.description for s in term.synonyms])print("Xrefs:", [str(x) for x in term.xrefs])print("========================")if __name__ == "__main__":ont = initialize_hpo()print_random_term(ont)
怎么模仿
这是官网的学术海报,通过这一张图可以看看,我们怎么去模仿学习做这类研究。

基于AI的总结:
这张海报展示的研究思路非常清晰,可以分为 问题提出 → 方法设计 → 性能评估 → 结果与讨论 → 应用前景 五个环节,我们完全可以模仿这种逻辑来利用 大语言模型 (LLM) 开发工具并产出论文。
-
研究问题:指出传统工具(如 Doc2HPO、ClinPhen)在表型识别中的局限,缺乏上下文理解、假阳性多、需要人工审核。引入 LLM 的潜力,但同时说明其缺点(幻觉、不精确),从而提出改进的需求。
-
方法设计:构建一个新框架(RAG-HPO),将 LLM 与检索增强生成 (RAG) 相结合。具体流程是:
- 从临床文本中提取表型短语
- 使用 向量数据库(包含 54,000+ HPO 短语)进行语义检索
- 将最相关的候选结果反馈给 LLM,用于最终的 HPO 术语分配
→ 这样既利用了 LLM 的语境理解,又避免了幻觉输出。
-
性能评估:基于 真实病例报告队列(人工标注的金标准),与传统工具进行对比,评估指标包括 精度、召回率、F1 分数。结果显示 RAG-HPO + LLaMA-3.1 70B 在 三项指标上均优于对照工具,尤其是假阳性显著减少。
-
结果与讨论:
- 分析假阳性类别(祖先、远亲、不相关、幻觉),证明大多数假阳性仍具临床价值
- 分层分析(按器官系统、节点深度),说明工具在复杂语义和深层术语上的优势
- 指出 RAG-HPO 可迭代升级,随着 LLM 架构和 HPO 数据库的扩展,性能会进一步提升。
-
应用前景:提出该工具可以加速 基因型-表型匹配、临床诊断和基因发现,并且通过开源(Github 发布)方便科研和临床转化。
🔑 我们可以模仿的地方:
- 研究切入点:找出某个生物医学领域中已有工具的痛点(如假阳性高、人工依赖强),提出 LLM + RAG/知识库 的改进方案。
- 方法框架:搭建一个流程,将 文本/数据输入 → 特征抽取 → 向量检索/知识对齐 → LLM 输出 → 指标评估。
- 数据来源:用公开数据库(病例报告、电子病历、文献摘要、基因组数据)建立评估集,并人工标注一部分作为金标准。
- 性能评估:与现有工具做横向比较,使用 Precision/Recall/F1 等标准指标,必要时做子分析(分层或分系统)。
- 文章结构:模仿这篇,把 背景 → 方法 → 结果 → 讨论 → 展望 清晰串联,最好配合图表(流程图、性能对比图、误差分析图)。
举一反三
例如我这边随意拟定三个方向:
方向一:免疫相关不良事件(irAE)表型智能挖掘
免疫检查点抑制剂(ICI)治疗相关的 免疫相关不良事件 (irAE) 表型复杂且异质性强,给临床评估和预后预测带来挑战。本课题拟构建基于 RAG-LLM 的智能表型识别工具,将临床病例文本自动映射到 HPO/MedDRA 术语,实现对 irAE 的标准化与结构化表征。该方法不仅能减少人工审核负担,还可用于建立系统的 irAE 表型数据库,从而支持 免疫治疗分型、风险预测与精准管理。
- 背景:irAE 的表型异质性大,传统字典工具难以捕获上下文信息。
- 方法:收集病例报告和电子病历,结合 irAE 术语库(如 CTCAE、MedDRA)与 RAG 检索机制,增强 LLM 的精确提取。
- 结果预期:构建 irAE 表型数据库,提升 irAE 风险预测与免疫治疗精准分型。
方向二:单细胞数据中的细胞死亡机制自动注释
新兴细胞死亡机制(如 铁死亡、杯凋亡、PANoptosis)在肿瘤免疫和炎症中发挥关键作用,但现有单细胞注释方法难以精确识别其特征。本课题提出开发结合 RAG 与 LLM 的细胞死亡注释工具,通过整合 死亡机制基因集与本体数据库,对单细胞差异表达基因和通路进行自动化标注。该框架将提高对细胞死亡亚型的识别能力,推动对 免疫微环境与程序性细胞死亡交叉机制 的深入理解。
- 背景:铁死亡、杯凋亡、PANoptosis 等新兴细胞死亡方式难以通过传统方法精确注释。
- 方法:构建死亡机制相关的基因集与本体数据库,通过向量化检索 + LLM 对差异基因/通路进行自动匹配。
- 结果预期:提升单细胞层面对细胞死亡亚型的识别能力,推动免疫微环境与死亡机制交叉研究。
方向三:NHANES 队列中的环境暴露–疾病表型映射
环境重金属和污染物暴露与多种慢性疾病风险密切相关,但大规模队列(如 NHANES)中表型与暴露指标的异构性限制了系统化分析。本课题计划建立 RAG-LLM 驱动的表型映射工具,将 NHANES 变量自动对齐到 标准化疾病本体(HPO/ICD-10),并结合暴露数据实现高通量表型挖掘。该方法可揭示 环境暴露–疾病表型–遗传背景 的复杂关联,为精准流行病学与公共卫生干预提供新思路。
- 背景:NHANES 包含丰富的临床表型和暴露指标,但变量异构,传统匹配依赖人工。
- 方法:利用 RAG-LLM 框架,将 NHANES 变量与标准化疾病/表型本体(HPO、ICD-10)自动对齐。
- 结果预期:实现高通量表型挖掘,揭示 环境暴露–疾病表型–遗传背景 的三重关联。
传统的 靶点挖掘套路 已被反复使用,不仅难以创新,还很难与高分文章竞争;如果缺乏新思路和优质数据,不妨尝试 基于大语言模型的工具开发。这类方法不同于传统深度学习框架,具备更高的灵活性与可扩展性,足以支撑 课题申报 或在 二区期刊 发文,完全绰绰有余。
