【论文阅读笔记】VeloCycle
文章目录
- 流形
- 似然
- 贝叶斯模型
- 变分分布(SVI)
- 变分分布(LRMN)
- 模型实现
- 估算恒定细胞周期速度的近似点
- 数据集
流形
-
通过单细胞RNA测序(scRNA-seq),得到一个细胞的基因数n∼104n \sim 10^4n∼104
-
ucu_cuc,scs_csc表示未剪切mRNA和已剪切mRNA的表达量,记yc=(uc,sc)y_c = (u_c, s_c)yc=(uc,sc),uc,sc∈Nnu_c, s_c \in \mathbb{N}^nuc,sc∈Nn
-
每个细胞的潜在坐标 xxx 映射到通过确定性函数 s(x)s(x)s(x)(其中 s 表示“剪接”)描述的剪接基因表达水平流形 MMM
-
根据问题的生物学结构选择流形拓扑。例如,给定一个周期性过程,如细胞周期,我们可以取 x∈S1x \in S_1x∈S1来体现周期性。
-
每个细胞ccc的测量值将通过真实的噪声模型与MMM上的相应位置相关联。在scRNA-seq的情形下,相关的噪声模型由负二项分布(NB)组成:
Ygc∼NB[yg(xc),αg]yg(xc)=E[Ygc]=(sg(xc),ug(xc))αg=(αgs,αgu)Y_{gc} \sim NB[y_g(x_c), \alpha_g] \\ y_g(x_c) = \mathbb{E}[Y_{gc}] = (s_g(x_c), u_g(x_c)) \\ \alpha_g = (\alpha^s_g, \alpha^u_g) Ygc∼NB[yg(xc),αg]yg(xc)=E[Ygc]=(sg(xc),ug(xc))αg=(αgs,αgu)
ccc是细胞,ggg是基因。这里假设了αg\alpha_gαg与xxx独立。 -
在高维基因表达空间中,我们希望有一个描述RNA速度ds~dt\frac{d\tilde{s}}{dt}dtds~的速率方程,该方程取决于剪接和非剪接RNA计数的预测:
ds~gdt=F(s~g,u~g)=βgu~g−γgs~g\frac{d \tilde{s}_g}{d t} = F(\tilde{s}_g, \tilde{u}_g) = \beta_g \tilde{u}_g - \gamma_g \tilde{s}_g dtds~g=F(s~g,u~g)=βgu~g−γgs~g
其中,s~g\tilde{s}_gs~g 和 u~g\tilde{u}_gu~g 分别是时间依赖的估计的剪接和未剪接RNA水平,βg\beta_gβg 和 γg\gamma_gγg 是基因依赖的RNA剪接和降解速率。
方程中的 FFF 不显式依赖时间,剪接和降解速率被视为常数。 -
假设:存在一个自洽且确定性的方程描述潜在空间 x(t) 的动力学:
dxdt=V(x)\frac{dx}{dt} = V(x) dtdx=V(x)
其中 V(x)V(x)V(x) 是潜在空间中的向量场。于是s~,u~\tilde{s}, \tilde{u}s~,u~通过xxx传递成为时间依赖的:
s~(t)=s(x(t))u~(t)=u(x(t))\tilde{s}(t) = s(x(t)) \\ \tilde{u}(t) = u(x(t)) s~(t)=s(x(t))u~(t)=u(x(t)) -
由上述假设可以得到流形限制条件下的RNA速度:
dsg(x(t))dt=(∇xsg)⋅V(x(t))=βgug(x(t))−γgsg(x(t)),∀g\frac{ds_g(x(t))}{dt} = (\nabla_x s_g) \cdot V(x(t)) = \beta_g u_g(x(t)) - \gamma_g s_g(x(t)), \ \forall g dtdsg(x(t))=(∇xsg)⋅V(x(t))=βgug(x(t))−γgsg(x(t)), ∀g
这里使用了链式法则。这个式子将左侧的低维流形拓扑与右侧的生物学连接起来。
βg\beta_gβg和γg\gamma_gγg是基因特异性剪接和降解率。值得注意的是,控制基因动力学的参数(βββ和γγγ)原则上也可能取决于xxx。
∇xs\nabla_x s∇xs 形成了切空间的 mmm 维基,V(x(t))V(x(t))V(x(t)) 提供了速度向量在该基中的分量。 -
我们可以通过这个式子估算生物过程的实际持续时间:
Δts0,s1=∫Γs0s11sds=∫Γx0x11V(x)dx=Δtx0,x1\Delta t_{s_0, s_1} = \int_{\Gamma^{s_1}_{s_0}} \frac{1}{s} ds = \int_{\Gamma^{x_1}_{x_0}} \frac{1}{V(x)} dx = \Delta t_{x_0, x_1} Δts0,s1=∫Γs0s1s1ds=∫Γx0x1V(x)1dx=Δtx0,x1
其中 Γx0x1\Gamma^{x_1}_{x_0}Γx0x1 是连接两个点 x0x_0x0 和 x1x_1x1 的轨迹 x(t)x(t)x(t),并使用了轨迹变量 s(x)s(x)s(x) 的变化。 -
假设MMM在拓扑上是一个圆,将坐标xxx写成φ∈S1\varphi \in S^1φ∈S1,于是动力学方程变成:
ddtsg(φ(t))=ddφsg(φ)ω(φ)=βgug−γgsgE[sgc]=sg(φc)=exp(∑fvgfζ~f(φc))\frac{d}{dt} s_g(\varphi(t)) = \frac{d}{d\varphi} s_g(\varphi) \omega(\varphi) = \beta_g u_g - \gamma_g s_g \\ E[s_{gc}] = s_g(\varphi_c) = \exp\left(\sum_f v_{gf} \tilde{\zeta}_f(\varphi_c)\right) dtdsg(φ(t))=dφdsg(φ)ω(φ)=βgug−γgsgE[sgc]=sg(φc)=expf∑vgfζ~f(φc)
其中我们假设 βg\beta_gβg 和 γg\gamma_gγg 在细胞周期内是常数。 -
典型的细胞周期基因表现出只能用少数谐波描述的特征,因此,在展开中,我们将考虑kkk个傅里叶分量(在实践中,默认使用一个谐波)。又由于s(φ)s(\varphi)s(φ)是正的,记:
log(sg(φc))=∑fvgfζ~f(φc)vg=(ag0ag1bg1⋯agkbgk)⊤ζ~(φ)=(1cos(φ)sin(φ)⋯cos(kφ)sin(kφ))⊤\log(s_g(\varphi_c)) = \sum_f v_{gf} \tilde{\zeta}_f(\varphi_c) \\ v_g = \begin{pmatrix} a_g^0 & a_g^1 & b_g^1& \cdots & a_g^k & b_g^k \end{pmatrix}^\top \\ \tilde{\zeta}(\varphi) = \begin{pmatrix} 1 & \cos(\varphi) & \sin(\varphi) & \cdots & \cos(k\varphi) & \sin(k\varphi) \end{pmatrix}^\top log(sg(φc))=f∑vgfζ~f(φc)vg=(ag0ag1bg1⋯agkbgk)⊤ζ~(φ)=(1cos(φ)sin(φ)⋯cos(kφ)sin(kφ))⊤
这里vgv_gvg是用实数表示的基因傅里叶参数的向量。使用链式法则后得到u(φ)u(\varphi)u(φ):
ddtsg(φ(t))=ω(φ)sg(φ)∑fvgfddφζ~f(φ)log(ug(φ))=−log(βg)+log(ω(φ)∑fvgf∂φζ~f(φ)+γg)+log(sg(φ))∀gE[ugc]=ug(φ)=sg(φ)βg(ω(φ)∑fvgf∂φζ~f(φ)+γg)\frac{d}{dt} s_g(\varphi(t)) = \omega(\varphi) s_g(\varphi) \sum_f v_{gf} \frac{d}{d \varphi} \tilde{\zeta}_f(\varphi) \\ \log(u_g(\varphi)) = -\log(\beta_g) + \log(\omega(\varphi) \sum_f v_{gf}\partial_\varphi \tilde{\zeta}_f(\varphi) + \gamma_g) + \log(s_g(\varphi)) \ \ \ \forall g \\ E[u_{gc}] = u_g(\varphi) = \frac{s_g(\varphi)}{\beta_g}(\omega(\varphi) \sum_f v_{gf} \partial_{\varphi}\tilde{\zeta}_f(\varphi) + \gamma_g) dtdsg(φ(t))=ω(φ)sg(φ)f∑vgfdφdζ~f(φ)log(ug(φ))=−log(βg)+log(ω(φ)f∑vgf∂φζ~f(φ)+γg)+log(sg(φ)) ∀gE[ugc]=ug(φ)=βgsg(φ)(ω(φ)f∑vgf∂φζ~f(φ)+γg)
似然
- 可以根据剪接 RNA (ScS_cSc) 和未剪接 RNA (UcU_cUc) 的计数数据,计算每个细胞的似然函数。
- 全联合似然函数 P({(Sc,Uc)}∣θ)P(\left\{(S_c, U_c)\right\} | \theta)P({(Sc,Uc)}∣θ) 由以下表达式组成:
P({(Sc,Uc)}∣θ)=∏gcP(Sgc,Ugc∣ω(φ),φc,vg,βg,γg,αg)P(Sgc,Ugc∣θ)=Ps(Sgc∣vg,αgs,φc)×Pu(Ugc∣ω(φ),βg,γg,vg,φc,αgu)Ps(Sgc∣…)=NB(sg(φc)=F[vg,φc],αgs)P(Ugc∣…)=NB(ug(φc)=G[ω(φc),βg,γg,vg,φc],αgu)P(\left\{(S_c, U_c)\right\} | \theta) = \prod_{gc} P(S_{gc}, U_{gc} | \omega(\varphi), \varphi_c, v_g, \beta_g, \gamma_g, \alpha_g) \\ P(S_{gc}, U_{gc} | \theta) = P_s(S_{gc} | v_g, \alpha_g^s, \varphi_c) \times P_u(U_{gc} | \omega(\varphi), \beta_g, \gamma_g, v_g, \varphi_c, \alpha_g^u) \\ P_s(S_{gc} | \ldots) = \text{NB}(s_g(\varphi_c)=F[v_g, \varphi_c], \alpha_g^s) \\ P(U_{gc} | \ldots) = \text{NB}(u_g(\varphi_c)=G[\omega(\varphi_c), \beta_g, \gamma_g, v_g, \varphi_c], \alpha_g^u) P({(Sc,Uc)}∣θ)=gc∏P(Sgc,Ugc∣ω(φ),φc,vg,βg,γg,αg)P(Sgc,Ugc∣θ)=Ps(Sgc∣vg,αgs,φc)×Pu(Ugc∣ω(φ),βg,γg,vg,φc,αgu)Ps(Sgc∣…)=NB(sg(φc)=F[vg,φc],αgs)P(Ugc∣…)=NB(ug(φc)=G[ω(φc),βg,γg,vg,φc],αgu)
其中θ\thetaθ表示参数,F,GF, GF,G表示sg,ugs_g, u_gsg,ug与其他量的依赖关系。
贝叶斯模型
- 通过结合生物学定义的先验(priors)和从数据中得出的经验贝叶斯先验,近似计算联合后验概率分布 P(θ∣Sc,Uc)P(\theta | S_c, U_c)P(θ∣Sc,Uc):
P(θ∣Sc,Uc)=P(Sc,Uc∣θ)P(θ)P(Sc,Uc)=∏gcP(Sgc∣θ)P(Ugc∣θ)P(θ)∫∏gcP(Sgc∣θ)P(Ugc∣θ)P(θ)dθP(\theta | S_c, U_c) = \frac{P(S_c, U_c | \theta) P(\theta)}{P(S_c, U_c)} = \frac{\prod_{gc} P(S_{gc} | \theta) P(U_{gc} | \theta) P(\theta)}{\int \prod_{gc} P(S_{gc} | \theta) P(U_{gc} | \theta) P(\theta) d\theta} P(θ∣Sc,Uc)=P(Sc,Uc)P(Sc,Uc∣θ)P(θ)=∫∏gcP(Sgc∣θ)P(Ugc∣θ)P(θ)dθ∏gcP(Sgc∣θ)P(Ugc∣θ)P(θ)
其中先验P(θ)P(\theta)P(θ)为:
vωt∼N([0,0,0],[32,0.052,0.052])log(γg)∼N(0,0.52)log(βg)∼N(2,32)αg∼Gamma(1.0,2.0)vgt∼N(μgtv,σgtv2)φxyc=ProjNormal(φxc,φyc)v\omega_t \sim \mathcal{N}([0, 0, 0], [3^2, 0.05^2, 0.05^2]) \\ \log(\gamma_g)\sim \mathcal{N}(0, 0.5^2) \\ \log(\beta_g) \sim \mathcal{N}(2, 3^2) \\ \alpha_g \sim \text{Gamma}(1.0, 2.0) \\ v_{gt} \sim \mathcal{N}(\mu_{gt}^v, {\sigma_{gt}^v}^{2}) \\ \varphi xy_c = \text{ProjNormal}(\varphi x_c, \varphi y_c) vωt∼N([0,0,0],[32,0.052,0.052])log(γg)∼N(0,0.52)log(βg)∼N(2,32)αg∼Gamma(1.0,2.0)vgt∼N(μgtv,σgtv2)φxyc=ProjNormal(φxc,φyc) - 通过经验贝叶斯(Empirical Bayes)设置以下参数:
μgtv=[log(meanc(Sgc)),0,0]σgtv=[12⋅stdc(Sgc+1)14⋅stdc(Sgc+1)14⋅stdc(Sgc+1)]φxc=ϵcos(Φc)φyc=ϵsin(Φc)\mu_{gt}^v = [\log(\text{mean}c(S{gc})), 0, 0] \\ \sigma_{gt}^v = \begin{bmatrix} \frac{1}{2} \cdot \text{std}_c(S{gc} + 1)\\ \ \frac{1}{4} \cdot \text{std}_c(S{gc} + 1)\\ \ \frac{1}{4} \cdot \text{std}_c(S{gc} + 1) \end{bmatrix}\\ \varphi x_{c} = \epsilon \cos(\Phi_c) \\ \varphi y_{c} = \epsilon \sin(\Phi_c) μgtv=[log(meanc(Sgc)),0,0]σgtv=21⋅stdc(Sgc+1) 41⋅stdc(Sgc+1) 41⋅stdc(Sgc+1)φxc=ϵcos(Φc)φyc=ϵsin(Φc)
其中Φc=tan−1(ω2c,ω1c)\Phi_c=\tan^{-1}(\omega_{2c}, \omega_{1c})Φc=tan−1(ω2c,ω1c)
变分分布(SVI)
- 变分分布 P({vωt,{φc},{vgt},{βg},{γg},{αg})P(\{v\omega_{ t}, \{\varphi_c\}, \{v_{gt}\}, \{\beta_g\}, \{\gamma_g\}, \{\alpha_g\})P({vωt,{φc},{vgt},{βg},{γg},{αg}) 被分解为多个独立分量的乘积,其形式为:
P({vωt,{φc},{vgt},{βg},{γg},{αg})=∏cP(vωt)P(φc)P(vgt)P(βg)P(γg)P(αg)P(\{v\omega_{ t}, \{\varphi_c\}, \{v_{gt}\}, \{\beta_g\}, \{\gamma_g\}, \{\alpha_g\}) = \prod_c P(v\omega_{ t}) P(\varphi_c)P(v_{gt}) P(\beta_g)P(\gamma_g) P(\alpha_g) P({vωt,{φc},{vgt},{βg},{γg},{αg})=c∏P(vωt)P(φc)P(vgt)P(βg)P(γg)P(αg) - 变分分布参数化如下:
P(vωt)∼N(μvωt^,σvωt^2)P(vgt)∼N(μvgv^,σvgv2^)P(αg)=Delta(αg^)P(log(γg))∼N(μlogγg^,σlogγg2^)P(log(βg))∼N(μlogβg^,σlogβg2^)P(φxyc)∼N([φxc^,φyc^],[1,1])P(v\omega _{t}) \sim \mathcal{N}(\widehat{\mu v\omega_t}, \widehat{\sigma v\omega_t}^2) \\ P(v_{gt}) \sim \mathcal{N}(\widehat{\mu_{v_g}^v}, \widehat{\sigma_{v_g}^v{ }^2}) \\ P(\alpha_g) = \text{Delta}(\widehat{\alpha_g}) \\ P(\log(\gamma_g)) \sim \mathcal{N}(\widehat{\mu_{\log \gamma_g}}, \widehat{\sigma_{\log \gamma_g}^2}) \\ P(\log(\beta_g)) \sim \mathcal{N}(\widehat{\mu_{\log \beta_g}}, \widehat{\sigma_{\log \beta_g}^2}) \\ P(\varphi xy_c) \sim \mathcal{N}([\widehat{\varphi x_c}, \widehat{\varphi y_c}], [1, 1]) P(vωt)∼N(μvωt,σvωt2)P(vgt)∼N(μvgv,σvgv2)P(αg)=Delta(αg)P(log(γg))∼N(μlogγg,σlogγg2)P(log(βg))∼N(μlogβg,σlogβg2)P(φxyc)∼N([φxc,φyc],[1,1])
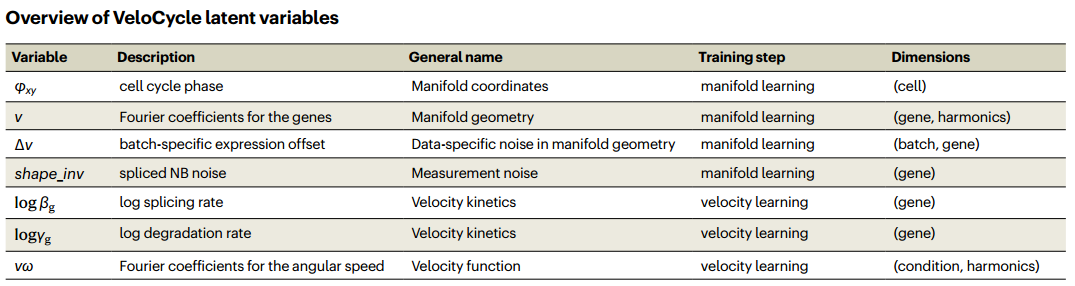
变分分布(LRMN)
- 低秩多变量正态(LRMN)模型考虑了观测数据之间的相关结构,基于变分推断(VI)构造的变分分布,观察到的联合后验由 MCMC 采样估计。具体而言,我们允许协方差和建立速度场 vωtv_{\omega t}vωt 以及动力学参数 βg\beta_gβg 和 γg\gamma_gγg 之间的关系。
- 后验因子分解如下:
P({νωt},{φc},{νgt},{βg},{γg},{αg})=P({γg},{νωt})∏gP(βg∣γg)P(αg)∏tP(νωt)P(νgt)∏cP(φc)\begin{align*} P\left(\{\nu \omega_t\}, \{\varphi_c\}, \{\nu_{gt}\}, \{\beta_g\}, \{\gamma_g\}, \{\alpha_g\} \right) &= P\left(\{\gamma_g\}, \{\nu \omega_t\}\right) \prod_g P(\beta_g \mid \gamma_g) P(\alpha_g) \prod_t P(\nu \omega_t) P(\nu_{gt}) \prod_c P(\varphi_c) \end{align*} P({νωt},{φc},{νgt},{βg},{γg},{αg})=P({γg},{νωt})g∏P(βg∣γg)P(αg)t∏P(νωt)P(νgt)c∏P(φc) - 具体公式是:
x≡[log(γ1),log(γ2),…,log(γng),νω0,νω1,…,νωnt−1]Σ=F^F^⊤+diag(d^)where F^∈R(ng+nt)×k,with k=5P({log(γg)},{νωt})=P(x)=MultivariateNormal(m^,Σ)μlogβg∣γ=μ^logβg+ρ^g⋅μ^logβg⋅log(γg)−μ^logγgσlogγgwith ρ^g∈[0,1]σlogβg∣γ=μ^logβg1−ρ^g2P(log(βg)∣log(γg))=N(μlogβg∣γ,σlogβg∣γ2)P(φc)=N([φ^xc,φ^yc],[1,1])P(νgt)=N(μ^gtν,σ^gtν2)P(αg)=Delta(αg^)\mathbf{x} \equiv \left[ \log(\gamma_1), \log(\gamma_2), \ldots, \log(\gamma_{n_g}), \nu \omega_0, \nu \omega_1, \ldots, \nu \omega_{n_t-1} \right] \\ \boldsymbol{\Sigma} = \hat{\mathbf{F}} \hat{\mathbf{F}}^\top + \mathrm{diag}(\hat{\mathbf{d}}) \quad \text{where } \hat{\mathbf{F}} \in \mathbb{R}^{(n_g+n_t) \times k}, \text{ with } k = 5 \\ P(\{\log(\gamma_g)\}, \{\nu \omega_t\}) = P(\mathbf{x}) = \text{MultivariateNormal}(\hat{\mathbf{m}}, \boldsymbol{\Sigma}) \\ \mu_{\log \beta_g | \gamma} = \hat{\mu}_{\log \beta_g} + \hat{\rho}_g \cdot \hat{\mu}_{\log \beta_g} \cdot \frac{ \log(\gamma_g) - \hat{\mu}_{\log \gamma_g} }{ \sigma_{\log \gamma_g} } \quad \text{with } \hat{\rho}_g \in [0,1] \\ \sigma_{\log \beta_g | \gamma} = \widehat{\mu}_{\log \beta_g} \sqrt{1 - \widehat{\rho}_g^2} \\ P(\log(\beta_g) \mid \log(\gamma_g)) = \mathcal{N}(\mu_{\log \beta_g | \gamma}, \sigma_{\log \beta_g | \gamma}^2) \\ P(\varphi_c) = \mathcal{N}([\widehat{\varphi} \mathbf{x}_c, \widehat{\varphi} y_c], [1,1]) \\ P(\nu_{gt}) = \mathcal{N}(\widehat{\mu}_{gt}^{\nu}, \widehat{\sigma}_{gt}^{\nu 2}) \\ P(\alpha_g) = \text{Delta}(\widehat{\alpha_g}) x≡[log(γ1),log(γ2),…,log(γng),νω0,νω1,…,νωnt−1]Σ=F^F^⊤+diag(d^)where F^∈R(ng+nt)×k, with k=5P({log(γg)},{νωt})=P(x)=MultivariateNormal(m^,Σ)μlogβg∣γ=μ^logβg+ρ^g⋅μ^logβg⋅σlogγglog(γg)−μ^logγgwith ρ^g∈[0,1]σlogβg∣γ=μlogβg1−ρg2P(log(βg)∣log(γg))=N(μlogβg∣γ,σlogβg∣γ2)P(φc)=N([φxc,φyc],[1,1])P(νgt)=N(μgtν,σgtν2)P(αg)=Delta(αg)
模型实现
- 模型实现旨在估算联合后验概率分布的近似值,涉及角细胞周期速度 (vωtv\omega_tvωt),和 S1S^1S1 流形上的参数()。该实现分两个步骤进行:流形学习和速度学习。
- 在流形学习中,我们估计每个细胞沿细胞周期流形 (ϕ\phiϕ) 的位置,以及每个基因的傅里叶级数(vvv)。
- 所有变量初始化为先验的均值。先验均值通过以下两种方式确定:
-
- 使用数据的前两个主成分 (ϕ\phiϕ),这是一种降维方法,提取数据的低维结构。
-
- 使用每个基因剪接表达量 (vvv) 的均值和标准差 (s.d.s.d.s.d.),以反映基因表达的统计特性。
剪接计数 (ElogS) 的期望值从真实数据和负二项分布 (NB) 建模得出,允许捕捉表达数据的离散性和过分散性。
- 使用每个基因剪接表达量 (vvv) 的均值和标准差 (s.d.s.d.s.d.),以反映基因表达的统计特性。
- 为适应不同数据集或批次间平均表达水平的差异,模型引入了第一个基因谐波系数的偏移项 (Δv\Delta vΔv)。
- 速度学习的目标是基于流形学习的结果,估算傅里叶系数、角速度 (vωv_\omegavω) 以及速度动力学参数 (γ\gammaγ 和 β\betaβ)。
- 所有变量初始化为先验的均值。特别地:
-
- 角速度 (vωv\omegavω) 的先验均值假设为零,反映了对零细胞周期速度的假设。
-
- 其他变量(如傅里叶系数和动力学参数)也初始化为先验均值,具体取决于流形学习阶段的估计结果。
-
- 为了确保模型输出满足生物学意义上的正值约束,特别是在方程 (10) 中 (ω(ϕ)∑fvgf∂ϕζf(ϕ)+γg\omega(\phi) \sum_f v_{gf} \partial_\phi\zeta_f(\phi) + \gamma_gω(ϕ)∑fvgf∂ϕζf(ϕ)+γg),学习过程中引入了 ReLU 函数。
- 我们使用SVI求解VeloCycle模型,并应用ClippedAdam优化器和ELBO损失函数,从第一次到最后一次训练迭代,学习率从0.03衰减到0.005。
- 提供了提前终止选项:如果前 100 次迭代的均值损失与前 10 次迭代的均值损失相差小于五个单位,则停止训练。
- 速度动力学参数 γ\gammaγ 和 β\betaβ 受到生物学约束的限制:
-
- γg\gamma_gγg 的范围为 [0.5, 1.5] h−1^{-1}−1,表示基因特定的动力学速率。
-
- 周期 T=2π/ω0T = 2\pi/\omega_0T=2π/ω0 的范围为 [6, 50] h,反映细胞周期的生物学合理时间范围。
- 速度谐波系数的先验均值设为0,标准差为 3.0,反映了对无初始速度的假设,同时允许较大的变异性以适应数据变化。所有先验可以通过 ‘velocycle.preprocessing’ 套件中的函数修改,并通过 Pyro 模型对象的元参数 (‘mp’) 项集成。
- 执行MCMC时,使用No-U-Turn(NUTS)核,从SVI首先获得的平均后验估计开始。
估算恒定细胞周期速度的近似点
- 模型通过求解一阶微分方程 ddtsg(t)=βgug−γgsg\frac{d}{dt}s_g(t) = \beta_g u_g - \gamma_g s_gdtdsg(t)=βgug−γgsg 来获得初始洞察,其中 γg\gamma_gγg 是基因依赖的降解率,βg\beta_gβg 和 ugu_gug 分别是与基因相关的参数。
- 假设未剪接读数 ug(t)u_g(t)ug(t) 遵循单谐波周期函数,即 ug(t)=u0g(1+ϵcos(ωt−φ0g))u_g(t) = u_{0g} (1 + \epsilon \cos(\omega t - \varphi_{0g}))ug(t)=u0g(1+ϵcos(ωt−φ0g)),其中 ω\omegaω 表示细胞周期速度,φ0g\varphi_{0g}φ0g 是相位偏移,ϵ\epsilonϵ 是幅度。
- 基于上述假设,剪接读数 sg(t)s_g(t)sg(t) 具有相同的函数形式,但幅度和相位经过调整,即 sg(t)=s0g(1+ϵ′cos(ωt−φig))s_g(t) = s_{0g} (1 + \epsilon' \cos(\omega t - \varphi_{ig}))sg(t)=s0g(1+ϵ′cos(ωt−φig))。其中,ϵ′=ϵcos(Δφg)\epsilon' = \epsilon \cos(\Delta \varphi_g)ϵ′=ϵcos(Δφg),Δφg=(φg−φ0g)\Delta \varphi_g = (\varphi_g - \varphi_{0g})Δφg=(φg−φ0g),且 tan(Δφg)=ωγg−1\tan(\Delta \varphi_g) = \omega \gamma_g^{-1}tan(Δφg)=ωγg−1。这表明相位差和幅度调整与细胞周期速度 ω\omegaω 和降解率 γg\gamma_gγg 相关。
- 假设存在多个条件(或重复实验),且寿命 τg=γg−1\tau_g = \gamma_g^{-1}τg=γg−1 与条件无关,观察到关系 δcg=tan(Δφcg)=ωcτg\delta_{cg} = \tan(\Delta \varphi_{cg}) = \omega_c \tau_gδcg=tan(Δφcg)=ωcτg。这表示相切值 δcg\delta_{cg}δcg 可看作细胞周期速度 ωc\omega_cωc 与寿命 τg\tau_gτg 的乘积。
- 通过奇异值分解 (SVD),δcg\delta_{cg}δcg 可以分解为秩-1 矩阵形式,即 δcg=ucdvg+\delta_{cg} = u_c d v_g +δcg=ucdvg+ 更高秩项,其中 ucu_cuc 和 vgv_gvg 分别是条件和基因的向量,ddd 是标量。
- 基于 SVD,结果可进一步表达为条件特定的细胞周期速度 ωc\omega_cωc,以逆平均半衰期单位(记为 ωc∗\omega_c^*ωc∗)表示,即 ωc∗=ucdvg\omega_c^* = u_c d v_gωc∗=ucdvg。其中 vgv_gvg 是基因的平均值。
- 周期长度以平均半衰期单位表示为 Tc∗=2πωc∗T_c^* = \frac{2\pi}{\omega_c^*}Tc∗=ωc∗2π,反映了细胞周期的周期性特性。
数据集
- ‘small’:包含 97 个基因。
- ‘medium’:包含 218 个基因。默认使用。
- ‘large’:包含 1,918 个基因。
- 使用 velocycle.utils.get_cycling_gene_set 函数访问这些人类和老鼠的基因集。
