RNA甲基化技术如何选择?
在表观遗传学研究中,DNA甲基化和RNA甲基化是两类关键的修饰类型,它们在基因表达调控、细胞分化、疾病发生等过程中发挥重要作用。然而,由于DNA和RNA的化学性质差异显著,且甲基化位点分布、功能机制各不相同,研究者需要根据具体的研究背景(如样本类型、目标修饰类型、经费预算、技术可行性等)选择合适的技术方案。上期我们已经介绍了DNA甲基化研究技术如何选择,本期将聚焦RNA甲基化技术。
RNA甲基化以N6-甲基腺苷(m⁶A)为主(占真核生物mRNA甲基化的80%以上),此外还有m⁵C、m¹A等修饰类型。m⁶A主要分布在mRNA的编码区外显子、3'非翻译区(3'UTR)及长链非编码RNA(lncRNA)中,参与调控mRNA的剪接、出核、翻译效率及稳定性(如METTL3/METTL14复合物负责甲基化写入,FTO/ALKBH5负责擦除,YTHDF1/2/3等蛋白介导识别)。根据研究目标,RNA甲基化技术同样可分为三类。
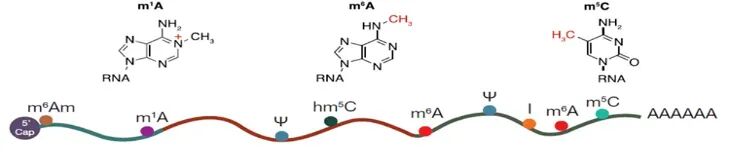
RNA甲基化类型
第一类
整体定量
适用于快速评估样本整体甲基化状态,但无法提供位点特异性信息。
1. RNA甲基化Dot Blot
原理:将变性后的总RNA直接点在尼龙膜上,经UV交联固定后,用抗m⁶A(或m⁵C等)抗体孵育,化学发光显色,信号强度即代表甲基化水平。
分辨率:只能给出整体甲基化水平,无法定位具体位点。
适用场景:快速比较不同样本/处理组的全局m⁶A变化;高通量测序(MeRIP-seq等)前的预实验;低预算。
优点:操作简单、成本低;可平行检测多份样本;无需复杂仪器。
缺点:半定量;抗体交叉反应可致假阳性;无法提供序列定位信息。
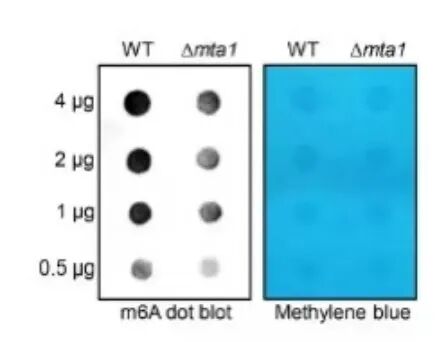
Dot Blot结果
2. RNA LC-MS/MS(液相色谱-串联质谱)
原理:RNA酶解成单核苷→LC分离→串联质谱定量m⁶A、Am、m⁵C等修饰核苷与对应未修饰核苷之比。
分辨率:单碱基水平(可区分修饰类型),但无序列位置信息。
适用场景:绝对定量样本整体m⁶A、m⁵C等含量;验证高通量测序结果。
优点:灵敏度高、线性范围宽、重现性好;一次上机可同时检测多种修饰。
缺点:需昂贵LC-MS/MS系统与标准品;无法知道修饰发生在哪些转录本或位点。
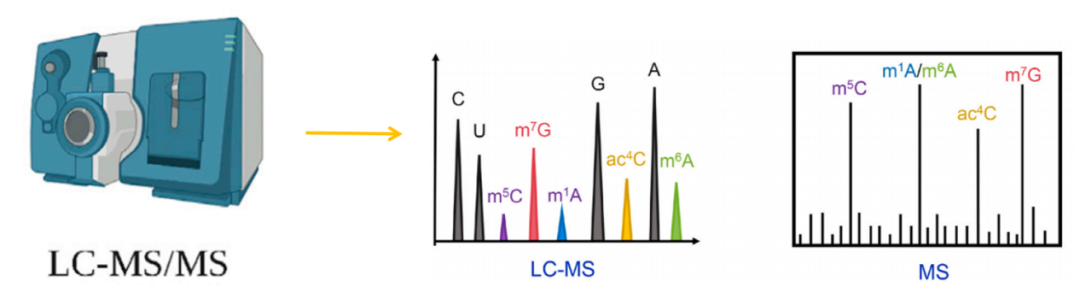
LC-MS/MS原理
第二类
位点鉴定
适用于全面解析甲基化图谱,包括机制探索、生物标志物发现等。
3. MeRIP-seq(m6A-IP-seq)
原理:将总RNA随机打断→用抗m⁶A抗体免疫沉淀富集甲基化片段→建库测序→与input对照比对,鉴定全转录组m⁶A位点与丰度。
分辨率:~100–200nt区域水平(取决于片段长度)。
适用场景:绘制细胞/组织全转录组m⁶A图谱;比较不同处理/疾病模型的m⁶A差异;与RNA-seq联用,研究甲基化对表达、剪接、翻译的影响。
优点:覆盖全转录组,灵敏度高;技术成熟,生信流程标准化;可与转录组等多组学整合。
缺点:分辨率仍在百碱基级,无法精确定位到单碱基;抗体批次差异和富集偏好可能导致假阳性;需设置input对照以校正表达量差异。
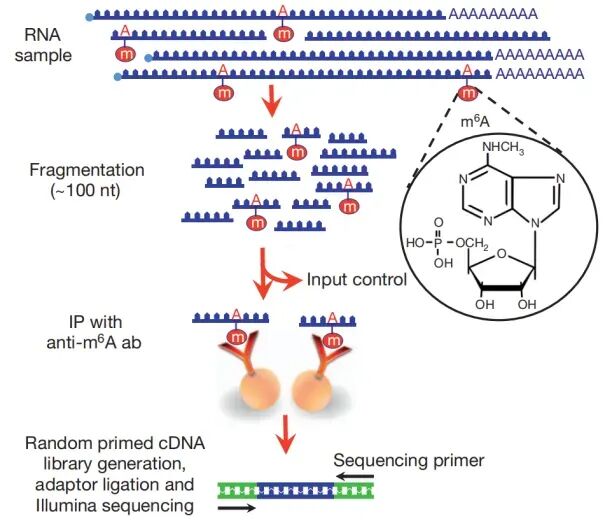
MeRIP-seq流程
4. miCLIP-seq(m6A-CLIP-seq)
原理:活细胞交联(UV)将m⁶A抗体与RNA共价固定→免疫沉淀→逆转录时交联位点产生特异突变/截短→高通量测序,通过突变信号精确定位m⁶A位点。
分辨率:单核苷酸。
适用场景:单碱基级m⁶A图谱、甲基化转移酶/去甲基酶靶点鉴定。
优点:目前唯一抗体依赖、单碱基分辨率的m⁶A检测技术;可同时获得m⁶Am信息;可与其他组学数据联合分析。
缺点:步骤繁琐(交联、胶回收、突变分析),实验周期长;抗体批次差异和交联效率影响重复性;对生信分析要求较高,需专门突变调用流程。
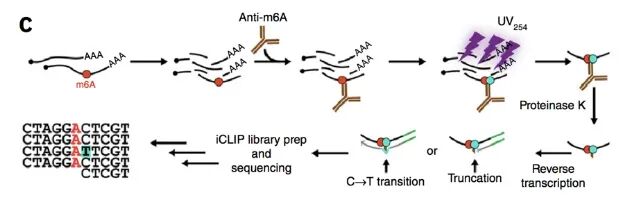
MiCLIP-seq流程
5. Nanoporedirect RNA-seq(DRS)
原理:RNA不经逆转录和PCR,直接由马达酶牵引穿过纳米孔;通过实时电流信号差异一次获得序列、m⁶A、m⁵C等修饰及poly(A)尾长度。
分辨率:接近单碱基,可定位到修饰位点;读长即全长转录本,无长度限制。
适用场景:全长转录本+修饰联合分析。
优点:保留天然RNA特征,无逆转录/扩增偏差;一次实验可同时检测多种修饰并估算poly(A)长度;可发现新型异构体与融合转录本。
缺点:单次错误率约5–10%,需高覆盖矫正;建库成本仍高于二代;修饰识别依赖训练模型,跨物种需重新训练。
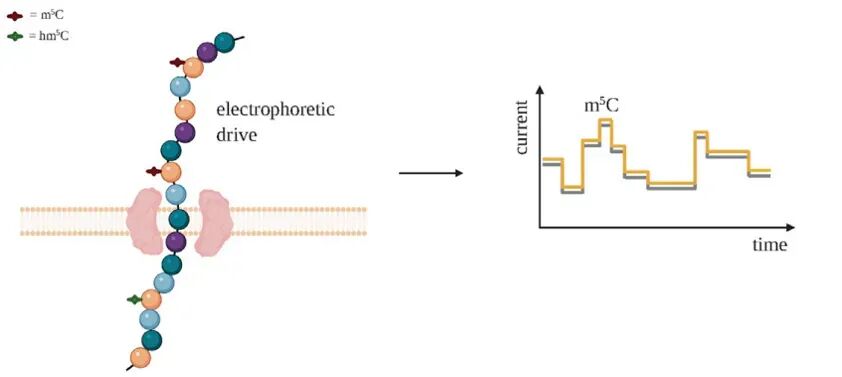
DRS原理
6. RNA亚硫酸氢盐测序(RNA-Bisseq)
原理:先用重亚硫酸氢盐处理RNA,未甲基化的胞嘧啶(C)脱氨成尿嘧啶(U),而5-甲基胞嘧啶(m⁵C)保持不变;随后逆转录、PCR扩增,通过比对C→T转化情况即可在单碱基水平定位并定量m⁵C位点。
分辨率:单核苷酸。
适用场景:特异性检测m⁵C修饰,适用于研究m⁵C的分布和功能。
优势:可精准定位m⁵C修饰位点;无需抗体,避免富集偏好。
局限:仅适用于m⁵C修饰,对样本质量要求较高;RNA二级结构区域转化不完全,可能产生假阳性;亚硫酸氢盐处理易导致RNA降解,需优化反应条件。
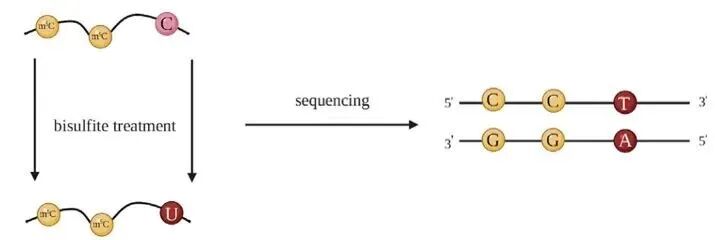
RNA-Bisseq原理
第三类
甲基化位点验证
适用于特定基因或区域的甲基化验证。
7. MeRIP/MiCLIP-qPCR
原理:先用MeRIP(或MeCLIP)免疫沉淀富集带m⁶A修饰的RNA片段(IP),同时留Input对照;随后对目标区域做qPCR,以%IP/Input或Fold enrichment值量化该区域的m⁶A修饰水平。
分辨率:MeRIP-qPCR为100–200nt区段;MiCLIP-qPCR可达单碱基。
适用场景:高通量测序(MeRIP-seq/MiCLIP-seq)后,对候选基因/位点进行验证;小样本快速比较不同处理组的m⁶A修饰差异。
优点:实验周期短、成本低;定量准确,与测序结果互补。
缺点:一次只能检测少数已知区域;需要优化抗体富集效率和qPCR引物特异性;IgG或突变信号需严格对照,避免非特异背景。
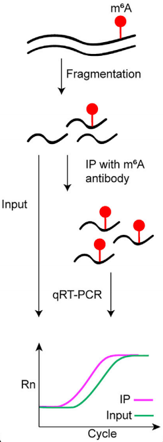
MeRIP-qPCR流程
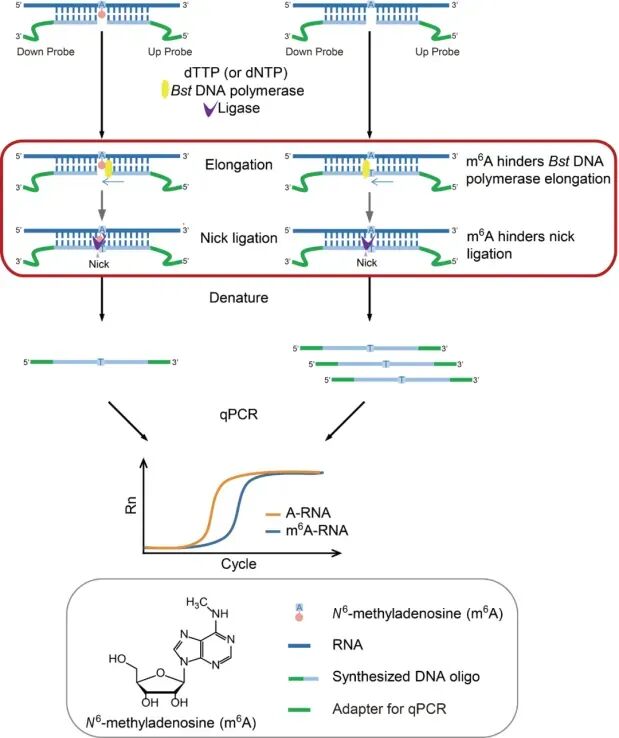
8. SELECT(Site-Specific Cleavage and Ligation-Enhanced qPCR)
原理:该技术基于引物延伸阻断与连接反应,能在单碱基分辨率下特异检测目标RNA位点的甲基化修饰,并结合qPCR定量分析。该方法灵敏度高,适合m⁶A等修饰的位点精确定量验证。
分辨率:单核苷酸,可精确定位并定量一个特定位点的m⁶A水平。
适用场景:高通量测序后候选位点的快速验证;低丰度RNA(tRNA、snRNA)或小样本的绝对定量;需要精确比例(%m⁶A)而非富集峰的研究。
优点:单碱基精度+绝对定量;对低丰度RNA仍灵敏;无需抗体,避免批次差异。
缺点:一次只能检测1个位点,无法高通量;Bst延伸与SplintR连接效率受探针设计影响,需严格阴性/阳性对照;若位点两侧二级结构复杂,易出现假阳性/阴性。
SELECT原理
如何选择?
若只是初步验证甲基化修饰水平是否有变化,可考虑Dot Blot;
若对甲基化修饰水平的绝对定量要求高,可考虑LC-MS/MS;
若需全转录组筛查甲基化修饰位点,优先选择MeRIP-seq;
若追求单碱基分辨率,优先选择MiCLIP-seq;
若仅需验证特定位点的甲基化修饰,MeRIP-qPCR/SELECT是较好的选择;
若研究m5C修饰,RNA-BisSeq是首选方法。
在实际应用中,您可根据研究需求结合多种技术,如先通过Dot Blot进行初步验证,再利用MeRIP-seq筛选目标基因,最后再用MeRIP-qPCR/SELECT进行验证。
爱基百客作为国内领先的表观服务商,配备了完善的表观组学实验平台和高通量测序平台。我们可以提供从实验设计-样本处理-数据分析一站式服务,为您的科研工作提供全方位支持。如果您在表观遗传学研究领域有任何技术需求,欢迎随时联系我们,我们期待为您的科研工作助力!
