解析蛋白质三维结构-Bio3D R包
最近休息时阅读了一本书:

在书本第5章结构信息学章节的末尾,看到了一个练习题,张贴如下:

这里作者提到了一个R包,
看着挺有意思的,所以就决定小学一下,毕竟这年头搞分子动力学起码是python重火力起步,看到R也能搞的属少数。
官网参考:http://thegrantlab.org/bio3d/

此处贴一个基本的介绍:
Bio3D是一个用于结构生物信息学分析的R语言包,主要用于生物大分子(如蛋白质和核酸)三维结构数据的处理和分析。它能够执行各种结构分析任务,如结构对接、同源建模、分子动力学模拟轨迹分析、结构比对等。
Bio3D的核心功能包括从PDB数据库中提取结构数据、计算结构相似性、生成结构比对和聚类分析,以及分析蛋白质动力学特征,如主成分分析(PCA)和正规模式分析(NMA)。
该包广泛应用于蛋白质功能研究、分子演化研究以及药物设计等领域,帮助研究者深入理解生物分子结构与功能的关系。
比如说我用python工具常做的分子动力学分析模块——
DCCM(Dynamic cross-correlation matrices, DCCM,动态相关性矩阵),表示蛋白质中每个氨基酸的特定原子,比如说Cα原子和其他氨基酸的Cα原子之间的相关性,提供蛋白质在大尺度范围内相关运动的一些信息。DCCM计算数值的取值范围从完全负相关的-1.0到完全正相关的+1.0。越接近数值1表示相关性越强,正负表示两个原子运动方向相同(反)。
计算时需要两个文件.dcd和.pdb,
前者是蛋白质的轨迹文件,可以使用VMD进行保存,一般是分子动力学模拟之后的后续分析。我一般用它只分析分子动力学模拟稳定后的轨迹,不一定是全部轨迹;
后者是轨迹配套的蛋白质文件,选定帧数之后单独保存为pdb格式即为蛋白质文件。
这个也能够使用这个R包分析。
1,6QNX分析示例
参考https://www.rcsb.org/structure/6QNX
是一个SA2/SCC1/CTCF complex复合物,X射线晶体衍射获取的结构数据。
一共是3个亚基:
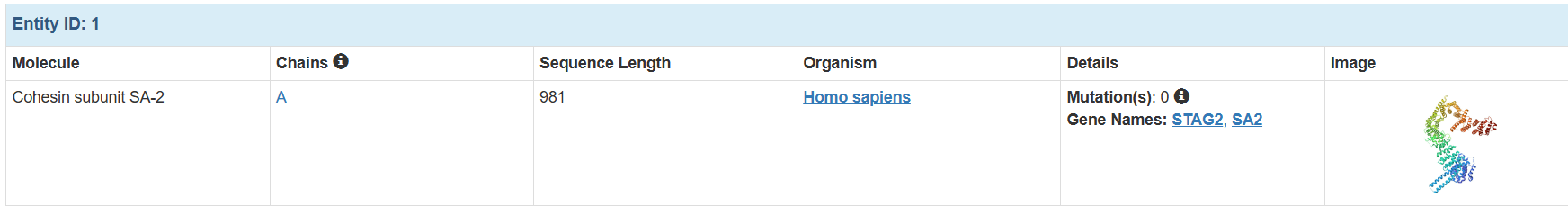
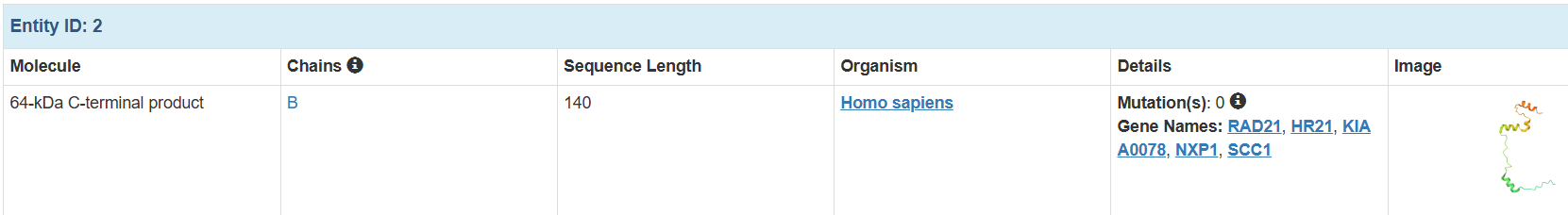
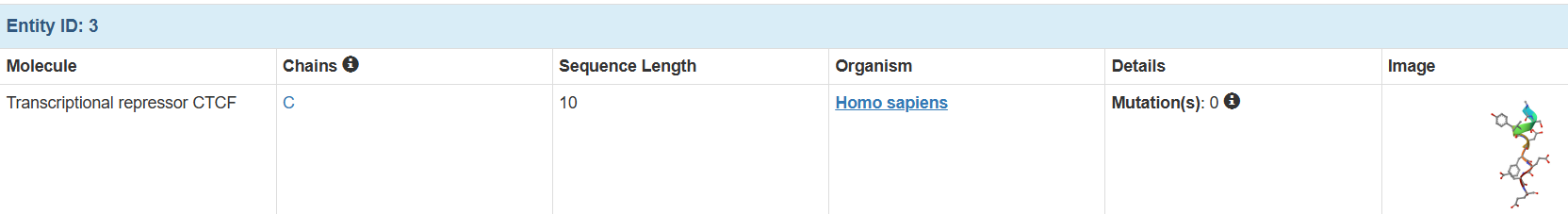
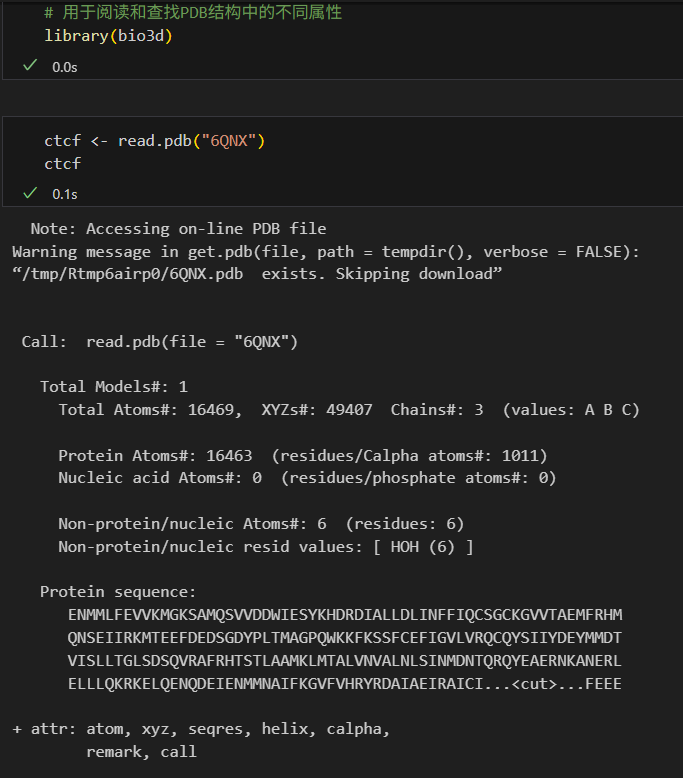
可以知道这个蛋白复合物,是由1011个氨基酸残基,16469个原子组成的三链复合物。
如果想要详细查看原子构成以及坐标信息,
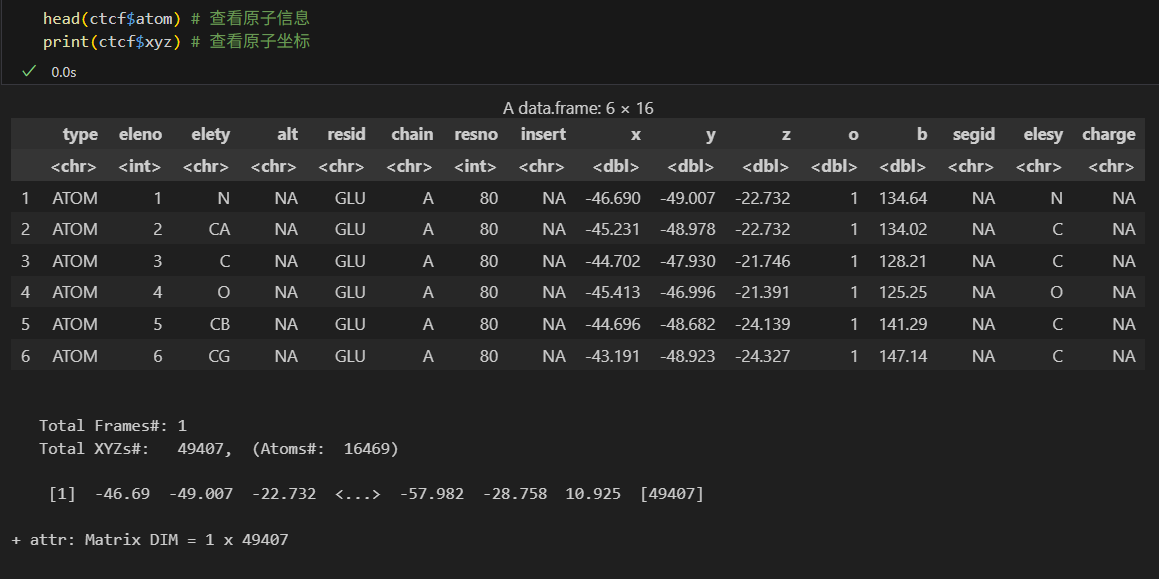
这些数据基本上都可以由mmcif文件中的坐标辅助佐证:
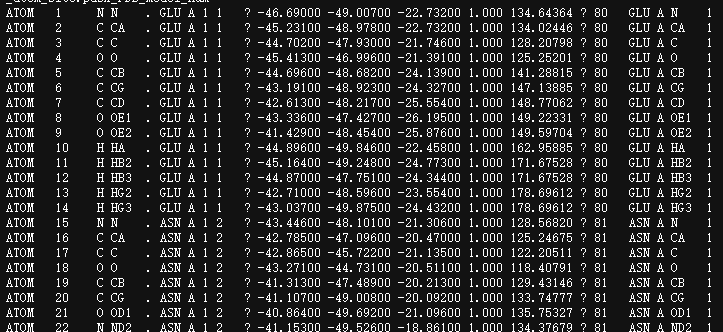
三维结构分析方面,因为api函数模块很多,都值得仔细研究以及阅读底层源码,
这里只介绍nma()函数,也就是正规模分析(NMA,Normal Mode Analysis),从而预测示例蛋白质的柔性。
参考:http://thegrantlab.org/bio3d/reference/nma.html

详细分析,有一个demo,可以参考:http://thegrantlab.org/bio3d_v2/tutorials/normal-mode-analysis
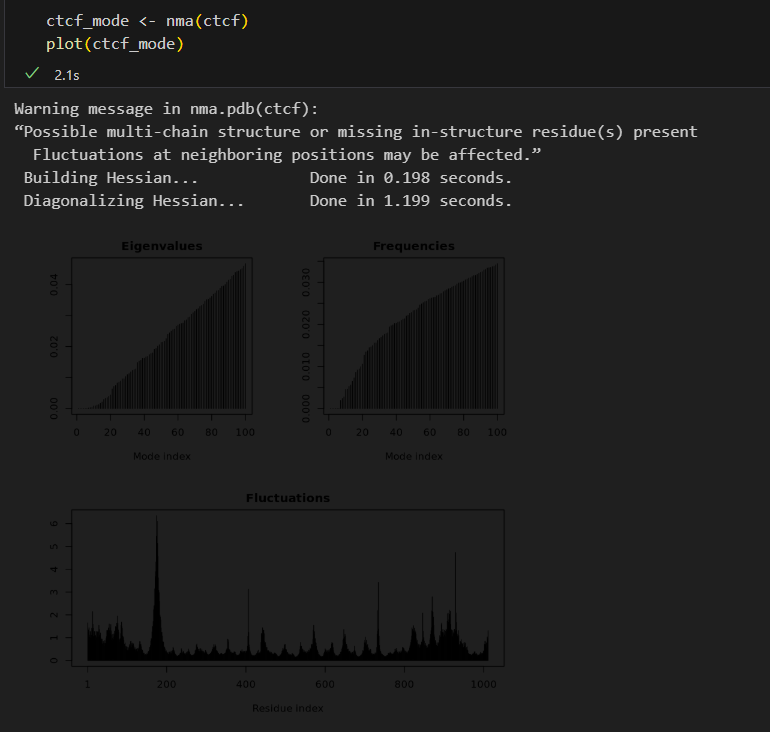
由图可知,上方两个图分别显示了特征值(Eigenvalues)和频率(Frequencies)随模式指数(Mode Index)的变化。特征值从左至右递增,说明高模式对应的刚性较大,运动较难发生。频率图与特征值密切相关,也表现出递增趋势,高频模式对应局部振动,低频模式则与全局运动相关。下方的波动图(Fluctuations)展示了蛋白质各残基的振幅波动情况。某些残基区域(如第185、400和730附近)显示出较大的波动,意味着这些区域更具柔性,可能在蛋白质功能中起重要作用。
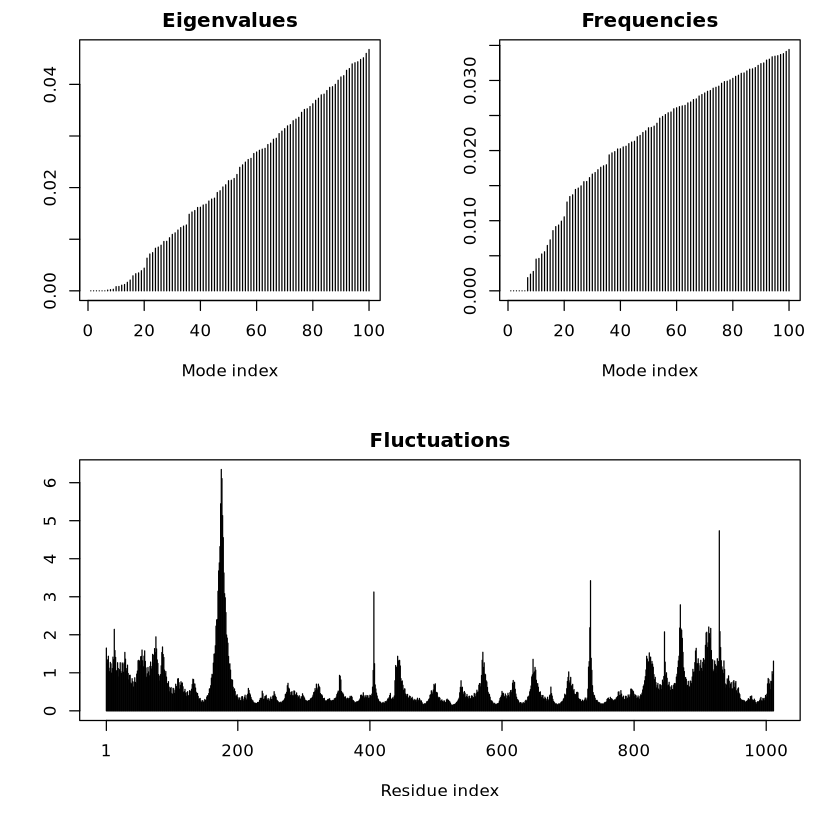
为了能够清晰的展示蛋白质的柔性和其二级结构之间的关系,我们可以使用plot.bio3d()函数中的参数在波动图(Fluctuations)上添加二级结构信息,
plot.bio3d(ctcf_mode$fluctuations, # NMA中每个残基的波动数据 sse=ctcf, # 提供PDB对象以获取蛋白质的二级结构信息sheet.col="orange", # 将β-折叠的颜色设置为橙色,用于在图中区分二级结构helix.col="purple", # 将α-螺旋的颜色设置为紫色,用于区分二级结构typ="l", # 设置图形类型为线条图(line plot),即用线连接数据点lwd=3, # 将线条宽度设为3,使线条更加粗壮,图形更清晰ylab="Fluctuations from NMA of CTCF-complex" # 设置y轴标签为自定义内容
)

由图可知,x轴表示残基编号,y轴表示波动幅度。图中的黑色曲线显示了各个残基的波动情况,波峰代表特定残基有较大的波动,表明这些区域在蛋白质运动中柔性更高。紫色的横条代表α螺旋区域,橙色横条代表β折叠区域(当然图中没有,code中示意),这些区域的波动通常较小,表明它们结构较为稳定,柔性相对较低。图像分析有助于研究蛋白质的动态特性,分析哪些区域更具柔性或稳定性。
2,2DFD分析示例
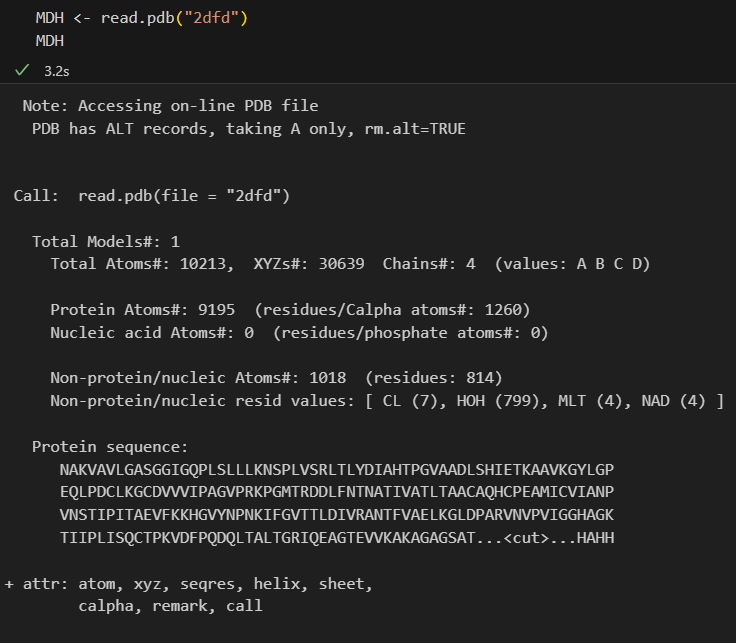
以苹果酸脱氢酶为例,苹果酸脱氢酶是一种在柠檬酸循环中催化草乙酸盐和苹果酸盐转化的同型二聚体酶,结构序列号为2DFD。

找出该结构中存在的链、原子和残基的总数。计算结合位点并绘制酶中残基的B因子:
这里解释一下什么是B-factor:
蛋白质结构的b因子也称为温度结构,它表示蛋白质由于C α原子在其平均位置附近波动而产生的灵活性。由于这种灵活性,蛋白质分子的多肽骨架和侧链会不断运动。高b因子表明蛋白质分子中的残基位置比平均值具有更高的柔韧性,而低b因子则反映了蛋白质分子中的刚性位置。隐藏在蛋白质分子核心的残基可能具有较低的b值,因此与存在于蛋白质表面的残基相比更具有刚性。
可以简单将B-factor理解为柔韧性、刚性描述符。

plot.bio3d(MDH$atom$b[MDH$calpha], sse=MDH, typ="l", ylab="Bfactor")
带有二级结构注释


苹果酸脱氢酶和乳酸脱氢酶是同源代谢酶。
序列号:人苹果酸脱氢酶(ID: 2DFD)和人乳酸脱氢酶(ID: 5ZJE)
(a)将存在于两种脱氢酶结构中的链进行拆分和对齐,并计算蛋白质结构不同链之间的序列同一性和均方根偏差(RMSD)
(b)绘制RMSD直方图并生成RMSD聚类的树状图
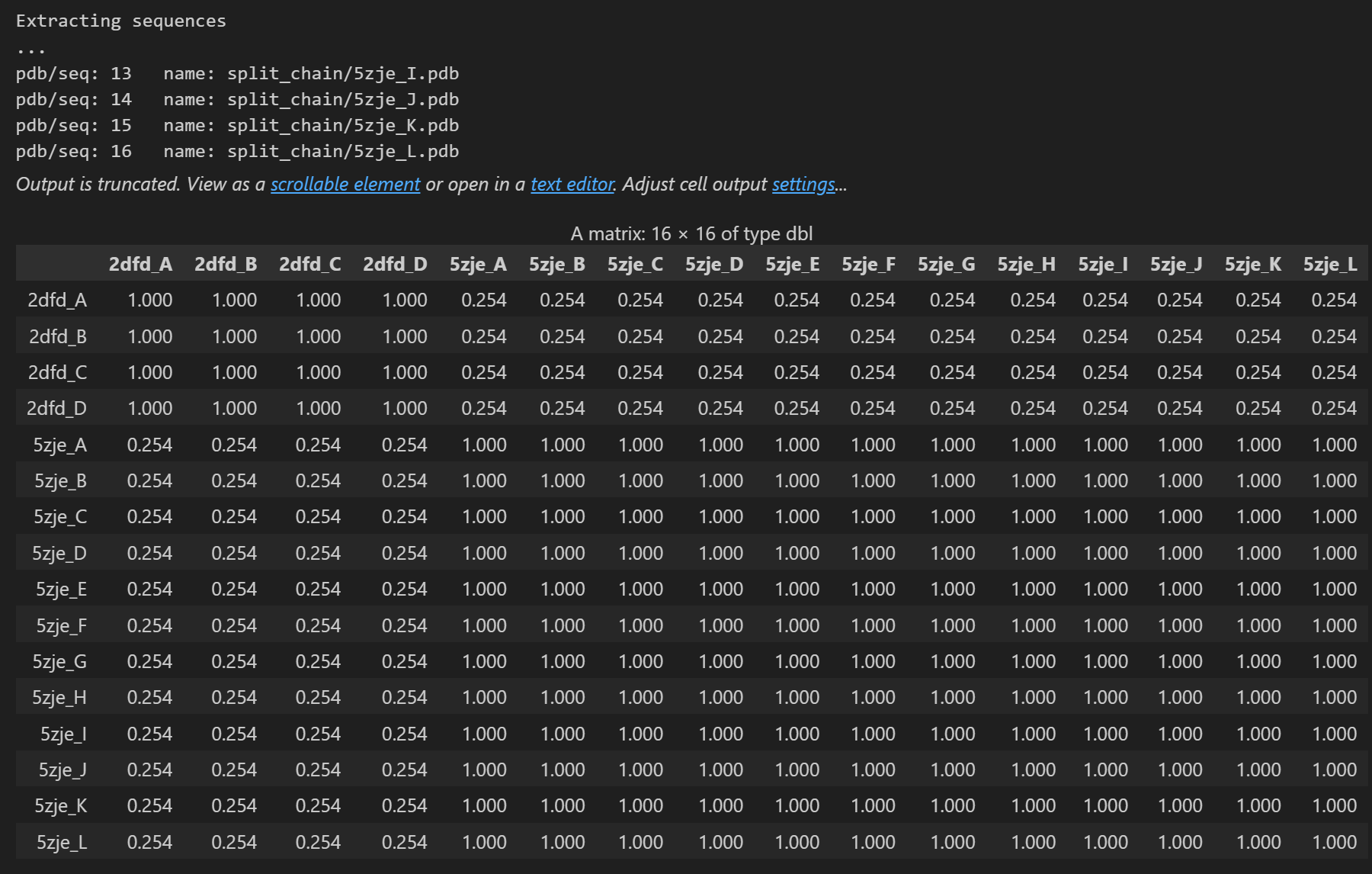
ids <- c("2dfd","5zje")
raw.files <- get.pdb(ids)
files <- pdbsplit(raw.files, ids)
pdbs <- pdbaln(files,exefile="msa")
pdbs$id <- substr(basename(pdbs$id),1,6)
seqidentity(pdbs)
core <- core.find(pdbs)
core.inds <- print(core, vol=1.0)
xyz <- pdbfit( pdbs, core.inds )
rd <- rmsd(xyz)
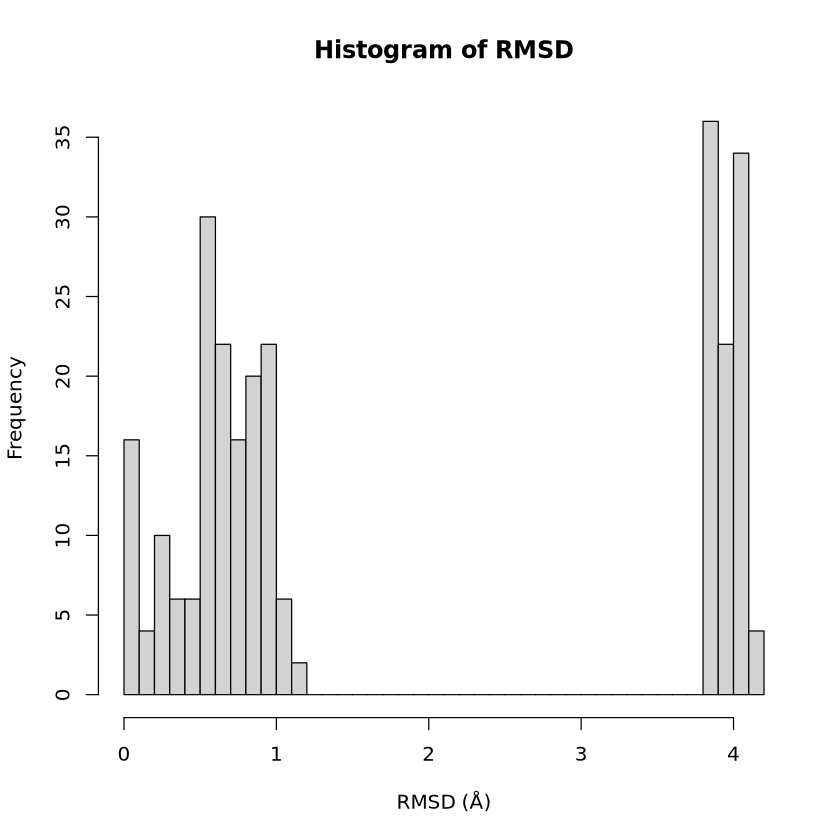
hist(rd, breaks=40, xlab="RMSD (Å)", main="Histogram of RMSD")
两个酶不同链间RMSD的直方图:
hc <- hclust(as.dist(rd))
hclustplot(hc, k=3, ylab="RMSD (Å)", main="RMSD Cluster Dendrogram")
基于苹果酸脱氢酶和乳酸脱氢酶不同链间RMSD的RMSD聚类图: