wgd v1.1.2 安装与使用-生信工具056
wgd - 简单命令行工具用于分析古代全基因组复制(WGD)
假如你对 wgd 中实现的方法感兴趣,可能还想考虑使用 Sensalari 等人的 ksrates 工具。该工具可用于仔细比较多个 Ks 分布并对其进行建模(ksrates 内部使用 wgd)。
官网
https://github.com/arzwa/wgd #github安装
wgd 是一个用于分析全基因组复制(WGD)的 Python 包和命令行界面(CLI)。已在 Linux 系统上使用 Python3 测试。如果你没有安装 Python 或 pip,可以通过运行以下命令来安装:
sudo apt-get install python3-pip
安装步骤:
克隆 GitHub 仓库并安装:
git clone https://github.com/arzwa/wgd.git cd wgd pip install --user .注意:根据你的 Python 安装和虚拟环境的不同,
pip可能默认使用pip2或pip3。如果安装失败,请尝试使用pip3代替pip。
使用命令行界面(CLI)
安装完成后,运行以下命令查看可用命令列表:
$ wgd
若要查看某个命令(例如 ksd)的使用说明:
$ wgd ksd --help
外部软件要求:
conda create -n wgd python=3.5 blast mcl muscle mafft prank paml fasttree cmake libpng mpi=1.0=mpich
conda activate wgd对于i-ADHoRe,需要先在http://bioinformatics.psb.ugent.be/webtools/i-adhore/licensing/同意许可,才能下载i-ADHoRe-3.0
代码能力差,就会不是很好用注意事项
如果遇到问题,请确保你安装了最新版本的 PAML。为了安装最新版本,最好不要依赖 apt-get 或其他包管理器,而是从源码安装。以下是安装步骤:
wget http://abacus.gene.ucl.ac.uk/software/paml4.9j.tgz
tar -xzf paml4.9j.tgz
pushd paml4.9j/src && make -f Makefile && popd
export PATH=$PATH:$PWD/paml4.9j/src/
快速开始
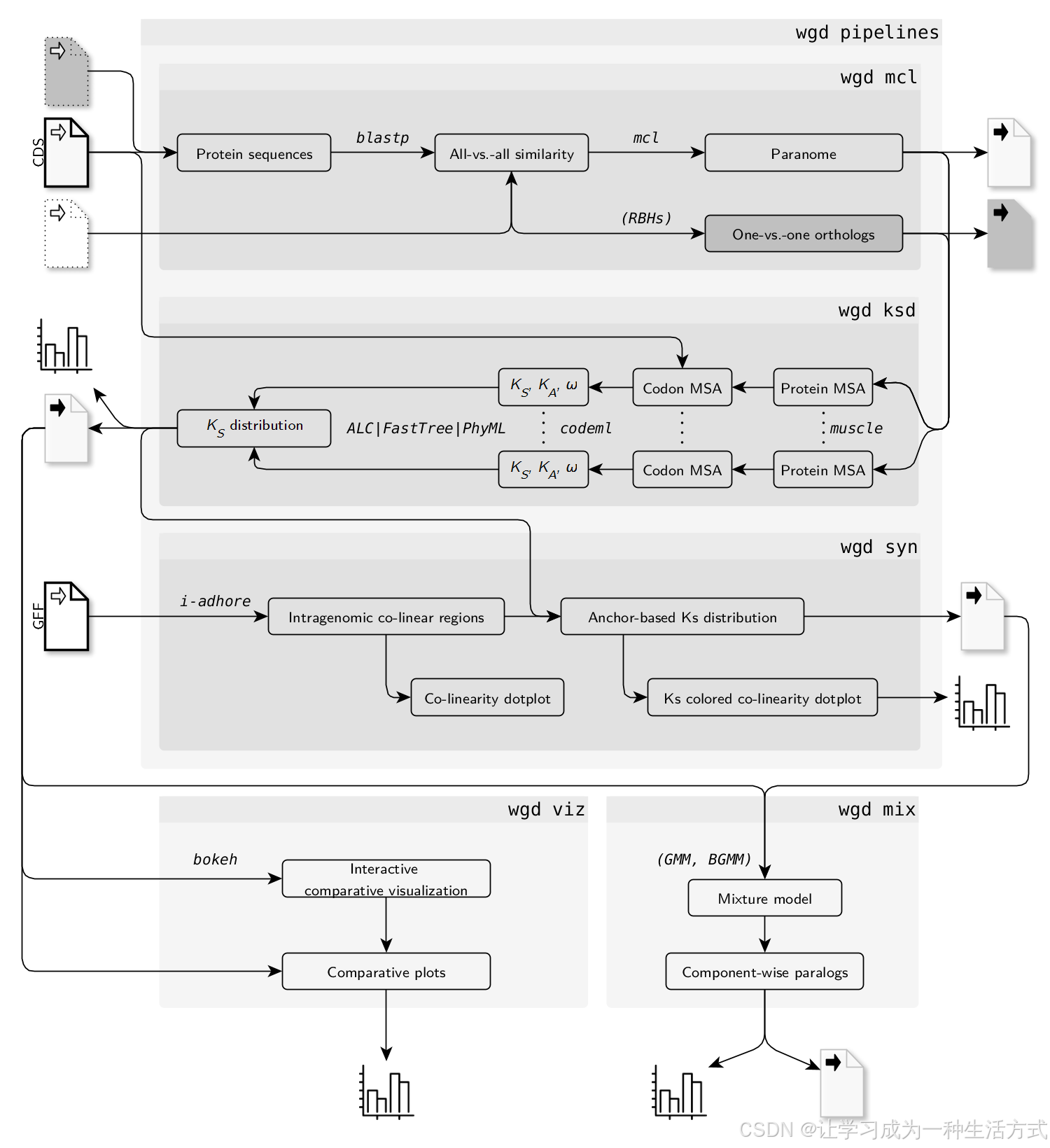
wgd 的主要目的是计算全基因组和物种间直系同源基因的 Ks 分布。以下是几种常见的用法:
计算 CDS 序列的全基因组 Ks 分布:
$ wgd dmd ath.cds.fasta $ wgd ksd wgd_dmd/ath.cds.fasta.mcl ath.cds.fasta计算物种间直系同源基因的 Ks 分布:
$ wgd dmd ath.cds.fasta vvi.cds.fasta $ wgd ksd wgd_dmd/ath1000.fasta_vvi1000.fasta.rbh ath.cds.fasta vvi.cds.fasta
比较一键傻瓜式出结果,方便!
Singularity 容器
wgd 提供了 Singularity 容器,可以让你在不安装所有必需软件的情况下使用 wgd 的所有工具。若要安装 Singularity,请按照此处的说明进行操作。
一旦安装了 Singularity(并且在 Windows 或 Mac 的虚拟机中运行时),你可以在本地构建容器镜像(需要 root 权限)。首先,从 wgd GitHub 仓库获取 Singularity 定义文件,然后运行构建命令:
git clone https://github.com/arzwa/wgd.git
cd wgd
sudo singularity build wgd.sif Singularity
然后可以使用 wgd,如下所示:
singularity exec wgd.sif wgd <command>
如果没有 root 权限,可以从 Singularity Hub 拉取旧版容器(但不支持 syn 和 dmd 命令):
singularity pull --name wgd.simg shub://arzwa/wgd
注意事项
Bug 跟踪:如果程序崩溃、意外退出或出现异常结果,请在感兴趣的子命令之前使用
--verbosity debug参数重新运行(例如wgd --verbosity debug ksd gf.mcl cds.fasta)。如果问题持续存在,请在 GitHub 上报告问题。输入数据:wgd 套件已通过 PLAZA 平台的数据进行广泛测试,因此可以参考该平台的示例数据格式(特别是 CDS fasta 文件和 GFF 文件)来确保数据格式正确。一般建议不要在基因 ID 中使用管道符号(|),因为它在 wgd 中的某些部分具有特殊含义。
虚拟环境:你可以在虚拟环境中安装 wgd(使用
virtualenv)。如果遇到运行可执行文件时的问题(例如wgd --help无效),可以直接调用 CLI,使用python3 ./wgd_cli.py --help(假设你在克隆 wgd 的目录中)。
参考文献
Chen, H., & Zwaenepoel, A. (2023). Inference of Ancient Polyploidy from Genomic Data. In Polyploidy: Methods and Protocols (pp. 3-18). New York, NY: Springer US.
