深入详解随机森林在放射治疗计划优化中的应用及实现细节

🧑 博主简介:CSDN博客专家、CSDN平台优质创作者,高级开发工程师,数学专业,10年以上C/C++, C#,Java等多种编程语言开发经验,拥有高级工程师证书;擅长C/C++、C#等开发语言,熟悉Java常用开发技术,能熟练应用常用数据库SQL server,Oracle,mysql,postgresql等进行开发应用,熟悉DICOM医学影像及DICOM协议,业余时间自学JavaScript,Vue,qt,python等,具备多种混合语言开发能力。撰写博客分享知识,致力于帮助编程爱好者共同进步。欢迎关注、交流及合作,提供技术支持与解决方案。\n技术合作请加本人wx(注明来自csdn):xt20160813
深入详解随机森林在放射治疗计划优化中的应用及实现细节
随机森林(Random Forest)作为一种集成学习算法,通过构建多棵决策树并结合投票或平均机制进行预测,具有高准确性、鲁棒性和可解释性。在放射治疗计划优化领域,随机森林因其能有效处理高维特征、整合多模态数据(如影像和临床数据)以及对噪声数据的鲁棒性,被广泛应用于靶区勾画和剂量分布预测。本文将深入探讨随机森林在放射治疗计划优化中的具体应用场景(靶区分割和剂量预测),详细讲解原理和实现细节,希望对你的学习有所帮助。

1. 随机森林在放射治疗计划优化中的应用概述
1.1 应用背景
放射治疗是肿瘤治疗的重要手段,旨在通过精确的辐射剂量杀灭肿瘤细胞,同时尽量减少对周围健康组织的损伤。优化放射治疗计划需要解决两个关键问题:
- 靶区勾画:精确勾画肿瘤区域(靶区)和需要保护的危及器官(OARs, Organs at Risk),以确保辐射集中在肿瘤区域。
- 剂量分布预测:根据患者影像和临床数据,预测最佳辐射剂量分布,平衡治疗效果和副作用。
随机森林在这些任务中通过提取CT或MRI图像的特征(如肿瘤边界、纹理、灰度值)并结合临床数据(如患者年龄、病史、肿瘤分期),实现自动化靶区分割和个性化的剂量预测。其优势包括:
- 高维特征处理:医学影像特征(如纹理、形状)通常高维,随机森林通过特征选择和重要性分析有效降低维度。
- 多模态数据整合:能无缝整合影像特征和非影像数据(如临床参数)。
- 鲁棒性:对噪声(如CT图像伪影)不敏感,适合复杂医学数据。
- 可解释性:提供特征重要性排名,辅助医生理解模型决策依据。
1.2 具体应用场景
- 靶区分割:基于CT图像提取肿瘤边界特征,自动勾画放疗靶区(如肿瘤体积和危及器官)。
- 剂量预测:结合CT/MRI图像特征和临床数据,预测最佳放疗剂量分布。
2. 应用场景一:靶区分割
2.1 原理
靶区分割的目标是将CT或MRI图像中的肿瘤区域(GTV, Gross Tumor Volume)与正常组织和危及器官区分开。随机森林通过以下步骤实现:
- 图像预处理:对CT/MRI图像进行去噪、标准化(如灰度归一化)和分割预处理。
- 特征提取:提取肿瘤区域的特征,包括:
- 几何特征:肿瘤的体积、形状(如长轴、短轴)、边界不规则性。
- 纹理特征:基于灰度共生矩阵(GLCM)的对比度、相关性、熵等。
- 灰度特征:区域的平均灰度值、标准差、直方图统计量。
- 随机森林分类:将每个像素(或体素)分类为“肿瘤”或“非肿瘤”,生成分割掩码。
- 后处理:平滑分割边界,消除孤立点,生成最终靶区轮廓。
随机森林通过构建多棵决策树,每棵树基于随机采样的特征和数据子集进行训练,最终通过投票机制确定每个像素的类别。其鲁棒性来源于对特征噪声的容忍和对过拟合的抑制。
2.2 实现流程
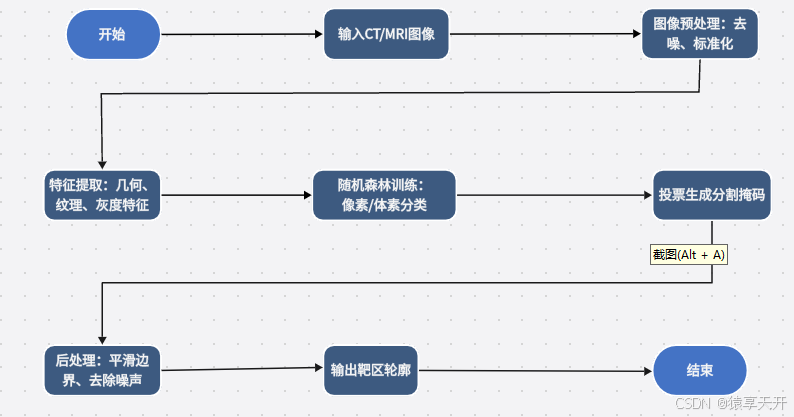
以下是靶区分割的实现流程,图示如下:
graph TDA[输入CT/MRI图像] --> B[图像预处理<br>去噪、标准化]B --> C[特征提取<br>几何、纹理、灰度特征]C --> D[随机森林训练<br>像素/体素分类]D --> E[投票生成分割掩码]E --> F[后处理<br>平滑边界、去除噪声]F --> G[输出靶区轮廓]
2.3 代码实现
以下是一个基于Python的靶区分割示例,使用scikit-learn的随机森林分类器和SimpleITK处理医学影像。
import SimpleITK as sitk
import numpy as np
from sklearn.ensemble import RandomForestClassifier
from skimage.feature import greycomatrix, greycoprops
import matplotlib.pyplot as plt# 1. 读取和预处理CT图像
def load_image(image_path):"""读取CT图像并进行预处理(去噪、标准化)Args:image_path: CT图像路径(DICOM或NIfTI格式)Returns:image: 预处理后的图像数组"""image = sitk.ReadImage(image_path)image = sitk.Cast(sitk.RescaleIntensity(image, 0, 255), sitk.sitkUInt8) # 归一化到0-255image_array = sitk.GetArrayFromImage(image)return image_array# 2. 特征提取
def extract_features(image, mask=None):"""提取图像特征:几何、纹理、灰度Args:image: 预处理后的图像数组mask: 可选的ROI掩码Returns:features: 特征矩阵 (n_samples, n_features)"""features = []for z in range(image.shape[0]): # 遍历每个切片slice_img = image[z, :, :].astype(np.uint8)# 灰度共生矩阵(GLCM)纹理特征glcm = greycomatrix(slice_img, distances=[1], angles=[0, np.pi