短讯 | 利用开源 Galaxy 平台简化临床细菌全基因组测序数据分析

临床微生物学家通常都缺乏专业的生物信息学数据分析训练。易用的、带有图形用户界面的工具能够有效降低生物信息数据分析门槛。本文介绍了开源的 Galaxy 平台在简化临床微生物全基因组测序数据分析方面的作用。
方法:构建的生物信息学分析流程包含以下连续工具:
• Fastp(输入数据修剪)
• FastQC(测序读长质量控制)
• SPAdes(基因组组装)
• Quast(基因组组装质量控制)
• Prokka(基因注释)
• Staramr(基于 ResFinder 数据库)和 ABRicate(基于 CARD 数据库),用于抗菌药物耐药性(AMR)基因筛选和分子菌株分型。
对一年内从血流感染患者中分离的所有产超广谱 β 内酰胺酶肠杆菌科细菌的双端短读长全基因组测序数据进行分析。
结果:Galaxy 平台无需命令行工具操作。该生物信息学分析流程可在一小时内构建完成,仅需上传 fastq 文件,即可一步实现基因组从头组装、多位点序列分型(MLST)和抗菌药物耐药性基因筛选的系统化分析。在分析的 66 株产超广谱 β 内酰胺酶菌株中,两种最常见的超广谱 β 内酰胺酶基因是 blaCTX-M-15(62.1%)和 blaCTX-M-27(13.6%)。
结论:开放获取的 Galaxy 平台提供图形界面和易用工具,适合无生物信息学专业人员的临床微生物学实验室常规使用。我们认为,该平台将有助于快速且低成本地进行细菌全基因组测序数据分析,尤其适用于资源有限的环境。
引言
下一代测序(NGS)降低了测序成本并显著提高了测序通量,使得在数小时内对细菌病原体全基因组进行常规测序成为可能,并能获得对获得性抗菌药物耐药性基因相当全面的分析结果 [7,8]。
然而,除参考实验室外,细菌全基因组测序(WGS)在常规诊断策略中很少用于临床菌株分析。这在一定程度上是因为大多数临床微生物学家未接受过直接通过命令行控制台进行生物信息学分析的培训。
Galaxy 是一个生物医学数据分析云平台,提供用户友好的工具和图形界面,允许用户共享、分析和可视化大型数据集(如下一代测序数据)[9]。它提供强大的计算能力、多种生物信息学开源软件以及许多详细说明如何高效使用这些工具的教程。该平台便于构建生物信息学分析流程,用户之间可共享这些流程以进行标准化分析。
生物信息学分析
使用在线开源生物信息学平台 Galaxy 分析测序基因组。2019 年为本研究创建的生物信息学分析流程快照如图 1 所示。使用的工具包括:
• Fastp(Galaxy 版本 0.20.1+galaxy0)用于质量修剪输入数据(Q30 阈值)
• FastQC(Galaxy 版本 0.72+galaxy1)用于测序读长质量控制 [11]
• SPAdes(Galaxy 版本 3.12.0+galaxy1)用于基因组组装 [12]
• Quast(Galaxy 版本 5.0.2+galaxy1)用于基因组组装质量控制
• Prokka(Galaxy 版本 1.14.5+galaxy0)用于基因注释 [13]
• Staramr(Galaxy 版本 0.7.1+galaxy1)(基于质粒数据库 PlasmidFinder 和耐药基因数据库 ResFinder)[14]
• ABRicate(Galaxy 版本 1.0.1)(基于综合抗生素耐药性数据库 CARD),用于抗菌药物耐药性(AMR)基因筛选和分子菌株分型 [15]。
• 通过 β 内酰胺酶数据库(http://bldb.eu/)[16] 确认已鉴定基因的超广谱 β 内酰胺酶活性。
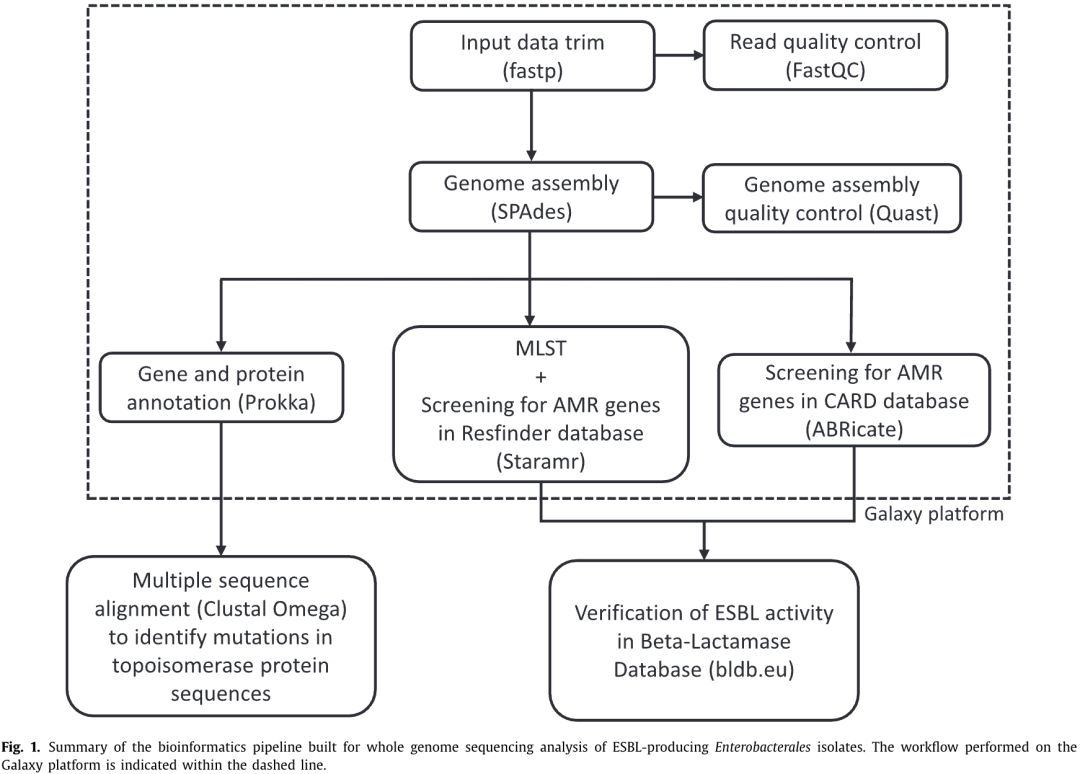
喹诺酮类耐药决定区突变检测。采用 Clustal Omega(https://www.ebi.ac.uk/Tools/msa/clustalo/)进行多序列比对,手动鉴定喹诺酮类耐药决定区(QRDRs)的突变。将分离株拓扑异构酶亚基 GyrA、GyrB、ParC 和 ParE 的蛋白质序列与美国国家生物技术信息中心(https://www.ncbi.nlm.nih.gov/)和通用蛋白质数据库(https://www.uniprot.org/)提供的大肠杆菌、肺炎克雷伯菌、阴沟肠杆菌复合体、产酸克雷伯菌和粘质沙雷氏菌参考菌株的参考序列进行比对分析(见补充材料)。
讨论
尽管下一代测序已彻底改变了医学和诊断学的许多方面,但全基因组测序在常规临床微生物学中的应用仍通常局限于参考中心、罕见耐药机制的鉴定、研究目的或回顾性流行病学监测。
然而,尽管表型抗生素敏感性测试(AST)方法仍是金标准,但近期测序成本的下降和大量抗菌药物耐药性数据库的可用,使全基因组测序成为一种强大的工具,在常规临床微生物学中将有许多具有成本效益的应用。
可使用多种便捷工具分析全基因组测序数据并预测抗菌药物耐药性决定因子;然而,大多数分析流程要么需要生物信息学技能来在多种开放获取工具中进行选择并高效运行,要么需要使用商业解决方案,如:
• 生物梅里埃 Episeq 平台
• 凯杰 CLC 基因组工作台
• Ridom SeqSphere
• Geneious Prime)
2012 年启动的 ResFinder 网络服务器和 2013 年发布的综合抗生素耐药性研究数据库(CARD)为无专业生物信息学技能的用户铺平了道路,通过免费在线提交全基因组测序数据,便于抗菌药物耐药性基因的检测 [8,17,18]。
Galaxy 平台是少数提供图形界面的开放获取解决方案之一,同时仍允许用户选择单个工具构建定制的生物信息学分析流程(例如,选择组装工具和抗菌药物耐药性数据库进行数据分析)并选择多种参数。流程可在用户之间共享,且当工具发布新版本时,每种工具都可轻松更新。
然而,该平台并不总是提供所有生物信息学工具的最新版本,因为这些工具需要额外时间在平台上实施。BV-BRC 平台(https://www.bv-brc.org/app/ComprehensiveGenomeAnalysis)提供另一种易用的图形界面,便于细菌基因组的全面分析;然而,它使用内部流程,无法选择单个工具或参数。
结论
随着下一代测序和生物信息学技术的发展,抗菌药物耐药性预测和基于全基因组测序的诊断工具未来将对应对耐药细菌的传播和快速鉴定多重耐药细菌极为有帮助,有助于缩短有效治疗时间。Galaxy 平台便于缺乏高级生物信息学技能的临床微生物学家进行细菌全基因组测序数据分析。其使用可促进全基因组测序方法在许多临床微生物学环境中的应用,成本低且无需大量培训。
推荐阅读
中国银河生信云平台(UseGalaxy.cn)致力于零代码生信分析。平台拥有海量计算资源、3000 多个生信工具和数十条生信流程,并且为用户提供 200G 免费存储空间。我们还为进阶用户提供高质量课程培训:
培训课程 | 第2期《RNA-seq数据分析实战》,现在报名,即可获得上期课件和直播回放视频
