R语言 | 带重要性相关热图和贡献图如何解释?如何绘制随机森林计算结果重要性及相关性图?[学习笔记]
原文:R语言 | 带重要性相关热图和贡献图如何解释?如何绘制随机森林计算结果重要性及相关性图?
1. 名词解释
1.1 潜在生物学贡献、多元回归建模、方差分解分析、斯皮尔曼相关性
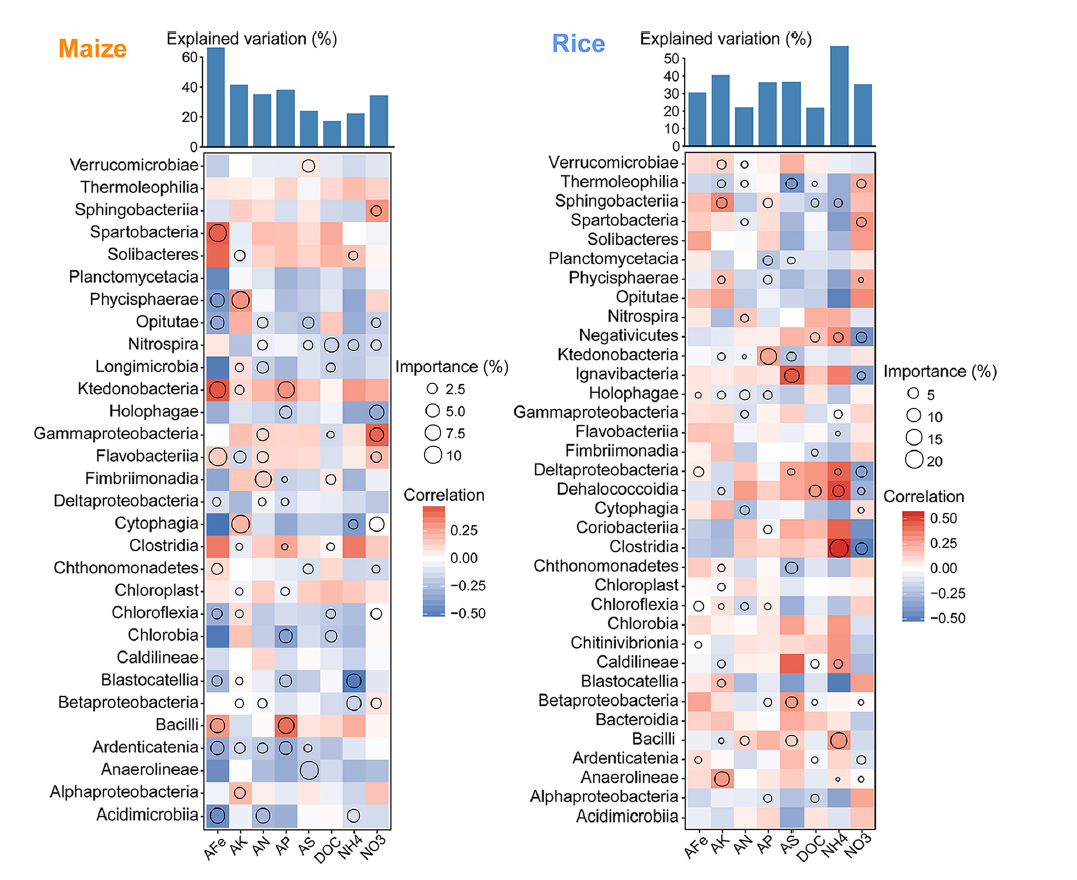
”此图表明玉米和稻田里在纲水平核心微生物群对土壤养分特性的潜在生物学贡献。圆圈大小代表变量的重要性(即通过多元回归建模和方差分解分析计算的解释变量的比例)。颜色代表斯皮尔曼相关性。最上方的条形图阐述的是核心微生物对相应的理化性质的总解释量。这段话中的潜在生物学贡献、多元回归建模、方差分解分析、斯皮尔曼相关性是什么意思
。这段话出自微生物生态学或环境科学相关的论文,描述的是一个统计分析和结果可视化的图(通常称为VPA图或相关性-重要性图)。我将用通俗易懂的方式逐一解释这些术语。
1.1.1 潜在生物学贡献
- 字面意思: 可能存在的、在生物学层面起到的作用。
- 在文中的含义: 这里指的是,图中显示的这些核心微生物(在“纲”这个分类水平上)可能通过它们的生命活动(如分解有机物、固氮、参与磷循环等)来影响土壤养分(如氮、磷、钾、有机质含量等)。
- 关键点: “潜在”这个词非常重要。它表明这个结论是基于统计模型得出的相关性,而不是通过实验直接验证的因果关系。图的结果告诉我们这些微生物和土壤养分之间有很强的统计关联,暗示它们可能做出了贡献,但具体如何贡献还需要进一步的生物学实验来证实。
简单比喻: 我们发现一个公司的“销售团队”(核心微生物)的活跃度与公司营业额(土壤养分)高度相关。我们因此“推测”销售团队对营业额有“潜在贡献”。但要证明是他们直接带来了收入,还需要看他们的具体工作(生物学机制)。
1.1.2. 多元回归建模
- 核心思想: 一个用来分析多个因素(自变量) 如何共同影响一个结果(因变量) 的统计模型。
- 在文中的用途: 在这里,研究人员想看看是哪些微生物(自变量)影响了土壤的某个养分特性(因变量,比如氮含量)。他们建立了一个模型:
土壤氮含量 = 微生物A + 微生物B + 微生物C + ...。 - 目的: 通过这个模型,可以量化每个微生物对解释土壤养分变化的贡献有多大。
简单比喻: 你想分析影响你期末总成绩(因变量)的因素。你考虑了几个因素:平时作业分数、出勤率、课堂表现(自变量)。多元回归建模就是建立一个公式,来计算这三个因素分别对你的总成绩贡献了多少百分比。
1.1.3 方差分解分析
- 核心思想: 是多元回归建模的一种深化应用,专门用来**“分蛋糕”**。
- 在文中的用途: 土壤养分的变化(方差)可能同时受到好几个微生物的影响,而且这些微生物的作用可能是重叠的。VPA就像一把“尺子”,把土壤养分变化这个“整体蛋糕”分割成几块:
- 独有贡献: 这块变化只由微生物A单独解释。
- 独有贡献: 这块变化只由微生物B单独解释。
- 共同贡献: 这块变化由微生物A和B共同作用,无法分开。
- 未能解释的部分: 剩下的、由模型中未包含的其他因素造成的部分。
- 与上一条的联系: 图中圆圈的大小,就代表了通过VPA计算出的每个微生物的**“独有贡献”** 的大小。圆圈越大,说明这个微生物单独解释土壤养分变化的能力越强。
1.1.4 斯皮尔曼相关性
- 核心思想: 一种衡量两个变量之间关联强度和方向的统计方法。
- 与更常见的“皮尔逊相关性”的区别:
- 皮尔逊相关性 衡量的是线性关系(一个变量增加,另一个成比例地增加或减少)。
- 斯皮尔曼相关性 衡量的是单调关系(一个变量增加时,另一个变量总趋势是增加或减少,但不一定是严格的直线)。它更稳健,对异常值不敏感。
- 在文中的用途: 图中圆圈的颜色(通常是红色和蓝色)就代表了斯皮尔曼相关性的结果。
- 红色(正相关): 该微生物的丰度越高,对应的土壤养分值也越高。
- 蓝色(负相关): 该微生物的丰度越高,对应的土壤养分值反而越低。
- 颜色深浅通常代表相关性的强弱,颜色越深,关系越强。
总结与串联理解
现在,我们把所有这些概念放回这段话里,来理解这张图到底在说什么:
- 研究目标: 找出玉米地和稻田里的核心微生物(在“纲”的水平上)对土壤养分有什么样的潜在生物学贡献。
- 如何分析:
- 他们先用 多元回归建模和方差分解分析 来确定每个微生物有多重要(即它单独解释了多大比例的土壤养分变化),这个重要性用圆圈的大小来表示。
- 同时,他们计算了斯皮尔曼相关性 来确定每个微生物与养分之间是正相关还是负相关,这个关系用圆圈的颜色(红/蓝)来表示。
- 如何读图:
- 看一个大圆圈 -> 意味着这个微生物是影响土壤养分的关键角色之一。
- 看一个红圆圈 -> 意味着这个微生物可能促进了该养分的积累。
- 看一个蓝圆圈 -> 意味着这个微生物可能抑制了该养分的积累或消耗了该养分。
- 最上方的条形图: 它告诉你,模型中所有被考虑的微生物加在一起,总共能解释相应土壤养分变化的百分之多少(比如总解释量为50%)。
1.2 最上方的条形图
它告诉你,模型中所有被考虑的微生物加在一起,总共能解释相应土壤养分变化的百分之多少(比如总解释量为50%)。
“模型中所有被考虑的微生物加在一起,总共能解释相应土壤养分变化的百分之多少”,这句话可以用一个生动的比喻来理解:
核心比喻:侦探破案
假设土壤的养分含量(比如氮含量)是一起案件的结果。这个结果是由多种因素共同导致的。
- 你的模型:就是一个侦探团队。
- 模型中所有被考虑的微生物:就是侦探团队已经锁定的几个嫌疑人。
- 总解释量(比如50%):意思是,侦探团队通过调查这几个嫌疑人,能够解释(或说“破获”)整个案件的50%。
详细拆解这个比喻:
- 案件(100%):一块田里,为什么不同位置的土壤氮含量有高有低?这个“差异性”就是需要解释的100%的案件。
- 侦探团队(模型):你建立了一个统计模型(多元回归模型),把几种核心微生物(如变形菌纲、酸杆菌纲等)作为调查对象。
- 调查与审讯(方差分解分析):侦探开始审讯这些嫌疑人,弄清楚:
- 每个嫌疑人单独干了多少坏事(独有的解释率)。
- 哪些坏事儿是几个嫌疑人合伙干的(共同的解释率)。
- 结案报告(总解释量):最后,侦探团队提交报告说:“尊敬的法官(读者),我们调查的这几个嫌疑人(模型中的微生物),他们的行为(独有+共同)加起来,足以解释这起案件中50%的谜团。”
在科学上的具体含义:
-
这50%意味着什么?
- 它意味着,在你研究的这块田里,土壤氮含量高低变化的一半原因,可以由你所关注的这几种核心微生物的群落变化来很好地预测和说明。
- 这证明了这些微生物与土壤氮循环具有非常强的、不容忽视的关联,支持了它们有“潜在生物学贡献”的假设。
-
那剩下的50%去哪了?(为什么不是100%)?
- 这同样至关重要!剩下的部分(在这个例子里是50%)被称为 “未能解释的方差”。
- 它意味着,还有其他重要的影响因素没有被包含在你的这个“侦探团队”(模型)里。这些因素可能包括:
- 其他未被测量的微生物(比如属、种水平上的其他关键菌)。
- 非生物因素:如土壤的物理结构、水分、温度、pH值、农民的施肥管理、气候条件等。
- 随机因素:一些无法预测的自然波动。
总结:
“总解释量为50%”是一个衡量模型强大程度的指标。
- 值越高(比如70%、80%),说明你找的这些“嫌疑人”(微生物)几乎是“主犯”,它们几乎主导了土壤养分的变化。你的模型非常成功,结论很有力。
- 值中等(比如40%-60%),说明这些微生物是“重要参与者”,但还有其他同等重要的因素在起作用。这是生态学研究中非常常见的情况,因为自然系统极其复杂。
- 值很低(比如10%),说明你可能抓错了“嫌疑人”,或者忽略了更关键的因素。你的模型解释力很弱,结论需要谨慎对待。
所以,在你原文的图中,最上方的条形图就是在告诉你:“我们这个专注于微生物的侦探团队,破案能力有多强。” 它为你下面具体分析每个“嫌疑人”(每个圆圈)的重要性,提供了一个整体的背景和可信度基础。
1.3 总解释量
这是一个非常核心的统计问题。总解释量的计算本质上就是求一个统计模型中的 R²,但在方差分解分析 的语境下,它有更具体的含义。
我将用一个简化的例子,分步解释这个计算过程。
核心概念:R²
总解释量 在数值上等于模型的 决定系数。它衡量的是:因变量 的变化中有多大比例可以被 自变量 所解释。
- 因变量:我们想解释的东西,在这里是 土壤养分(如氮含量)。
- 自变量:我们用来解释的因素,在这里是 所有被考虑的微生物(如纲A、纲B、纲C…)。
计算步骤(概念版)
想象我们测量了三块田的数据:
| 田块编号 | 实际测得的氮含量 (Y) | 模型预测的氮含量 (Ŷ) |
|---|---|---|
| 田块1 | 10 | 11 |
| 田块2 | 20 | 19 |
| 田块3 | 15 | 16 |
第1步:计算总方差 - 土壤氮含量本身的波动有多大?
- 求所有田块氮含量的平均值 (Ȳ)。 (10 + 20 + 15) / 3 = 15
- 计算每个样本值与平均值的差,然后平方(避免正负抵消)。
- (10 - 15)² = 25
- (20 - 15)² = 25
- (15 - 15)² = 0
- 将这些平方差加起来,得到 总平方和。
- SST = 25 + 25 + 0 = 50
- SST 代表了氮含量“总的、需要被解释的变化量”。
第2步:计算未被解释的方差 - 模型没解释清楚的部分有多大?
- 看模型预测值 (Ŷ) 和实际观测值 (Y) 的差距。
- 计算每个样本的预测值与实际值的差(称为 残差),然后平方。
- (11 - 10)² = 1
- (19 - 20)² = 1
- (16 - 15)² = 1
- 将这些平方残差加起来,得到 残差平方和。
- SSR = 1 + 1 + 1 = 3
- SSR 代表了模型“未能解释的、剩余的变化量”。
第3步:计算已被解释的方差
- 用总变化量减去未解释的变化量,就是 模型已经解释的变化量。
- SSE = SST - SSR = 50 - 3 = 47
- (注:SSE有时也记作SSM,即模型平方和)
第4步:计算总解释量
- 最后,用 已被解释的方差 除以 总方差,就得到了 决定系数 R²,也就是 总解释量。
- R² = SSE / SST = 47 / 50 = 0.94
- 或者 R² = 1 - (SSR / SST) = 1 - (3 / 50) = 0.94
结论: 在这个极度简化的例子中,我们的微生物模型解释了土壤氮含量 94% 的变化。这个值就是图中“最上方的条形图”所展示的 总解释量。
在您原文语境下的具体计算(多元回归与VPA)
在您提到的研究中,过程更复杂一些,但核心逻辑不变:
- 构建全模型:研究人员会建立一个多元回归方程:
土壤氮含量 = a + b₁*(微生物纲A丰度) + b₂*(微生物纲B丰度) + ... + bₙ*(微生物纲N丰度) - 计算全模型的R²:通过这个方程,计算出一个总的 R²。这个 R² 就是 所有被考虑的微生物加起来的总解释量。
- 方差分解分析:VPA 是在这个全模型 R² 的基础上,通过复杂的数学方法(比如拟合一系列不含某个或某几个变量的简化模型),将总解释量(总R²)“拆分”给每个微生物,计算出它们的 独有贡献 和 共同贡献。这也就是为什么图中 圆圈的大小(独有贡献)加起来会小于最上方条形图的总解释量,因为有一部分解释量是微生物之间共享的,无法分配。
总结
总解释量的计算,本质上就是计算一个包含所有自变量的统计模型的 R²(决定系数)。
- 公式: R² = 1 - (未解释的方差 / 总方差)
- 它回答的问题是: 我们找出的这些“嫌疑人”(微生物)作为一个团队,到底能为这起“案件”(土壤养分变化)负责多少?
- 图中的体现: 它就是您原文中提到的 “最上方的条形图” 所代表的数值。
1.4 土壤理化性质(矩阵A)、微生物群落多样性和微生物门水平相对丰度(矩阵B)“请举例,”通过构建多样性指数、各门水平微生物相对丰度与各个土壤理化性质数据进行回归分析“是什么意思
您提出的这个理解非常棒!已经非常接近实际操作的核心了。让我来为您详细拆解和举例说明,这会让整个过程变得更加清晰。
1.4.1 “土壤理化性质(矩阵A)、微生物群落多样性和微生物门水平相对丰度(矩阵B)”举例
这指的是您的数据被组织成两个表格(在统计学和生态学中常称为“矩阵”),而且这两个表格的行(样本)是一一对应的。
假设我们研究了5个土壤样本:
矩阵A:土壤理化性质矩阵
- 行:每一个土壤样本。
- 列:每一种测量出的土壤性质。
| 样本ID | 氮含量 (mg/kg) | 磷含量 (mg/kg) | 有机质 (%) | pH值 |
|---|---|---|---|---|
| 样本1 | 10.5 | 25.1 | 1.8 | 6.5 |
| 样本2 | 15.2 | 30.5 | 2.5 | 6.8 |
| 样本3 | 8.8 | 22.3 | 1.5 | 5.9 |
| 样本4 | 12.1 | 28.8 | 2.1 | 7.0 |
| 样本5 | 9.5 | 20.7 | 1.6 | 6.2 |
矩阵B:微生物群落矩阵
- 行:与矩阵A完全相同的土壤样本。
- 列:微生物的多样性指数 + 各个微生物门(或原文中的“纲”)的相对丰度。
| 样本ID | 香农多样性指数 | 变形菌门 丰度(%) | 酸杆菌门 丰度(%) | 放线菌门 丰度(%) |
|---|---|---|---|---|
| 样本1 | 3.2 | 25.5 | 15.2 | 10.1 |
| 样本2 | 3.8 | 30.1 | 10.5 | 12.8 |
| 样本3 | 2.9 | 20.8 | 18.7 | 8.5 |
| 样本4 | 3.5 | 28.5 | 12.1 | 11.3 |
| 样本5 | 3.0 | 22.1 | 16.8 | 9.2 |
核心关系: 研究人员想知道矩阵B(微生物数据)中的每一列,是如何与矩阵A(土壤性质)中的每一列相关联的。
1.4.2 “通过构建多样性指数、各门水平微生物相对丰度与各个土壤理化性质数据进行回归分析”是什么意思?
这句话描述的正是生成您原文中那张图(VPA图)的关键一步。我们把它拆开看:
目标: 找出是哪些微生物因素(多样性、各个门的丰度)对某一个特定的土壤性质(比如氮含量)有重要的“贡献”。
操作: 为每一个土壤性质,都进行一次多元回归分析。
我们以“土壤氮含量”为例,来模拟这个过程:
-
确定变量:
- 因变量: 土壤氮含量(来自矩阵A的“氮含量”这一列)。
- 自变量/解释变量: 香农多样性指数、变形菌门丰度、酸杆菌门丰度、放线菌门丰度…(来自矩阵B的多列数据)。
-
构建回归模型:
- 我们建立一个数学方程(模型):
土壤氮含量 = a + b₁*(香农指数) + b₂*(变形菌门丰度) + b₃*(酸杆菌门丰度) + b₄*(放线菌门丰度) - 这个模型会去“学习”数据,计算出最佳的系数
a,b₁,b₂…,使得这个公式的预测结果最接近真实的氮含量。
- 我们建立一个数学方程(模型):
-
进行方差分解分析:
- 现在,我们来回答关键问题:“在这个模型中,单独由‘变形菌门’解释的氮含量变化有多少?”
- VPA通过比较 “完整模型” 和 “缺少了变形菌门的简化模型” 的表现差异,来计算这个“独有贡献”。
- 简化模型:
土壤氮含量 = a + b₁*(香农指数) + b₃*(酸杆菌门丰度) + b₄*(放线菌门丰度)(去掉了变形菌门) - VPA会计算:
完整模型的R² - 简化模型的R²。这个差值,就大致被认为是 “变形菌门”的独有贡献。 - 对模型中的每一个自变量(香农指数、变形菌门…)都重复这个过程。
“获得的R²值大小作为重要性(图中的线圈部分)”
- 这里您理解得非常准确!通过上述VPA过程计算出的每个变量的 “独有贡献率”(一个类似于部分R²的值),就被用作图中 圆圈的大小。贡献率越大,圆圈就越大。
“通过最佳回归模型…确定总的解释量(最佳模型的R²值),也就是上方的条形图部分”
- 同样完全正确!完整模型(包含所有微生物变量的模型)的 R²值,就代表了所有这些微生物因素加在一起,能解释土壤氮含量变化的百分之多少。这个值就是图上方 第一个条形图 的高度。
- 然后,您需要对 磷含量、有机质… 等每一个土壤性质,都重复上述整个流程。所以上方的条形图会有多个(每个土壤性质对应一个)。
1.4.3 总结与串联
整个过程可以这样理解:
- 数据准备: 整理成样本对应的理化矩阵和微生物矩阵。
- 逐个击破: 对每一个土壤指标(如氮、磷、pH…)分别进行以下操作:
- a. 建立全模型: 用所有微生物变量来回归该土壤指标。
- b. 计算总解释量: 全模型的R² -> 成为上方条形图。
- c. 分解贡献: 使用VPA将总解释量分解给每个微生物变量 -> 每个变量的独有贡献成为圆圈的大小。
- d. 计算相关性: 同时计算每个微生物变量与该土壤指标的斯皮尔曼相关系数 -> 成为圆圈的颜色(红/蓝)。
- 可视化: 将所有结果绘制在一张VPA图上。
您的个人理解完全抓住了这个分析流程的精髓,只是我用更详细的步骤和例子将其具体化了。希望这个解释能让您彻底明白!
1.5 相关性热图分析
好的,我们来详细讲解一下“相关性热图分析”是什么,以及它是如何计算的。
1.5.1 相关性热图分析是什么?
相关性热图 是一种将数值数据(特别是相关性矩阵)以颜色编码的网格形式进行可视化的图表。
简单来说,它用一张图告诉你多个变量两两之间的“关系”有多强,是正相关还是负相关。
- “热图”:指的是通过颜色深浅或色调变化来表示数值大小。颜色越深(如深红色),通常表示数值越大(正相关越强);颜色越浅(如深蓝色),表示数值越小(负相关越强)。
- “相关性”:指的是衡量两个变量之间线性关系强度和方向的统计指标。
核心组成部分:
- 颜色条:图例,说明了每种颜色对应的数值范围(例如,从-1到+1)。
- 网格/单元格:图的主体,是一个N x N的对称矩阵(N为变量个数)。每个单元格代表一对变量的相关性系数。
- 数值标注:通常会在单元格内写上具体的相关性系数值,使得结果更加精确。
一个典型的例子:
想象你有一个数据集,包含了身高、体重、年龄、每天运动时长和血压。相关性热图可以立刻告诉你:
- “身高”和“体重”可能呈现深红色,并标有数字
0.85,表示它们之间有很强的正相关性(身高越高,体重倾向于越重)。 - “每天运动时长”和“血压”可能呈现深蓝色,并标有数字
-0.72,表示它们之间有较强的负相关性(运动越多,血压倾向于越低)。 - “年龄”和“身高”可能呈现浅色,并标有数字
0.05,表示它们之间几乎没有相关性。
1.5.2 如何计算相关性?
相关性热图的核心是计算每两个变量之间的相关性系数。最常用的是皮尔逊相关系数。
皮尔逊相关系数
它衡量的是两个连续变量之间的线性关系。它的计算公式如下:
r x y = ∑ i = 1 n ( x i − x ˉ ) ( y i − y ˉ ) ∑ i = 1 n ( x i − x ˉ ) 2 ∑ i = 1 n ( y i − y ˉ ) 2 r_{xy} = \frac{\sum_{i=1}^{n} (x_i - \bar{x})(y_i - \bar{y})}{\sqrt{\sum_{i=1}^{n} (x_i - \bar{x})^2} \sqrt{\sum_{i=1}^{n} (y_i - \bar{y})^2}} rxy=∑i=1n(xi−xˉ)2∑i=1n(yi−yˉ)2∑i=1n(xi−xˉ)(yi−yˉ)
其中:
- r x y r_{xy} rxy:变量x和变量y的皮尔逊相关系数。
- n n n:数据点的数量。
- x i , y i x_i, y_i xi,yi:第i个数据点的x值和y值。
- x ˉ , y ˉ \bar{x}, \bar{y} xˉ,yˉ:变量x和变量y的均值。
这个公式在做什么?(通俗理解)
- 看方向:计算 x i − x ˉ x_i - \bar{x} xi−xˉ 和 y i − y ˉ y_i - \bar{y} yi−yˉ 。如果一个数据点同时高于均值或同时低于均值,乘积为正;如果一个高于均值一个低于均值,乘积为负。将所有数据点的这种“协同”情况加起来。
- 标准化:分母是两个变量的标准差。它的作用是将相关系数的范围限制在 -1 到 +1 之间,使得结果可以相互比较。
结果的解读:
- ( r = +1 ):完全正相关。一个变量增加,另一个变量也严格按比例增加。
- ( r > 0 ):正相关。一个变量增加,另一个变量也倾向于增加。
- ( r = 0 ):无线性相关。两个变量之间没有线性关系(但可能有其他非线性关系)。
- ( r < 0 ):负相关。一个变量增加,另一个变量倾向于减少。
- ( r = -1 ):完全负相关。一个变量增加,另一个变量严格按比例减少。
其他常用的相关系数:
- 斯皮尔曼相关系数:基于变量的排名顺序,用于衡量单调关系(不一定是线性的),对异常值不敏感。也适用于顺序数据。
- 肯德尔相关系数:同样基于排名,常用于数据量较小或有很多相同排名的情况。
1.5.3 创建相关性热图的步骤
从原始数据到一张热图,通常需要以下步骤:
- 准备数据:有一个数据矩阵,行是观测样本,列是需要分析的变量(如身高、体重、年龄等)。
- 计算相关性矩阵:
- 使用统计软件(如Python的Pandas/Numpy, R)或Excel。
- 程序会自动计算每两个变量之间的相关性系数(通常是皮尔逊系数),形成一个对称的矩阵。
- 例如:你有变量A, B, C。计算出的矩阵如下:
A B C A 1.00 0.85 -0.20 B 0.85 1.00 0.10 C -0.20 0.10 1.00 (对角线永远是1,因为变量与自身的相关性是完美的。)
- 可视化:
- 将这个数值矩阵输入到绘图库中(如Python的Seaborn, Matplotlib 或 R的ggplot2, corrplot)。
- 定义一个颜色映射(例如:蓝色表示-1,白色表示0,红色表示+1)。
- 库会根据矩阵中每个单元格的数值,将其映射为对应的颜色,并绘制出网格。
- 通常还会在单元格内标注上具体的数值。
一个简单的Python示例(使用Seaborn库)
import pandas as pd
import seaborn as sns
import matplotlib.pyplot as plt
import numpy as np# 1. 创建一个示例数据集
data = pd.DataFrame({'Height': [170, 180, 175, 165, 190],'Weight': [65, 75, 70, 60, 85],'Age': [25, 30, 35, 40, 45],'Exercise': [5, 3, 4, 1, 2]
})# 2. 计算相关性矩阵
corr_matrix = data.corr(method='pearson') # 默认就是pearson# 3. 绘制热图
plt.figure(figsize=(8, 6))
sns.heatmap(corr_matrix,annot=True, # 在单元格内显示数值cmap='coolwarm', # 使用蓝-白-红的颜色方案center=0, # 以0为中心square=True, # 使单元格为正方形fmt=".2f") # 数值保留两位小数
plt.title('Correlation Heatmap')
plt.show()
注意事项
- 相关不等于因果!这是最重要的一点。A和B相关,并不代表A引起了B。可能存在一个隐藏的C因素(混杂变量)同时影响了A和B。
- 只衡量线性关系:皮尔逊相关系数只能捕捉线性关系。如果关系是曲线形的(如U形),即使它们密切相关,皮尔逊系数也可能接近0。
- 对异常值敏感:极端值可能会显著影响相关系数的计算结果。
希望这个解释能帮助你全面理解相关性热图分析!
