单细胞多数据集整合和去除批次效应教程,代做各领域生信分析
单细胞多数据集整合和去除批次效应教程
每个数据集的数据分别单独进行读取单细胞数据构建Seurat分析对象
读取各种来源的单细胞数据构建Seurat分析对象的教程
做这一步的时候可以查看我这篇写的非常详细的教程文章:
【腾讯文档】单细胞分析步骤1读取各种来源格式的单细胞数据集构建seurat分析对象教程
单细胞分析步骤1读取各种来源格式的单细胞数据集构建seurat分析对象教程
以要整合GSE159115,GSE223373,GSE155468这几个数据集为例
这几个数据集都先单独进行读取构建Seurat分析对象
读取GSE159115的数据构建seurat分析对象
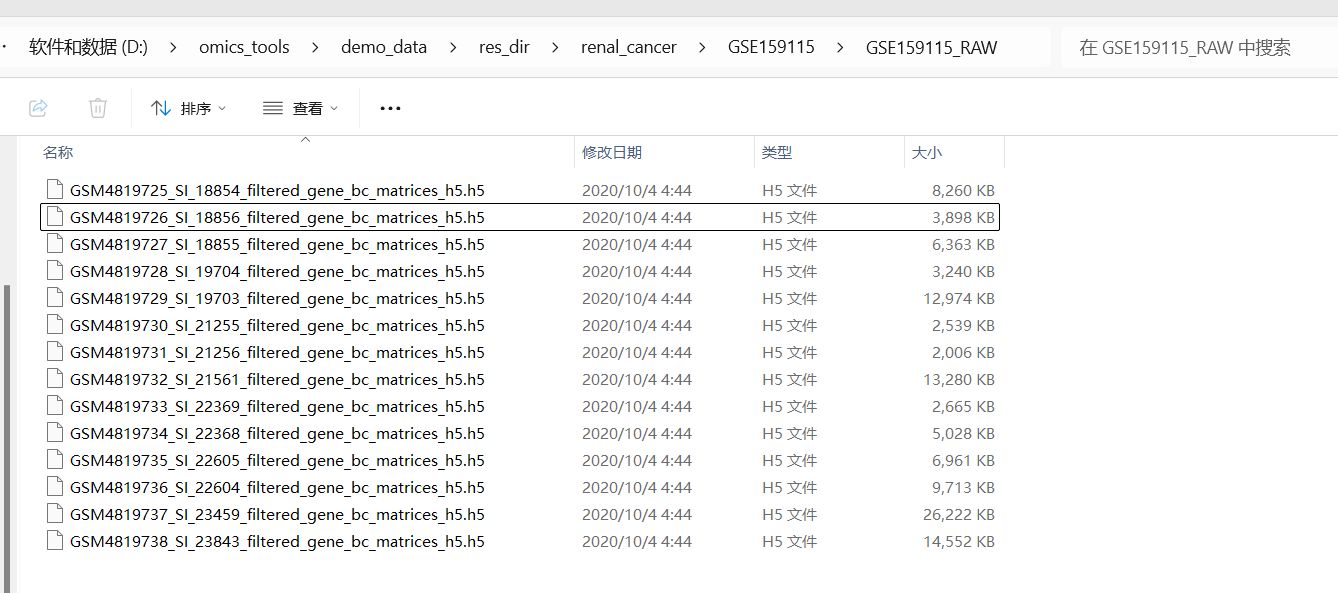
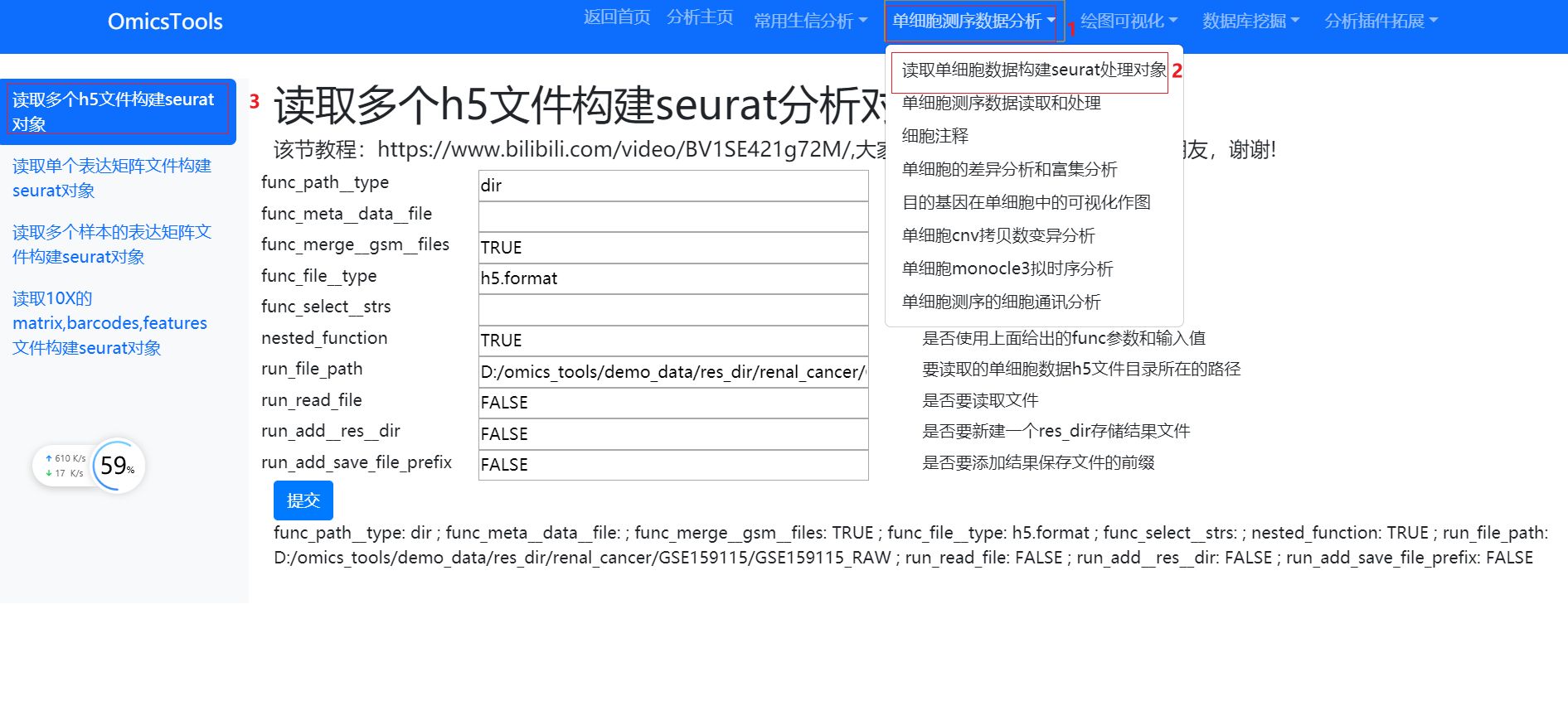
做这一步的时候可以查看我这篇写的非常详细的教程文章:
【腾讯文档】单细胞分析步骤1读取各种来源格式的单细胞数据集构建seurat分析对象教程
https://docs.qq.com/doc/DWWx1eUFaRUV3TW5S
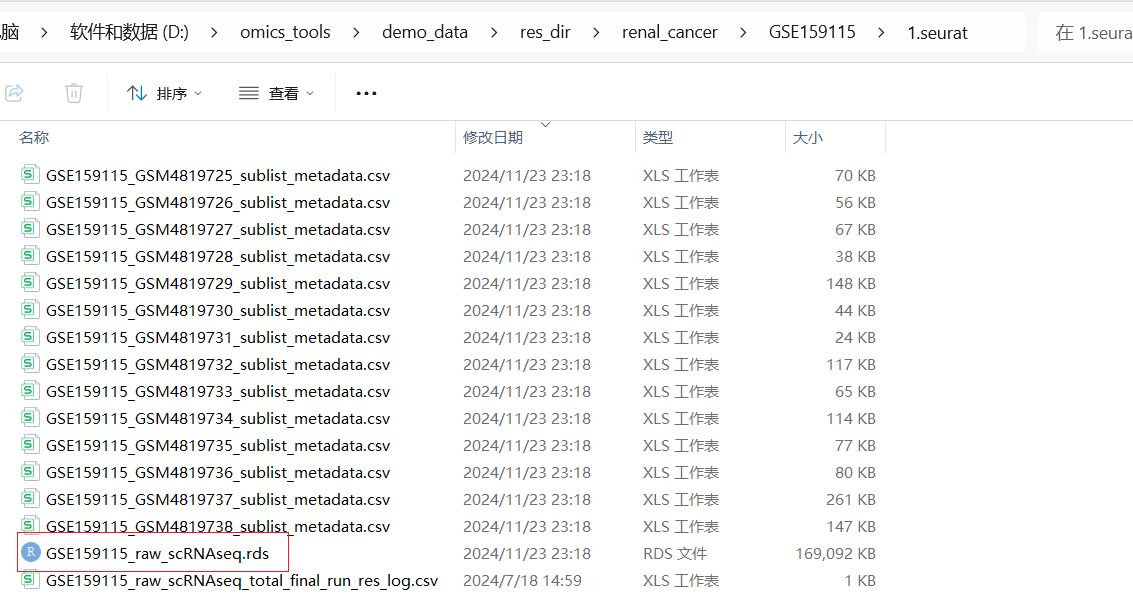
软件运行结果文件得到构建好的seurat对象的rds文件和metadata文件
读取GSE223373的数据构建seurat分析对象
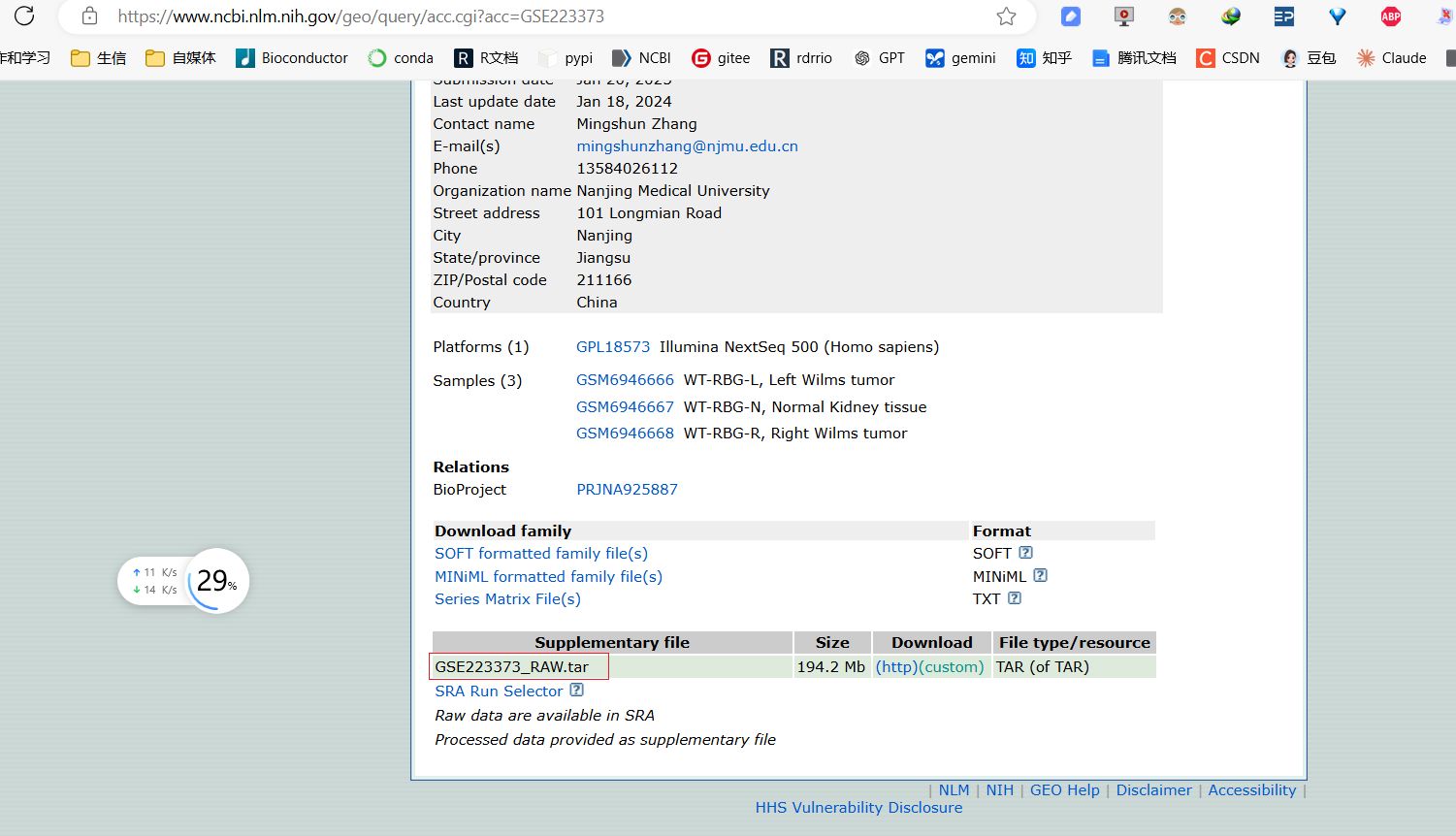
这是一个10X的数据集,做这一步的时候可以查看我这篇写的非常详细的教程文章:
【腾讯文档】单细胞分析步骤1读取各种来源格式的单细胞数据集构建seurat分析对象教程
https://docs.qq.com/doc/DWWx1eUFaRUV3TW5S
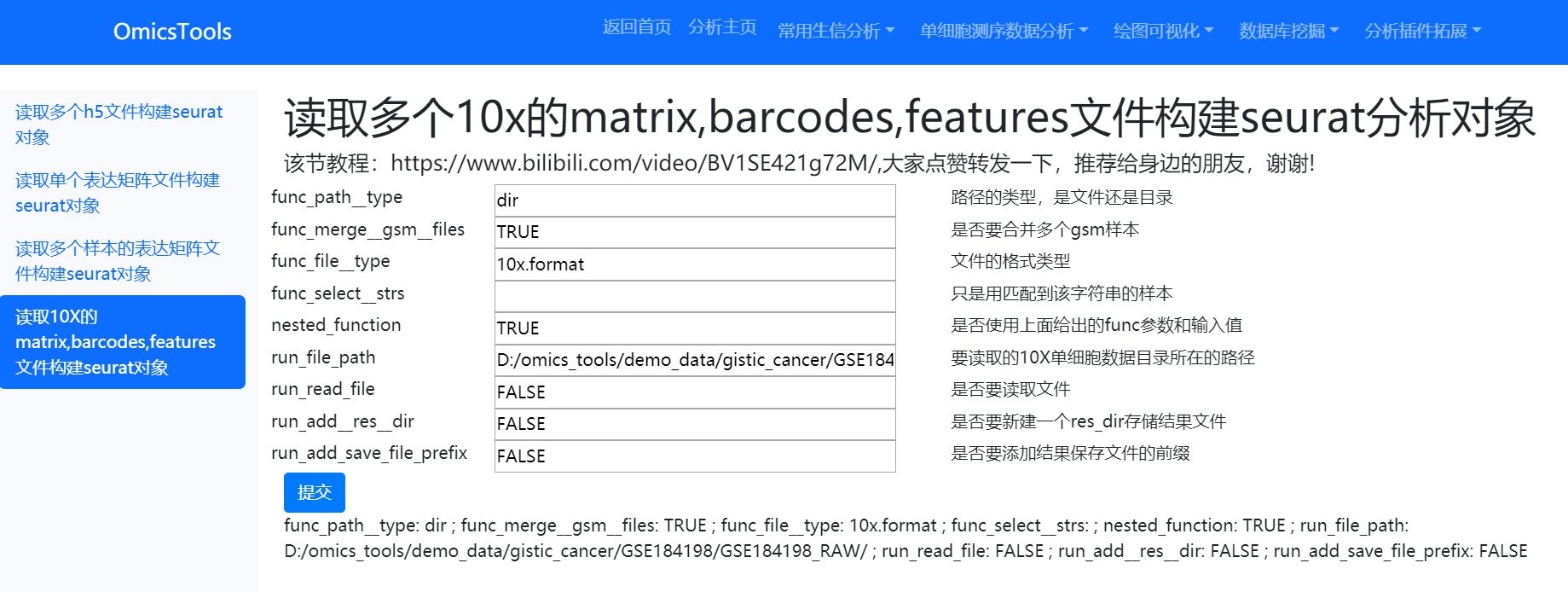
软件运行结果文件得到构建好的seurat对象的rds文件和metadata文件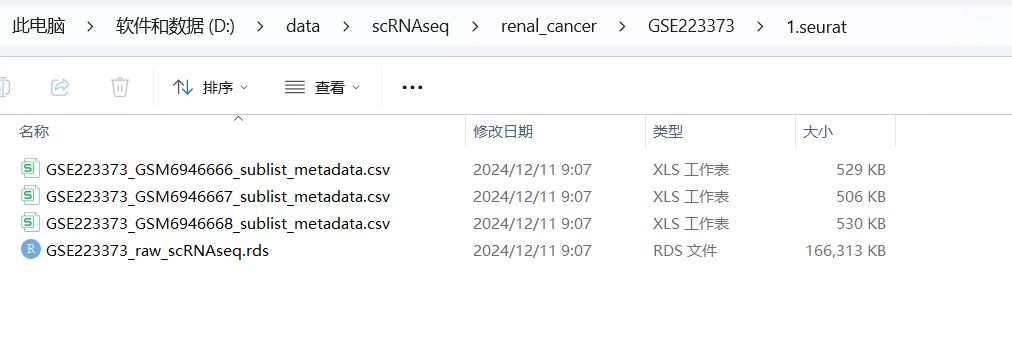
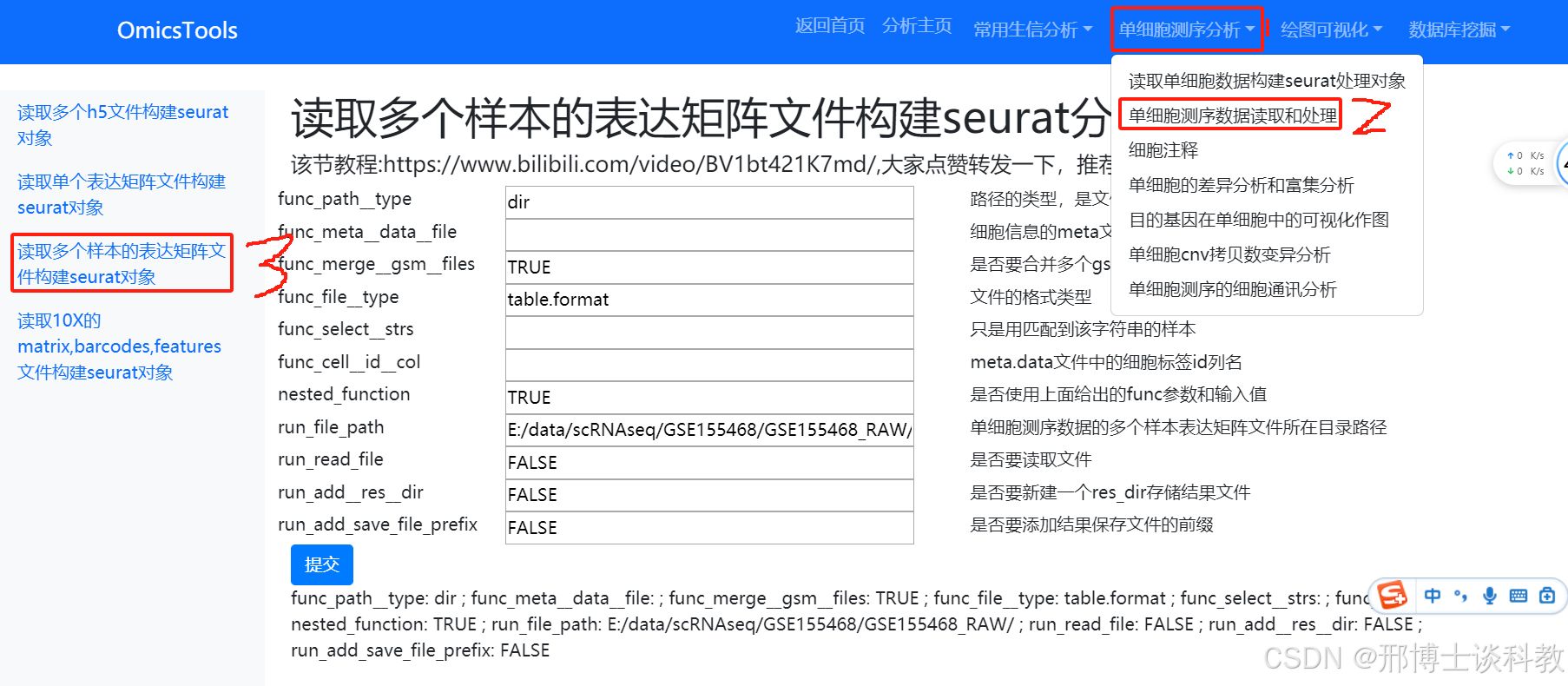
读取一个GSE155468多个样本的表达矩阵文件构建seurat分析对象
表达矩阵文件可以是CSV,TXT, TSV或者csv.gz,txt.gz, tsv.gz 等格式的表格文件
软件运行窗口
演示数据文件
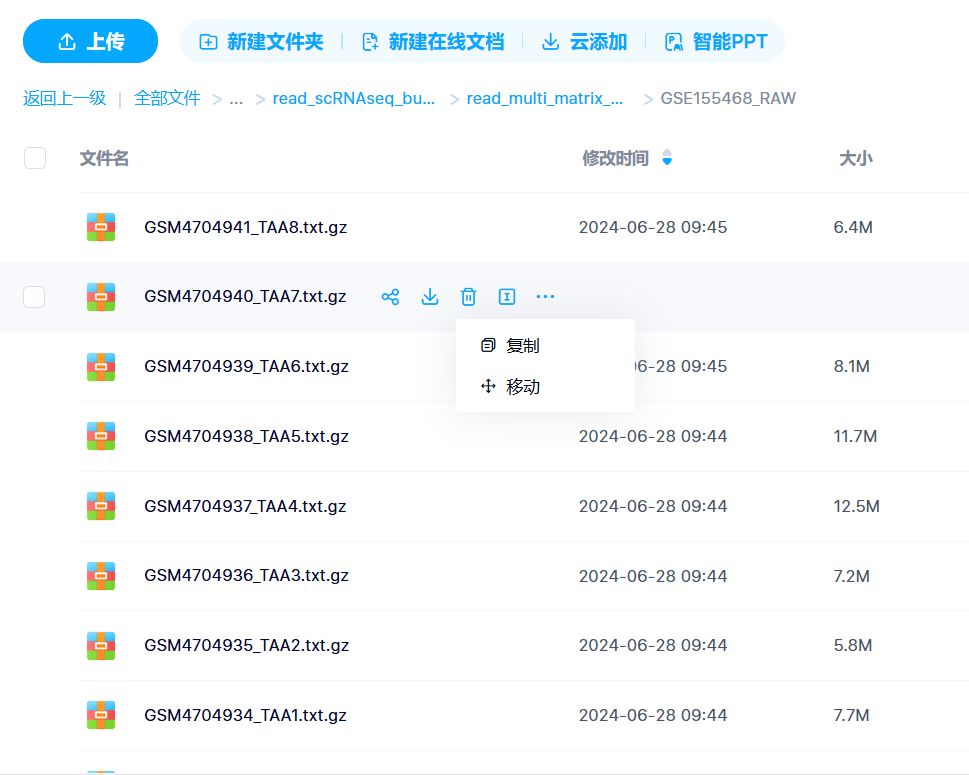
不管是GEO的单细胞数据,还是自己的单细胞数据或其他来源的数据的时候,大家在对文件名可能也需要做一定的修改,就是文件名开头在第1个下划线_之前的那个文件名的名字就要把它变成是唯一的,一般GEO的数据开头就用GSM编号开头,每个样本的gsm编号就是唯一的。
如果是自己的数据也要让他前面的那个名字变成唯一的,再用一个下划线_跟后面的文件名的部分进行分隔开,然后软件只会提取出第1个下划线前面的这个名字作为样本的 id和创建出这个样本的目录.
大家注意,如果你第1个下划线前面的这个名字跟其他样本不是唯一的,那么就会造成样本的一个重复,所以的话每个样本在第1个下划线之前的名字编号都要把它变成是唯一的跟其他的样本的地名字不重复的,这是在文件名字修改和读取的时候特别重要的注意事项。
软件运行结果文件得到构建好的seurat对象的rds文件
所有数据集放在一起进行质控
0. 质控分析模块所在位置和分析教程
质控分析模块所在位置
质控模块的详细分析教程指导
【腾讯文档】2.单细胞seurat的质控分析教程最新版
2.单细胞seurat的质控分析教程最新版
将第一步多个数据集单独构建出的Seurat分析对象的rds文件和上面构建的多个数据集的总的分组文件放在一个目录下
在这里run_file_path应该是一个目录,该目录下有要整合的多个单细胞数据集第一步构建出的Seurat分析对象的rds文件
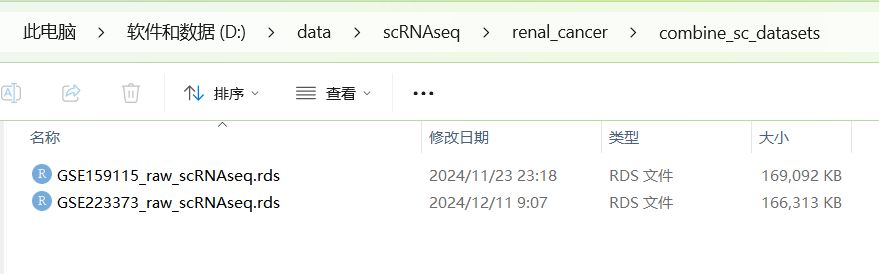
像这样,我们把这两个数据集第一步读取构建Seurat分析对象得到的rds文件都复制粘贴到了combine_sc_datasets这个文件夹下
构建出要整合的数据集的所有样本的样本编号sample.id列,批次效应组sub.group列,和生物学分组group.level列这样三列的样本注释文件
质控分析的时候func_group_file参数需要提供一个样本分组信息文件,这个样本分组信息文件对于多数据集整合的质控步骤和下一步的多数据集多样本整合和去除批次效应是非常关键的,一定要构建好正确的分组信息文件。
构建单细胞质控步骤的样本分组文件的教程
构建分组信息文件的详细方法可以查看我前面在质控步骤写的这篇教程:
【腾讯文档】2.单细胞seurat的质控分析教程最新版
2.单细胞seurat的质控分析教程最新版
这篇教程是构建单个数据集的分组信息的教程,我们可以按照这篇教程把每个数据集的分组信息构建出来,构建出列的每个数据集的分组文件的三列列名都是sample.id, sub.group和group.level这三列,可以很方便的按行把这几个数据集的分组文件整合在一个分组文件中。
同时在整合的含有多个数据集的样本编号的分组文件中,批次效应sub.group列应该是要整合的这几个数据集的GSE编号,比如我们这里是GSE159115和GSE223373这两个数据集编号,并跟每个数据集中的样本编号对应起来。
如果单个数据集内部也有不同批次信息的时候,比如GSE159115单个数据集里面还有date1,date2不同的检测时期,GSE223373单个数据集里面还有不同的测序平台plateform1,plateform2的时候,sub.group列对应的批次信息可以分得更细,我们可以把GSE数据集编号跟每个数据集内部的批次名称这两个名字拼接成一个名字,弄成更细分的批次效应分组,比如如果单个数据集内部都没有不同批次的时候,那就是GSE159115和GSE223373这两个批次效应分组,如果单个数据集内部还有不同的批次的时候,就把sub.group列弄成GSE159115_date1, GSE159115_date2, GSE223373_plateform1, GSE223373_plateform2这样更细分的批次效应分组,批次效应分组信息应该是跟样本编号都准确对应起来的。
构建出来的整合多个数据集的分组信息文件的内容应该我的这个csv这样:
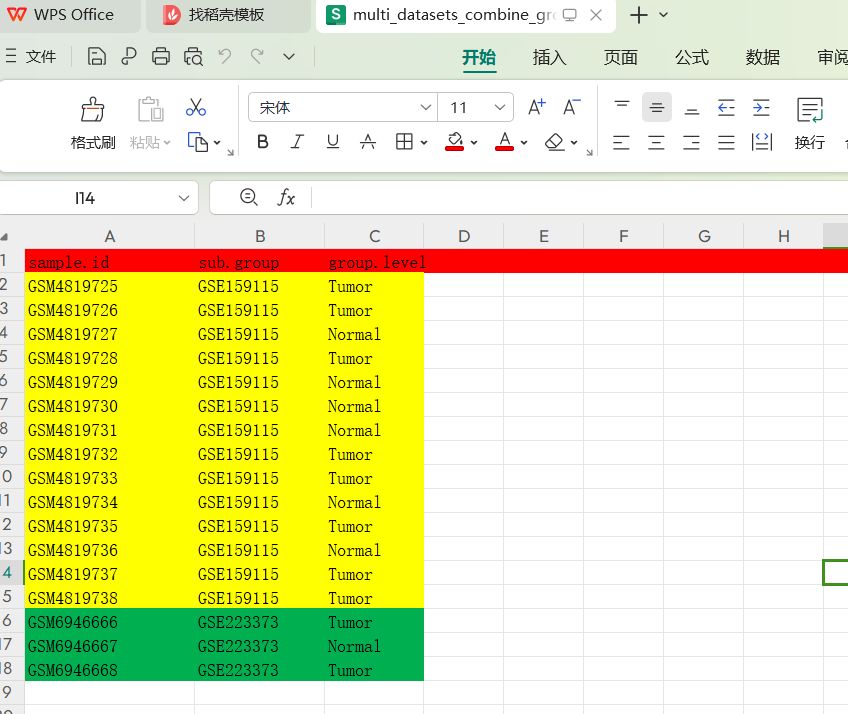
分组文件的里么是sample.id,sub.group和group.level三列含有多个数据集批次和分组信息的这个一个表格。
构建单细胞质控步骤的多数据集整合的样本分组文件的注意事项
- 我们按照教程找出这几个数据集的样本编号,并把这几个数据集的样本编号都整合在一个分组文件中,作为sample.id列,指导教程为:单细胞seurat的质控分析教程最新版,代做各领域生信分析和辅导 - 邢博士谈科教的文章 - 知乎https://zhuanlan.zhihu.com/p/11356697051
- 单个数据集内部都没有不同批次的时候,批次效应sub.group列就填要整合的这几个数据集的GSE编号,比如我们这里是GSE159115和GSE223373这两个数据集编号,并跟每个数据集中的样本编号对应起来。
- 如果单个数据集内部也有不同批次信息的时候,比如GSE159115单个数据集里面还有date1,date2不同的检测时期,GSE223373单个数据集里面还有不同的测序平台plateform1,plateform2的时候,sub.group列对应的批次信息可以分得更细,我们可以把GSE数据集编号跟每个数据集内部的批次名称这两个名字拼接成一个名字,弄成更细分的批次效应分组,比如如果单个数据集内部都没有不同批次的时候,那就是GSE159115和GSE223373这两个批次效应分组,如果单个数据集内部还有不同的批次的时候,就把sub.group列弄成GSE159115_date1, GSE159115_date2, GSE223373_plateform1, GSE223373_plateform2这样更细分的批次效应分组,批次效应分组信息应该是跟样本编号都准确对应起来的。
- 生物学分组group.level列就是这几个数据集的样本编号对应的分组信息,这些分组信息一般是根据样本的实际分组和我们的研究目的来定的。同时每个数据集的分组名称虽然意思可能相近,但是真实的分组名字可以能都是不同的,比如一个数据集里的分组名字叫Tumor, 一个数据集里的分组名字叫Cancer, 我们应该把不同数据集里相同分组的名字都整理成一模一样的分组名称,比如都整理成Tumor这个名字,如果到后面分析的时候会被识别成不同的分组。
进行多个数据集的质控分析
多数据集的质控分析细节
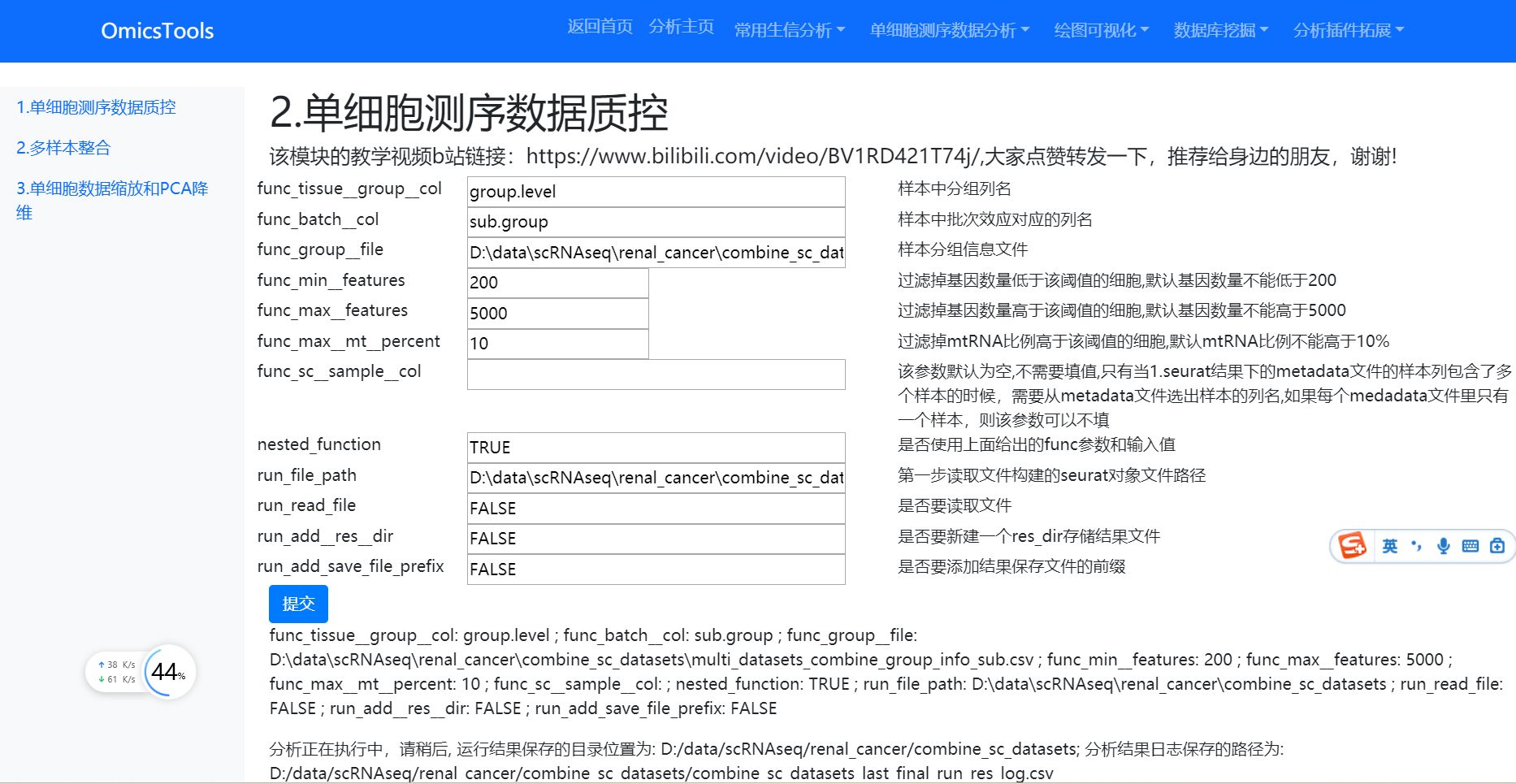
在做质控的时候,run_file_path提供的路径就是D:\data\scRNAseq\renal_cancer\combine_sc_datasets这个目录路径,而不是单个seurat rds文件的路径,到时候会读取该目录下所有数据集的rds文件进行质控处理。func_group_file填整合多个数据集样本批次和分组信息的分组文件路径。
多数据集的质控分析结果
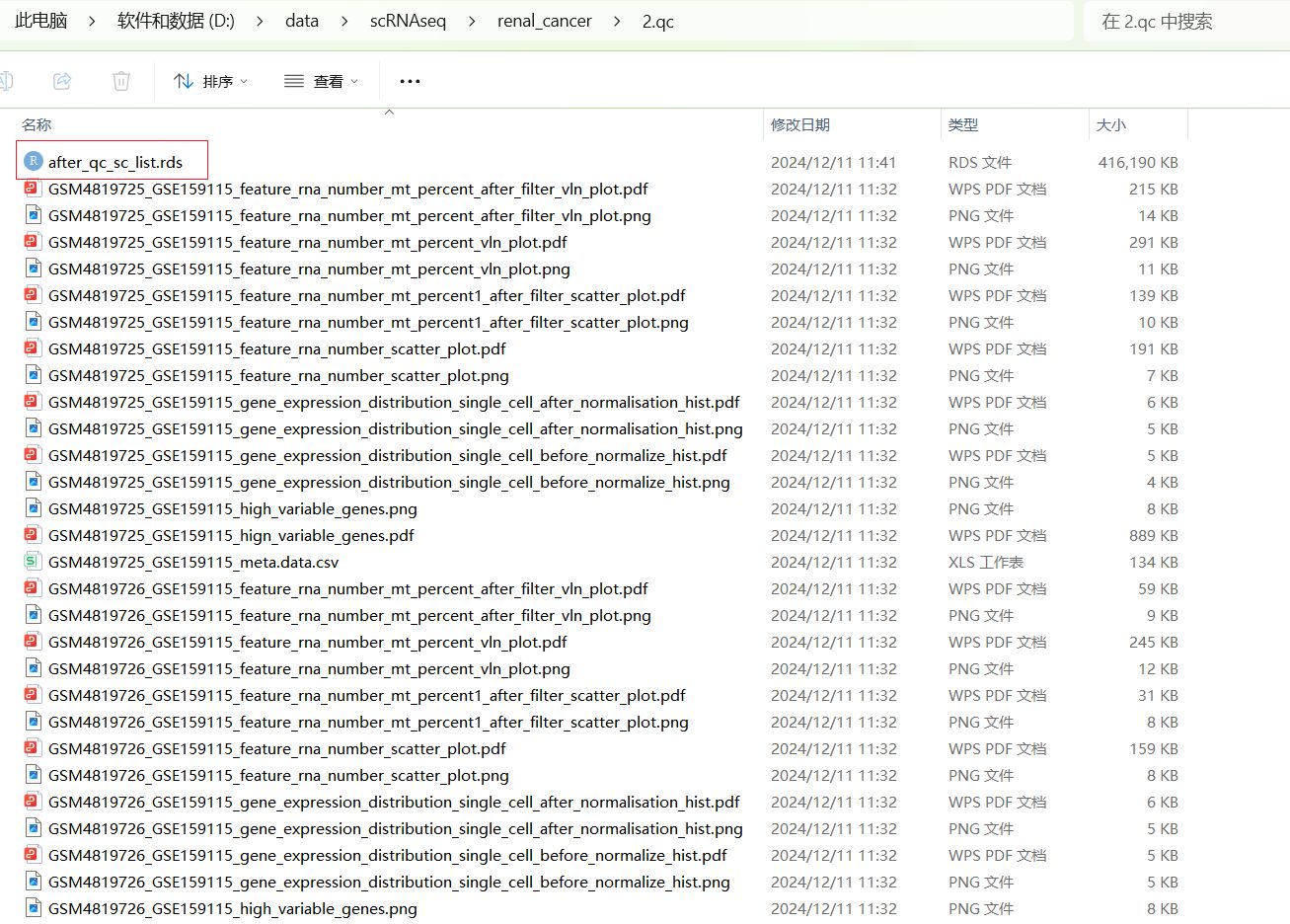
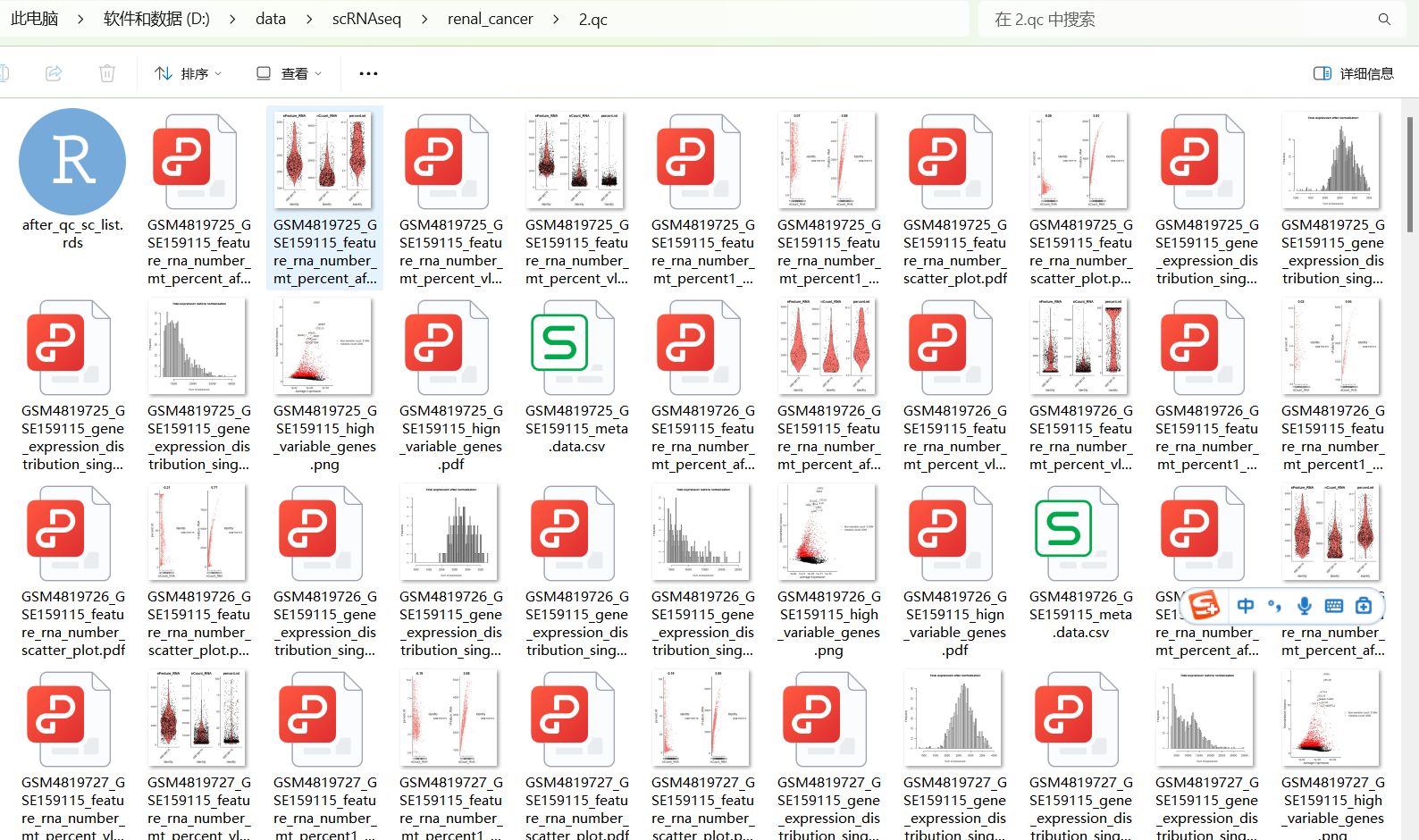
得到了多个数据集一起质控后的一个qc的rds文件和每个样本的质控前后的可视化质控图。
对多数据集一起质控后的rds文件结果进行多样本整合和去除批次效应
分析模块位置

在多数据集的多样本整合和去除批次效应的时候跟与单个数据集多样本整合所填的分析参数基本上是一样的,可以完全参考我的模块内的b站视频教程和单细胞的全流程分析文档教程来做。主要就是前面的多个数据集一起质控分析的时候填写对了就没问题。
这一步包括后续的分析都可以参考我的这篇零代码单细胞全流程分析图文文章教程:https://blog.csdn.net/qq_40073899/article/details/138866062
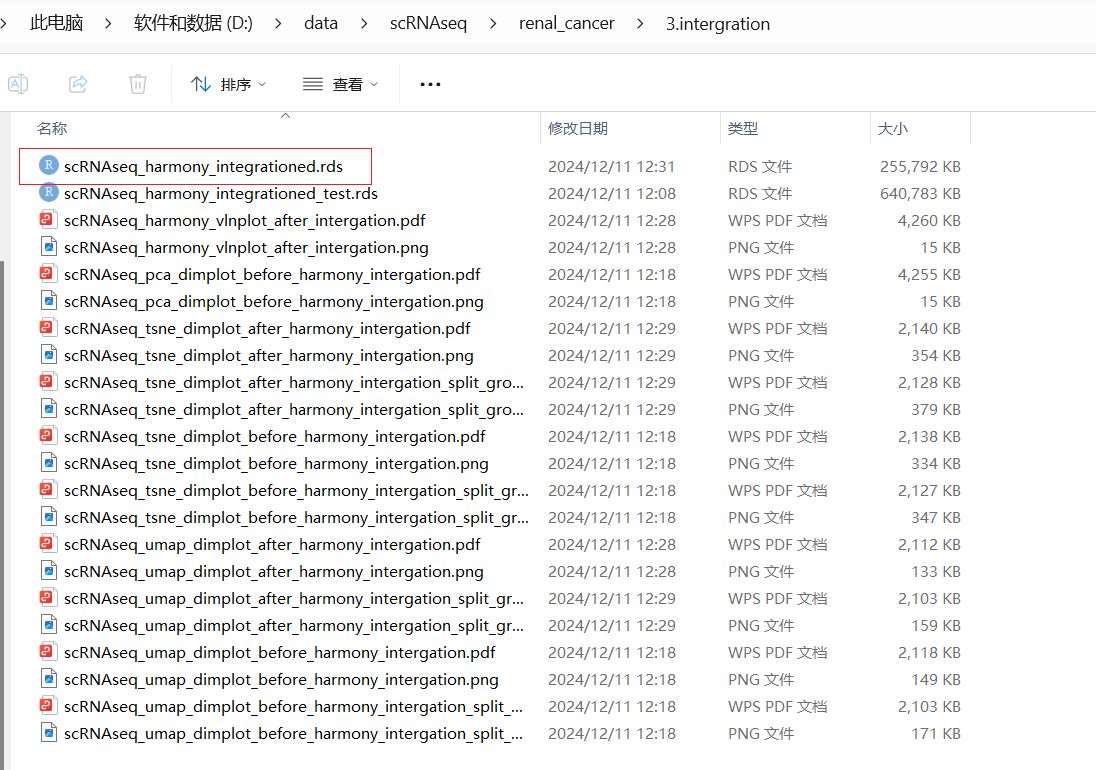
整合后的结果文件
OmicsTools软件和分析教程介绍
前言和简介
OmicsTools全能医学生物生信分析电脑软件简介
我开发了一款本地电脑无限使用的零代码生信数据分析作图神器一站式全流程电脑软件OmicsTools,旨在成为可以做各种医学生物生信领域科研数据分析作图的的全能科研软件,欢迎大家使用OmicsTools进行生物医学科研数据分析和作图,该软件件能让大家在不需要任何编程和代码编写的基础上,分析次数没有限制,可以无限使用,让您在自己电脑上快速进行大量的生信分析和加速大家的科研。
OmicsTools生信分析电脑软件可以做医学生物生信各个领域的科研数据分析和作图,并致力于成为医学生物生信领域的综合全能分析软件,一个软件帮助大家做医学生物生信领域的各种研究,快速出成果。

软件下载获取
我开发的本地电脑无限使用无限分析作图的生信零代码一键分析电脑软件神器OmicsTools 软件在github上的zihaoxingstudy1/OmicsTools(https://github.com/zihaoxingstudy1/OmicsTools)仓库中,也可以到我的生信交流q群群文件中下载,q群中的软件版本会更新一些,大家可以下载安装OmicsTools进行各种生信分析和可视化作图。
现在1群满员,会提示加2群,2群也可以下载到软件。
持续整理的各领域生信分析文档和答疑文档
持续整理更新的各领域生信分析教程文档--知乎版
生信分析全流程文档我都整理发布在了我的知乎专栏文章里,并汇总在了一起,并给大家提供了每篇文件教程的网址,大家都可以查看。
各领域生信分析全流程教程指导汇总版文档--持续更新 - 邢博士谈科教的文章 - 知乎
https://zhuanlan.zhihu.com/p/11754670943
这篇汇总的知乎文档有整理出各种分析教程对应的网址,大家都可以根据这边汇总的教程文章有针对性的学习和浏览对应领域的分析教程
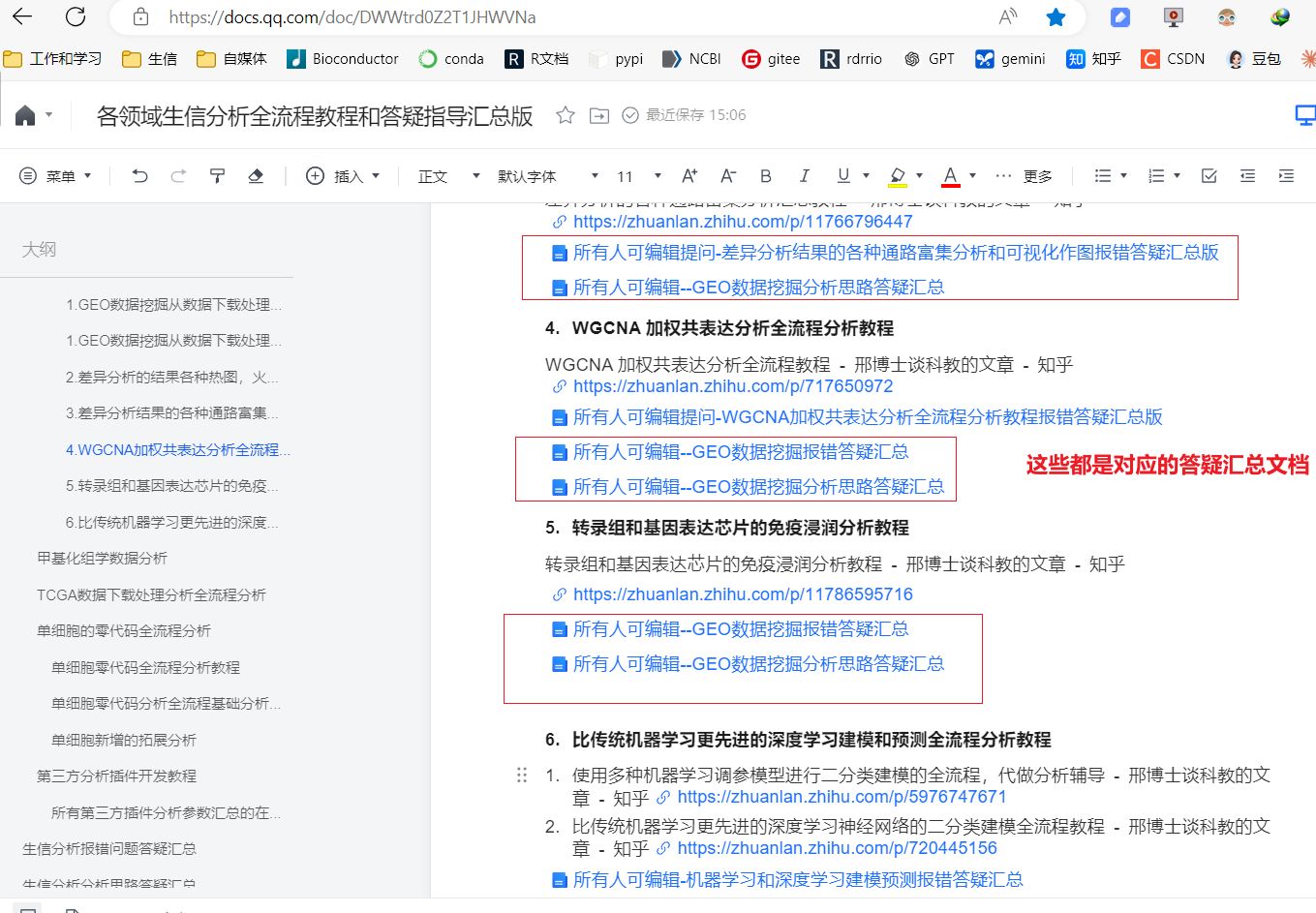
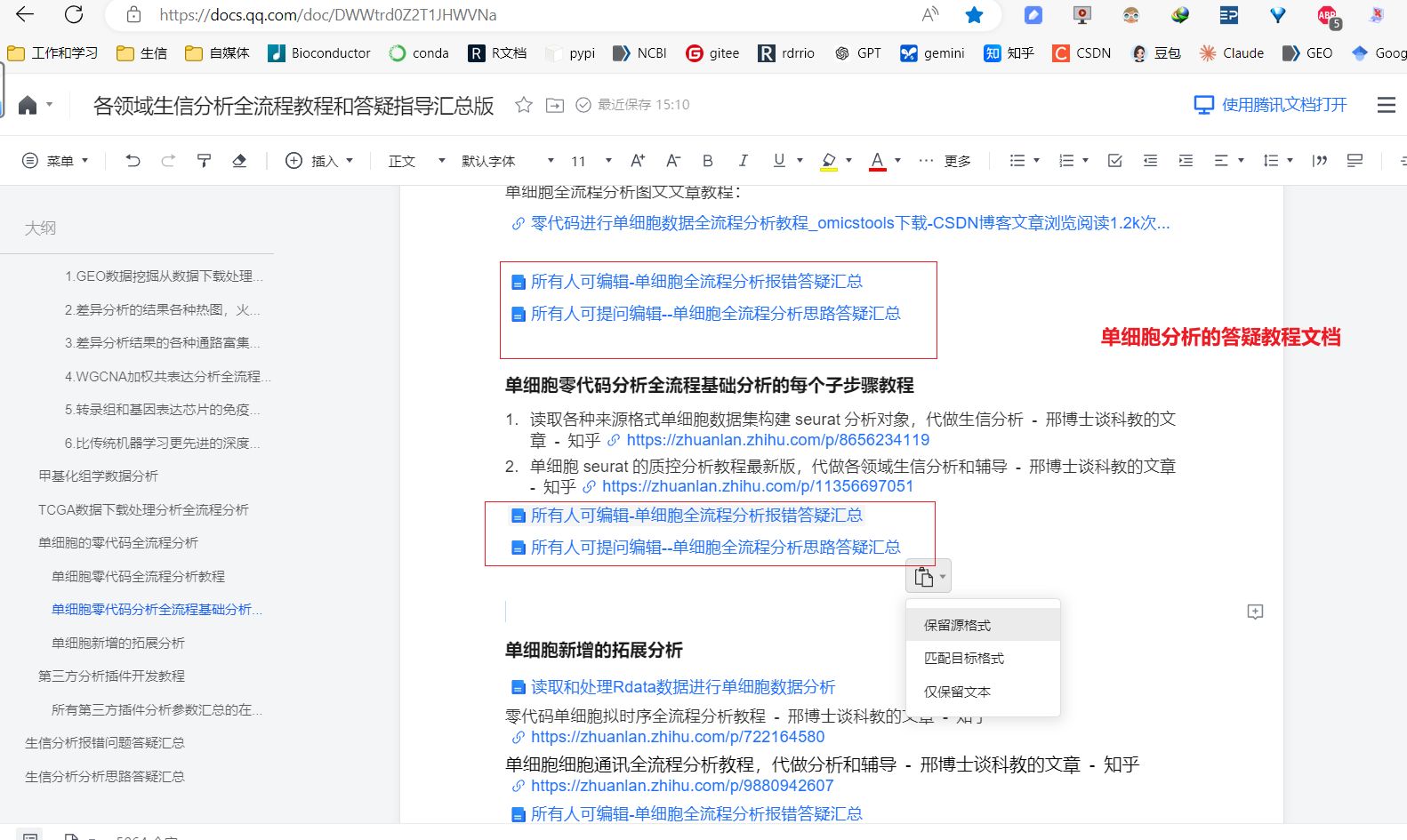
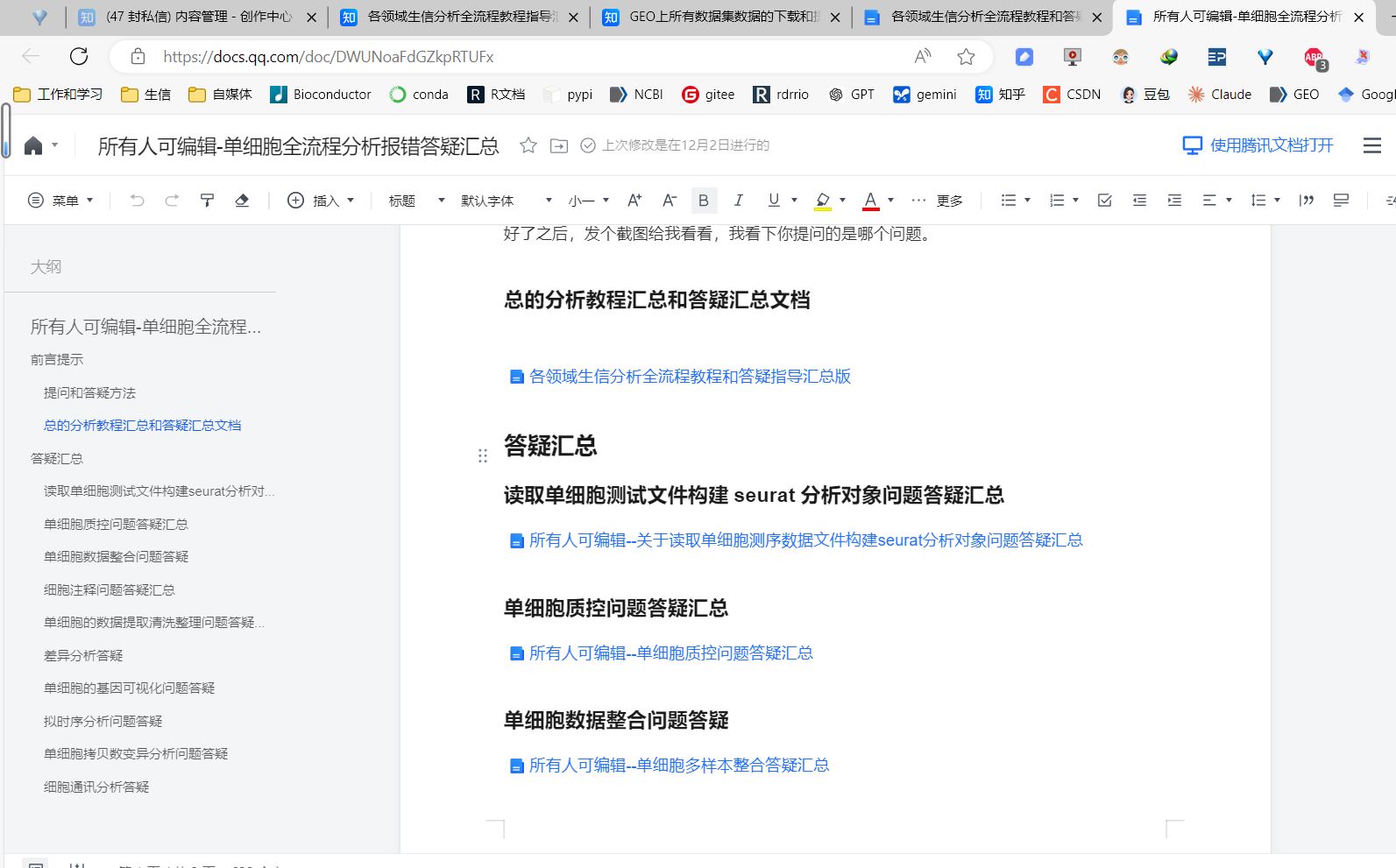
所有人可编辑提问我对各种问题跟答疑答疑的腾讯文档
【腾讯文档】各领域生信分析全流程教程和答疑指导汇总版
https://docs.qq.com/doc/DWWtrd0Z2T1JHWVNa
所有大家遇到的各种生信分析问题都在我的这篇腾讯文档对应的答疑文档中进行,腾讯文档的答疑文档支持所有人编辑和提问。
这篇总的腾讯文档是各领域生信分析答疑指导汇总文档的入口,以后所有的生信分析教程资料都在这个在线word文档中就能检索到,答疑汇总也能在这个word文档中检索到,都在这个在线word文档对应的提问答疑文档文件中提问,提问的问题在文档中用红色字体显示,我答疑在文档中用黑色字体显示,提问答个疑的文档和教程的文档所有人都可编辑。大家在腾讯文档里提问好了之后,现在答疑文档也是比较多的,发个截图和答疑文档的链接网址给我看看,这样我能快速定位到你提问的是哪个问题。
可以提供的科研服务清单

