【药物设计】三种分子碎片化方法介绍:Recap、BRICS与eMolFrag
三种分子碎片化方法介绍:RECAP、BRICS与eMolFrag
- 1. RECAP
- 1.1 简介
- 1.2 特点
- 1.3 方法
- 1.3.1 Concept of RECAP
- 1.3.2 断裂规则(Cleavage Rules)
- 1.3.3 断裂示例
- 1.4 代码实现
- 2. BRICS
- 2.1 BRICS做的事:
- 2.2 BRICS划分过程:
- 2.3 代码实现
- 2.3.1 BRICS拆解
- 2.3.2 BRICS合成
- 3. eMolFrag
- 3.1 方法
- 第一步:分子碎片化
- 第二步:减少片段冗余
- 3.2 总结
- 补充
- 参考资料
1. RECAP
Recap可以模仿实验室中的正向合成过程来进行逆向操作,对分子进行一系列的转换与分解,最终得到一组合理的分子片段。Recap可以对拆解过程进行追踪,并形成类似树的数据结构。原始分子记为根节点(root),被拆解的分子记为父节点(parent),拆解得到的分子记为子节点(children),某节点下的所有分支节点为子孙节点(all children),某节点上的所有父节点为祖先节点(ultimate parents),无法继续拆解分子记为叶子节点(leaves)。所有英文翻译对应recap中的属性或方法名。
1.1 简介
- 组合化学:若用一个构建块的n个单元和另一个构建块的n个单元同时反应,就能产生n*n个化合物,效率非常高。
- 组合化学的重点:找到高质量的构建块。
- 解决方法:运用RECAP, 基于化学知识将分子划分成构建块,并存入库中。之后就可以直接从这个库中取构建块使用。
这篇论文中motif和fragment是类似的意思。building blocks是要构建的库中的元素,它包含motif/fragment。
1.2 特点
RECAP的主要做法是:通过分割活性分子来获得活性构建块。我们知道有一些常见化学反应会形成的键,所以在分割分子的时候,我们就分割这些键。
RECAP的特点:在11个预定义的键上分割。这11个预定义的键保证了分出来的碎片是适合组合的,是适用于组合化学的。
1.3 方法
1.3.1 Concept of RECAP

- 1:收集一系列对特定目标有活性的结构。
- 2:使用RECAP进行碎片化——只保留最终碎片而不保留中间过程。
- 3:对碎片进行分析。包括该碎片在原始活性结构中出现频率的分析和集群分析(将类似的片段组合到一个群中)
- 4:碎片分析好后合并成为构建块,计入库中。
1.3.2 断裂规则(Cleavage Rules)
所谓的断裂规则指的就是:
- 预定义了11种bond,遇见这种bond就切一刀。
- 如果裂解完的碎片只包含小的官能团(氢、甲基、乙基、丙基和丁基),则这个碎片不裂解。目的:1.避免产生无用小片段。2.处于类药性考虑。
- 保留环结构
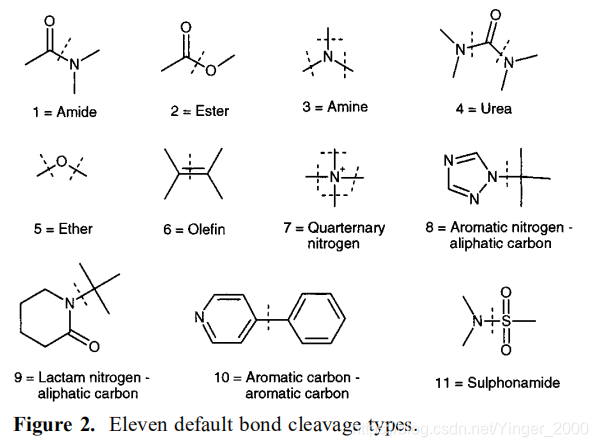
这些键选择了常见的化学反应会产生的键,之所以要这么选择,是为了使这些碎片易于合成。(基于这些碎片易于合成更大单位的构建块,另外,用户也可以自定义可切割的键。)
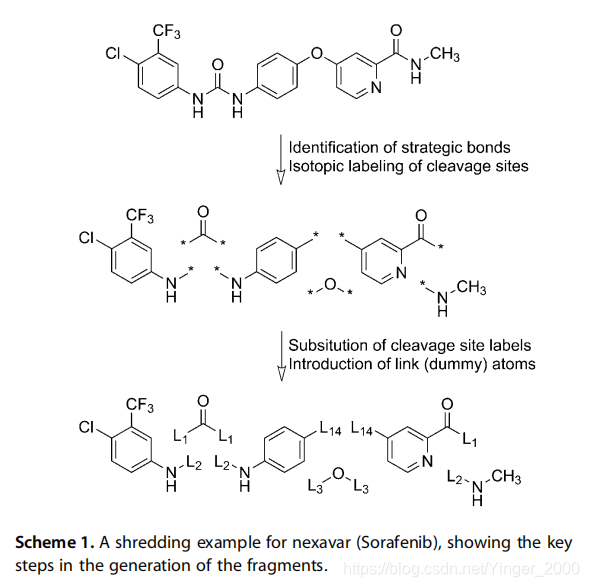
1.3.3 断裂示例
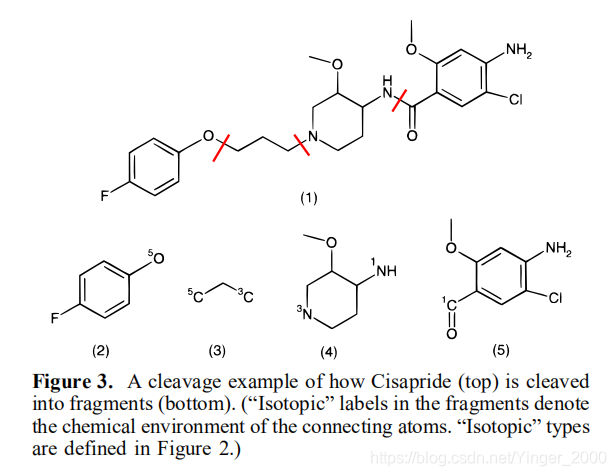
上图中一个分子有3条可断的键,形成4个碎片。碎片上有标记。文献中这个标记命名为“同位素标记”(Isotopic labels),但实际上它就是指:这条可断的键在11种可断键中的序号。作者认为,这个标记表示了裂解点(/碎片)所处的原子环境(/化学环境)。
化学环境的意义:
- 用于综合规划。在合成的时候可以依据这类信息选取部分碎片。
- 可以得到一个碎片有几个裂解点,这个信息也是很重要的。据此可以将碎片划分成terminal monomer(一个裂解点)和 core template(多个裂解点)。上图中(2)(5)属于terminal monomer,(3)(4)属于core template
1.4 代码实现
RDkit封装了RECAP的过程。可以用Chem.Recap.RecapDecompose()将分子碎片化,输入为mol类型,输出为RecapHierarchyNode类型。这个类型可以理解为:分子树。
from rdkit import rdBase, Chem
from rdkit.Chem import AllChem, Draw
from rdkit.Chem import Recap
import pubchempy as pcpcelecoxib = pcp.get_compounds('celecoxib', 'name')
celecoxib = celecoxib[0]
sm = celecoxib.canonical_smiles #转换成smiles
celecoxib = Chem.MolFromSmiles(sm) #转换成mol
hierarch = Chem.Recap.RecapDecompose(celecoxib) #碎片化
print(type(hierarch)) #碎片化后是一个特殊的数据类型可以从节点对象通过函数获取其叶子节点、子孙节点,得到的都是字典,键为节点的smiles,值为节点对象。
- 叶子节点函数:hierarch.GetLeaves()
- 子孙节点函数:hierarch.GetAllChildren()
- 祖先节点函数,返回列表:getUltimateParents()
以children为例,得到的字典的键是【一个分子二分后所有可能的碎片】的字符串表示,值是其RecapHierarchyNode类型。
RecapHierarchyNode类型还有mol方法和smiles方法,它可以把RecapHierarchyNode类型转换成mol形式和smiles形式。下面的第一行代码把一个分子树分了一次,得到的first_gen为所有可能的碎片的列表(mol形式)。
first_gen = [node.mol for node in hierarch.children.values()]
print(first_gen)
node_type = [node for node in hierarch.children.values()]
print(node_type)可以看到,first_gen枚举了所有二分后的可能性。
Draw.MolsToGridImage(first_gen, molsPerRow=4, legends=[Chem.MolToSmiles(m) for m in first_gen])这一行在PyCharm中运行显示不出来,但是可以在jupyter notebook中看到。这里应该有3条可断边,所以它断成了2*2=4种。所谓的可断边是确定的,就是11种可能性。
下面这段代码递归生成一棵分子-碎片树,描述了一个分子是怎样被一步步断开的(的所有可能性):
def get_leaves(recap_decomp, n=1):for child in recap_decomp.children.values():print('\t'*n+child.smiles)if child.children: ##さらなるフラグメント化のチェックget_leaves(child, n=n+1)def get_recap_tree(mol):recap = Chem.Recap.RecapDecompose(mol)print(Chem.MolToSmiles(mol))get_leaves(recap)get_recap_tree(celecoxib)除此之外,RecapHierarchyNode类型还有以下一些方法:
hierarch.GetLeaves():输出一个字典,键是所有叶子节点(即最终产生的最简单的化合物)的字符串表示;值是它们的RecapHierarchyNode类型。
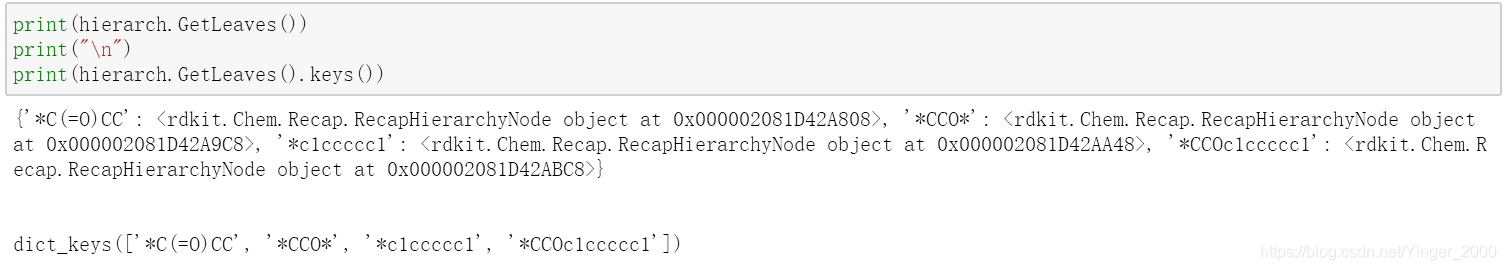
hierarch.GetAllChildren():输出一个字典,是所有的非根节点,其他与hierarch.GetLeaves()一致。
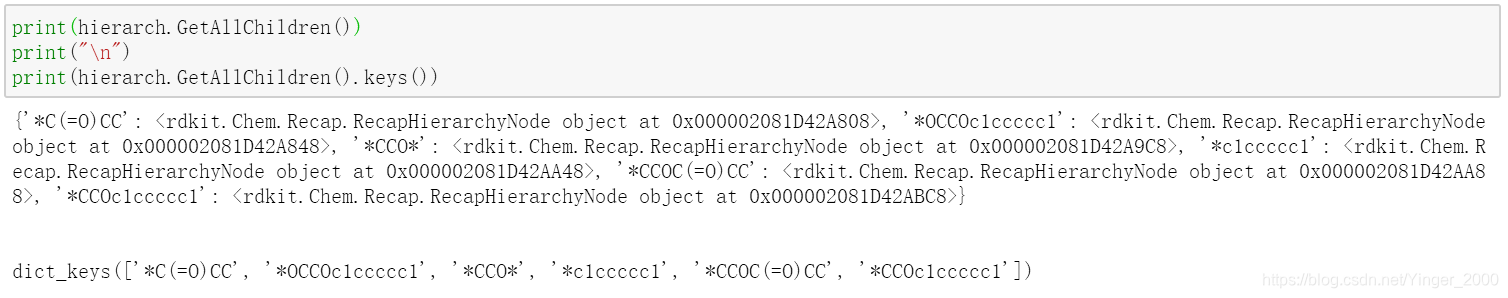
hierarch.getUltimateParents():返回一个只有一个元素的列表,这个就是根节点的RecapHierarchyNode类型。

2. BRICS
Rdkit中还有一种BRICS算法,同样可以用于拆解分子。与recap不同,该算法是根据键是否能够合成来进行拆解。该方法返回一个经过去重的列表,在原子上的序号对应一种特定的反应类型。
2.1 BRICS做的事:
- 给出了一套更好的碎片化的rules
- 给出一套重组模体(motif)的rules,以形成碎片空间。——正如前文所说,碎片空间除了碎片外,还需要重组模体/碎片的规则。
- 获得了一个高质量、高性能、公开可用的碎片集。
意义:更好的碎片集、更高性能的碎片空间。
特点:同时切所有可断的键,以避免产生冗余碎片。这与RECAP是一致的。
2.2 BRICS划分过程:
- RECAP直接在划分后注上“同位素标签”即可断键的id。但BRICS划分后先注上“同位素标签”,再用link atoms取代这些同位素标签——但这些link atoms看起来正是可断键的id。(在eMolFrag中解释,所谓的link )
- 每个键断裂之后会产生两个断裂点,对RECAP来说两个断裂点打的同位素标签对应的可断键id是相同的;而BRICS是不同的。
当然还有一些简单的事情,例如它也会丢弃/不划分很细枝末节的碎片。
下图是16种片段。只列出了断裂点的化学环境,由于R基的多样性,碎片也具有多样性。L1L6&L8L4出现了两次,它们的区分为:是否“appeared as cyclic or acyclic substituents or linkers”。
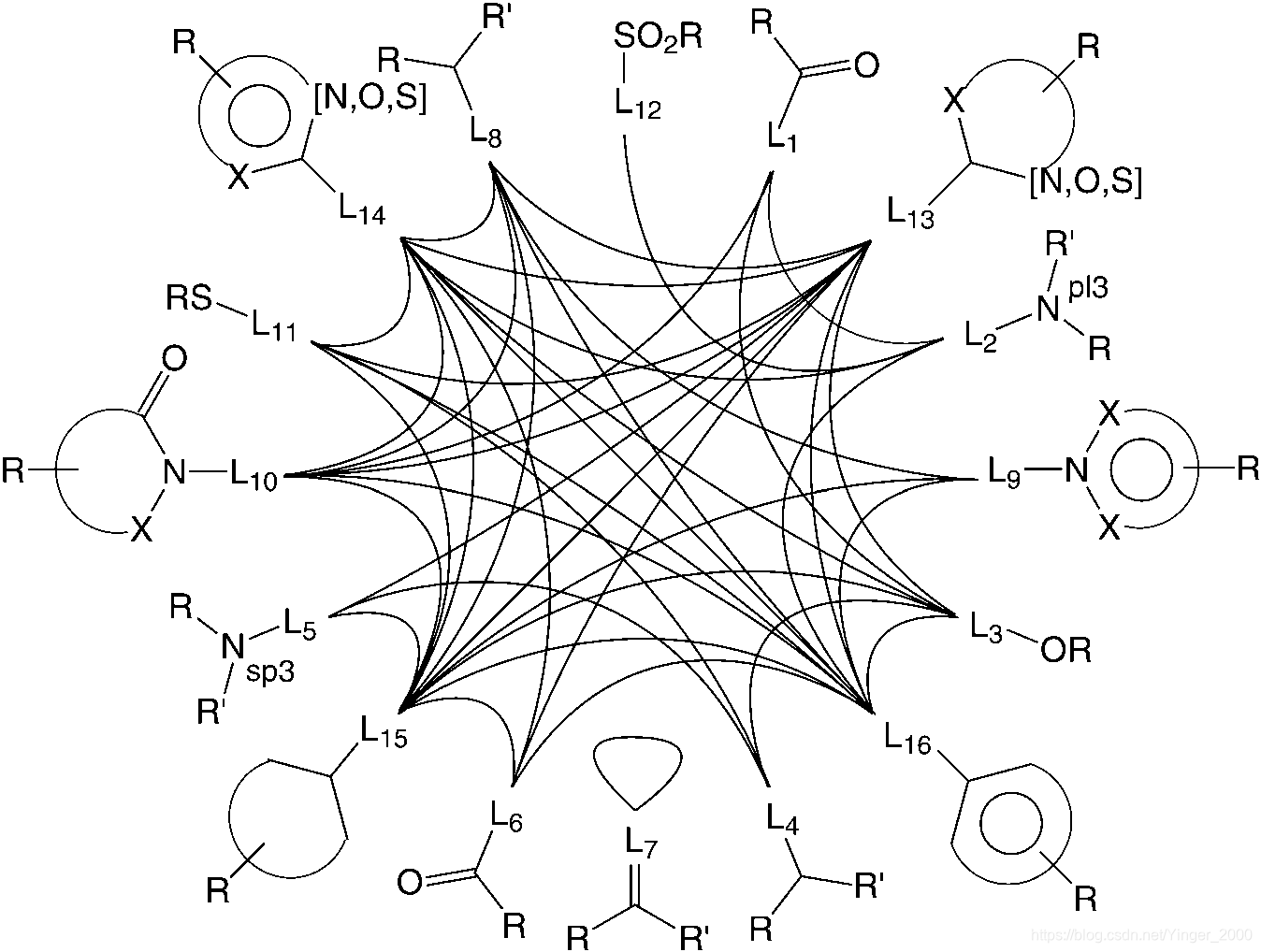
这里的16个rules和RECAP的11个rules形式上不同,RECAP列出的是可断键,而BRICS列出的是碎片结构。BRICS同样实现了用户自定义功能。
2.3 代码实现
2.3.1 BRICS拆解
由BRICS分解后的mol不再是一个用于表示分子树的新类型了,而是一个“一步到位的划分好的无冗余的碎片字符串”组成的列表
# BRICS算法拆解分子:BRICS.BRICSDecompose(m)
>>> from rdkit.Chem import BRICS
>>> m = Chem.MolFromSmiles('c1ccccc1OCCOC(=O)CC')
>>> BRICS.BRICSDecompose(m)
{'[1*]C(=O)CC', '[16*]c1ccccc1', '[3*]O[3*]', '[4*]CC[4*]'}# 对一组分子生成片段
>>> import os
>>> from rdkit.Chem import RDConfig
>>> cdk_file = os.path.join(RDConfig.RDDocsDir, 'Book\data\cdk2.sdf')
>>> cdk2mols = Chem.SDMolSupplier(cdk_file)
>>> allfrags = set()
>>> for m in cdk2mols:
>>> pieces = BRICS.BRICSDecompose(m)
>>> allfrags.update(pieces)
>>> print(len(allfrags))
90
>>> print(sorted(allfrags)[:3])
['NS(=O)(=O)c1ccc(N/N=C2\\C(=O)Nc3ccc(Br)cc32)cc1', '[1*]C(=O)C(C)C', '[1*]C(=O)NN(C)C']2.3.2 BRICS合成
BRICS模块除了支持拆解分子,也可以对一组片段进行合成:BRICS.BRICSBuild()
返回一个生成器,可以通过next()来获取元素
生成的分子也是未经过核对的,可以对分子重新计算一下隐式化合价、环相关等属性,并做更新:UpdatePropertyCache()
>>> fragms = [Chem.MolFromSmiles(x) for x in sorted(allfrags)]
>>> ms = BRICS.BRICSBuild(fragms)
>>> prods = [next(ms) for x in range(4)]
>>> [prod.UpdatePropertyCache(strict=False) for prod in prods]
>>> Chem.Draw.MolsToGridImage(prods, molsPerRow=4, subImgSize=(200, 200))
可以通过设置随机种子或设置scrambleReagents参数来保证可重复性:
# 在BRICSBuild执行前设置随机种子
>>> import random
>>> random.seed(100)
>>> fragms = [Chem.MolFromSmiles(x) for x in sorted(allfrags)]
>>> ms = BRICS.BRICSBuild(fragms)
>>> prods = [next(ms) for x in range(4)]
>>> [prod.UpdatePropertyCache(strict=False) for prod in prods]
>>> Chem.Draw.MolsToGridImage(prods, molsPerRow=4, subImgSize=(200, 200))# 设置scrambleReagents参数
>>> fragms = [Chem.MolFromSmiles(x) for x in sorted(allfrags)]
>>> ms = BRICS.BRICSBuild(fragms, scrambleReagents=False)
>>> prods = [next(ms) for x in range(4)]
>>> [prod.UpdatePropertyCache(strict=False) for prod in prods]
>>> Chem.Draw.MolsToGridImage(prods, molsPerRow=4, subImgSize=(200, 200))
3. eMolFrag
这是一个软件,可以把分子分成bricks和linkers两种类型的碎片,它们都属于构建块(building blocks)。在基于碎片的药物设计中,如果能够得到"带有碎片的连接信息"的构建块,效果会很好。但是众多碎片化工具在提取构建块时,并不会储存碎片的连接信息,所以这篇文章提出eMolFrag,在提取构建块的同时储存了碎片的连接信息。
3.1 方法
eMolFrag 采用基于图的表示法,其中分子被视作由节点组成的集合,这些节点代表原子,并通过边连接,边对应于化学键。片段则是分子中的一个子结构,可能包含该分子的所有原子和部分化学键,或仅部分原子与化学键。根据其功能,片段可分为“bricks”和“linkers”两类。给定一组分子后,如图1所示,eMolFrag将分子划分成碎片集经过两个步骤:第一步称为“Part I”,负责生成初始片段集;第二步则名为“Part II”,旨在确保最终得到的片段集完全无重复。

图1. eMolFrag 流程图。第一部分:利用 BRICS 算法对输入分子进行裂解,生成一套完整的构建模块;第二部分:通过 kcombu 程序进行两两化学比对,去除碎片冗余。最终,系统会报告去除了冗余后的非重复性bricks与linkers集合,并附带整合的连接信息,以及已被移除的相似片段列表。
第一步:分子碎片化
在eMolFrag中,首先利用RDKit实现的BRICS算法,将一组分子分解为各自的组成片段。根据BRICS模型定义的16种化学环境,化学化合物被拆解成更大的基团,即“bricks”,这些bricks通过linkers相互连接;碎片化过程的伪代码详见支持信息中的算法S1。其中,一个linkers片段是指至少包含四个非氢原子的分子结构单元。随后,从分子中移除linkers,剩余的片段则被归类为linkers(具体参见支持信息中的算法S2)。断裂的化学键会被虚拟原子取代,这些虚拟原子仅作为被移除特定化学键上原子的占位符。为了便于提供经验性的连接模式,每块bricks都会存储与其相关联的化学键类型等完整信息。而linkers则携带不同的辅助连接信息——即这些片段仅标注了各位置上可能的最大键数。支持信息中还分别提供了bricks和linkers的示例(示例S1和S2)。我们发现,这种方法能够高效地构建出一系列新分子,其化学性质与母体化合物高度相似。
解读:这一步使用BRICS算法将分子划分成碎片。其中含有至少4个非氢原子的碎片被归类为bricks,移除所有bricks后剩下的是linkers.
bricks储存完整的信息。下图为某bricks储存的信息。额外信息包括:
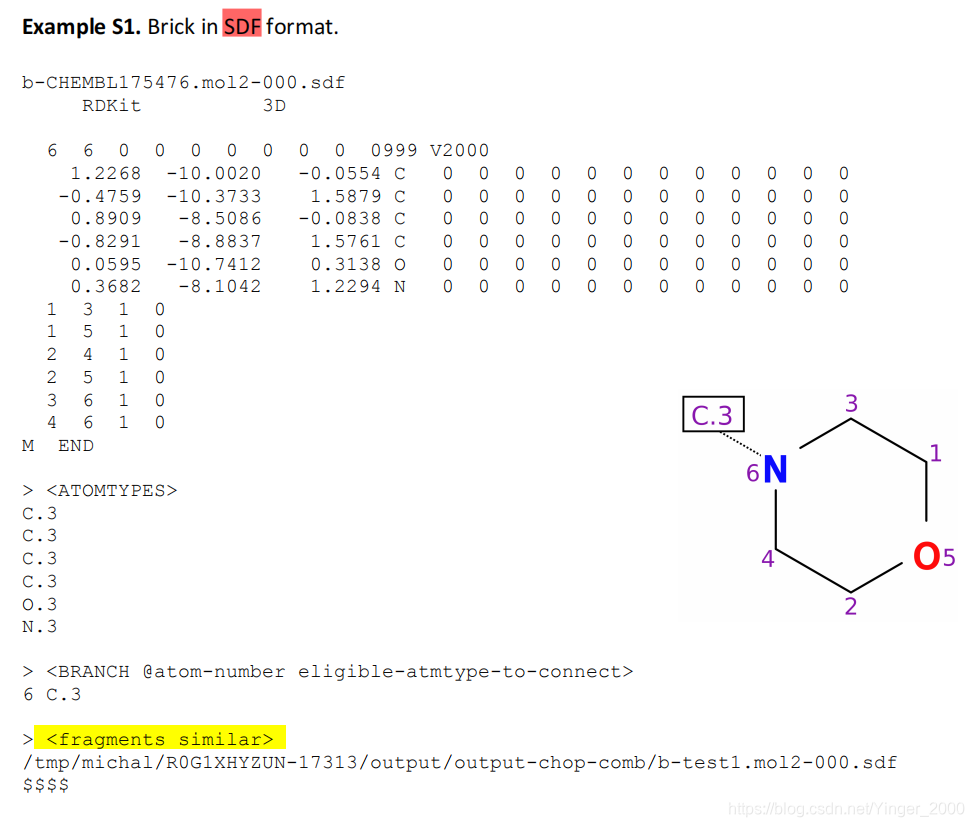
- ATOMTYPES:碎片中每个原子的类型
- BRANCH @atom-number eligible-atmtype-to-connect:所有的断裂键。每个断裂键是一个二维数组,第一维是碎片中的连着断裂键的原子的id,第二维是不在碎片中的、断裂键应连接的原子的类型。例如图中的6 C.3,指的是该碎片的第六个原子连着断裂键,断裂键的另一侧应该是一个类型为C.3的原子。
- fragments similar:被删除的类似片段。
下图为某linker储存的信息,额外信息只有:
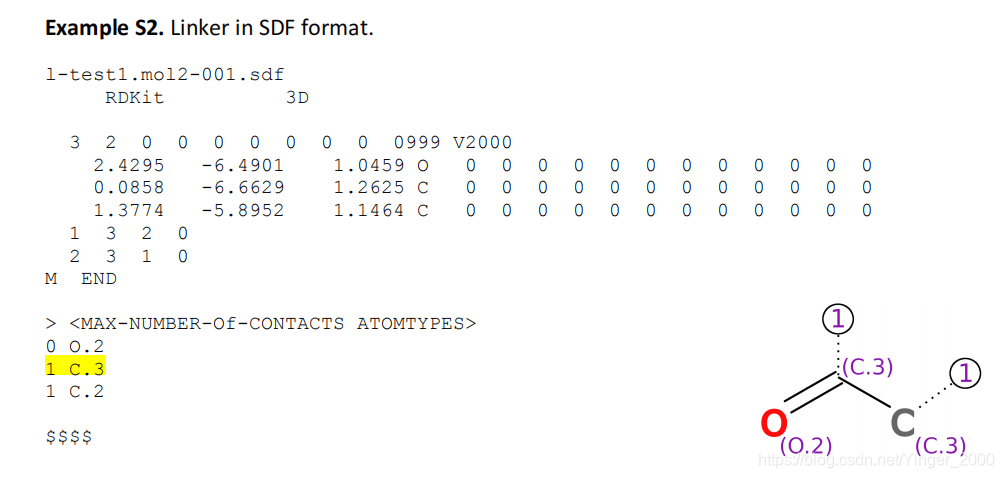
MAX-NUMBER-Of-CONTACTS ATOMTYPES:每个位置所能连接的最大键数和键另一端的原子类型,每个位置表示为一个二维数组。例如这个linker这栏信息的第二行为1 C.3, 表示这个linker的第二个位置最多可以连1个键,键的另一边连接的原子类型应为C.3。
第二步:减少片段冗余
由于有效碎片化程序的目标之一是将生成的碎片用于合成过程,因此最终碎片集合的基数至关重要。为此,eMolFrag尝试通过去除冗余来最小化bricks和linkers集合的大小,具体方法是在支持信息中提出的基于划分与筛分的去除方案(算法S3)。如果两个碎片的拓扑约束下最大共同子结构经由kcombu程序计算出的Tanimoto系数(TC)等于1.0,则认为这两个碎片等价。随后,利用kcombu提供的等价原子信息及其连接关系,将完全相同的碎片合并为单一、唯一的结构单元。
3.2 总结
eMolFrag 是一种快速且强大的工具,能够从小分子数据集中提取分子片段,这些片段被分类为“bricks”和“linkers”。随后,这些片段可用于构建目标导向的化合物库,以进行虚拟筛选。eMolFrag 的独特之处在于,它会存储所提取构建块之间的连接信息,从而帮助生成一系列化学上可行的新化合物。尽管 eMolFrag 经过优化,可与近期开发的分子合成算法 eSynth 协同工作,但它同样能与其他基于化学片段的化学生物信息学工具包无缝集成。目前,eMolFrag 已作为独立软件及Web服务器免费提供,用户可通过以下网址访问:www.brylinski.org/emolfrag 和https://github.com/liutairan/eMolFrag。
补充
一些文献对Recap的描述
In this context, the “fragment space” concept was introduced. In contrast to a fragment library, such a space consists not only of a set of fragments, but also of a set of rules that specifies how to recombine fragments by fusing the respective chemical motifs.
作者认为RECAP产生了“碎片空间”的概念。这个碎片空间和碎片库的区别是:它还包含了碎片融合的规则,即如何融合motifs以重组片段。
motif是什么?
motif是在许多蛋白质分子中,由几个具有二级结构的肽段在空间上相互接近、相互作用,所形成的折bai叠模样称为超二级结构,又称为模体。
BRICS考虑了每种断裂键的化学环境和周围的子结构。RECAP考虑了化学环境但没有考虑周围子结构
BRICS expands the bond type criteria used by RECAP from 11 to 16 taking into account the chemical environment of each bond type and the surrounding substructures.
参考资料
- 通过Recap和BRICS对分子拆解与合成
- A toolkit for fragmenting and analyzing molecular structures
- 分解以便积累:使用eMolFrag分解小分子,用于基于片段的药物设计
- 三种碎片化方法:RECAP, BRICS与eMolFrag
- Liu, Tairan, Misagh Naderi, Chris Alvin, Supratik Mukhopadhyay, and Michal Brylinski. “Break Down in Order To Build Up: Decomposing Small Molecules for Fragment-Based Drug Design with e MolFrag.” Journal of Chemical Information and Modeling 57, no. 4 (2017): 627-631.



