文献阅读 | iMetaMed | FigureYa:一个标准化可视化框架,用于增强生物医学数据解释和研究效率
文献介绍

文献题目: Fineya:一个标准化可视化框架,用于增强生物医学数据解释和研究效率
研究团队: FigureYa 项目组(https://github.com/ying-ge/FigureYa)
发表时间: 2025-09-15
发表期刊: iMetaMed
DOI: 10.1002/imm3.70005
摘要
生物医学研究数据可视化面临专业知识不足与方法论零散等多重挑战,严重制约研究效率与成果质量。FigureYa 是一个由 317 个模块化 R/Python 脚本构成的标准化可视化框架,而非独立软件或桌面应用程序。其覆盖表达谱分析、免疫分析、生存分析与单细胞数据可视化等关键领域,基于"替换数据即用"理念,显著降低技术门槛,使研究者无需深厚编程背景即可生成高质量图表。相较于普通在线 R 代码片段,FigureYa 提供由作者团队原创开发、经过严格验证且具有生物学上下文的可视化模块。每个脚本均包含版本匹配的环境配置、示例数据集与详细注释,在自动化程度、可复现性与科学专业性方面具有明显优势,从而为复杂生物医学数据提供标准化可视化解决方案。这一创新工具优化科研时间分配,促进跨学科协作,加速科学发现与临床转化,为生物医学研究提供强有力的数据可视化支撑。
前言
当代科学研究面临数据指数级增长带来的挑战。随着高通量测序、大规模临床试验和复杂计算模拟等技术的广泛应用,研究人员必须处理的数据量持续呈指数级增长。Dinov 的研究强调,现代医疗大数据不仅呈现传统的"4V"特征(数据量大、处理速度快、数据类型多样和数据准确性),还表现出更高的复杂性和异质性,这给数据可视化带来了前所未有的挑战。
医学研究人员在整合和可视化大规模多维数据方面面临重大挑战。在多组学研究中,需要同时分析基因组和转录组等多个数据源;然而现有工具往往仅限于单一数据类型。专业的可视化工具(如 R 语言的 ggplot2)需要高级编程技能,而 Excel 等用户友好型工具功能有限。即使是简单的可视化任务也经常需要大量编码工作,这对没有计算背景的研究人员造成了障碍。这些工具局限性显著降低了研究效率。研究人员常常难以平衡数据分析与技术学习,且不恰当的可视化可能误导结论并损害研究的整体影响力。另一个关键问题是可复现性。许多在线可视化工具缺乏透明代码,频繁的版本更新导致了严重的可复现性问题。当研究人员尝试复现先前的可视化结果时,常常发现工具更新导致无法生成完全相同的图形。相比之下,FigureYa 的代码完全透明,用户可以根据需要进行修改和调整,从而确保研究结果的可复现性。
科学数据可视化面临三大核心挑战:技术壁垒、时间效率与视觉质量。大多数研究人员缺乏数据可视化方面的系统培训,对视觉感知原理和色彩理论等基本概念的了解有限,难以处理复杂数据集。开发复杂可视化图表耗时冗长,与研究时限的紧迫性相冲突——特别是在高维数据分析中,传统静态可视化工作流往往难以满足快速数据探索的需求。使用传统工具时研究人员常需耗费大量时间调整参数,而 FigureYa 等新型工具通过预配置代码简化了这一过程。研究人员还难以生成符合出版标准的高质量可视化图表。高质量生物医学可视化必须兼顾数据准确性与视觉清晰度等多重因素。这些挑战共同限制了研究人员挖掘数据价值的能力,延长研究周期,并削弱研究成果的整体影响力。
数据可视化在科学研究中扮演三个关键角色:首先,作为强大的发现工具,帮助研究人员识别复杂数据中的模式与异常。例如 Pearce 团队开发的基因组浏览器显著加速了临床研究进展。其次,高质量图形能同时提高科学论文的接收率和引用率。交互式可视化正在改变科学出版实践并提升研究透明度。最后,随着科学研究日益跨学科化和开放科学的持续推进,从生物医学到社会科学等多个领域对专业化可视化的需求正在快速增长。主流期刊据此提高了可视化标准。这些发展表明,数据可视化已成为科学研究不可或缺的核心组成部分。
FigureYa 并非独立的软件平台,而是基于 R 语言的模块化可视化框架。它创新性地提出"即用型可视化代码与示例数据整合"理念,通过精心设计的预配置代码模板及配套示例数据集,有效消除科学可视化的技术壁垒。该框架基于三大核心原则构建:简洁性——通过直观界面与详细文档最大限度降低学习成本,使技术背景有限的研究人员能快速掌握工具使用;高效性——通过自动化工作流和预配置模板显著缩短可视化开发周期;专业性——融合视觉感知原理与图形设计最佳实践,确保输出成果符合学术出版标准。FigureYa 专门针对编程经验有限的研究人员,提供带有完整注释、经过验证且直接适用于常见生物医学场景的高质量实战驱动型 R 脚本,适用于面临时间压力的研究团队和追求高质量可视化成果的学者。其应用场景覆盖研究过程的关键阶段,包括论文图件制备、会议报告呈现和探索性数据分析。用户只需将数据格式调整为与示例相似,即可生成专业级可视化图表。每个脚本均包含双语注释、使用指南、示例数据集和预渲染输出结果。GitHub 仓库采用类似网页界面的可导航结构组织,使编程基础有限的用户能通过简单步骤完成操作。这一框架从根本解决了科学可视化长期面临的技术门槛高、效率低下和质量参差不齐等难题,为推动科学可视化的标准化与专业化提供了实用解决方案。
研究结果
1. FigureYa 资源库内容和结构的概述
1.1 功能分类分布
FigureYa 代码资源包包含 317 个高度专业化的可视化脚本,覆盖生物医学研究的主要数据类型和分析场景。按研究类型划分,基因表达谱分析(23.6%)、免疫表型分析(12.4%)、生存分析(11.5%)和单细胞分析(10.9%)占比最高。就分析类型而言,富集分析(14.3%)、差异分析(12.1%)和相关性分析(11.8%)使用频率最高。Figure 1 展示了可视化工具在研究类型、分析方法和输出格式三个维度的分布情况。输出类型方面,热图(22.7%)、折线图(15.8%)和散点图(10.2%)是最常用的可视化形式。这一分布反映了当前生物医学研究的关键趋势:多组学数据整合日益成为研究重点;单细胞技术的兴起推动了对相关可视化工具的需求;临床研究对预测模型和生存分析持续保持高度关注。值得注意的是,机器学习相关可视化工具占总量的 7.1%,凸显出人工智能技术正快速融入生物医学研究领域。
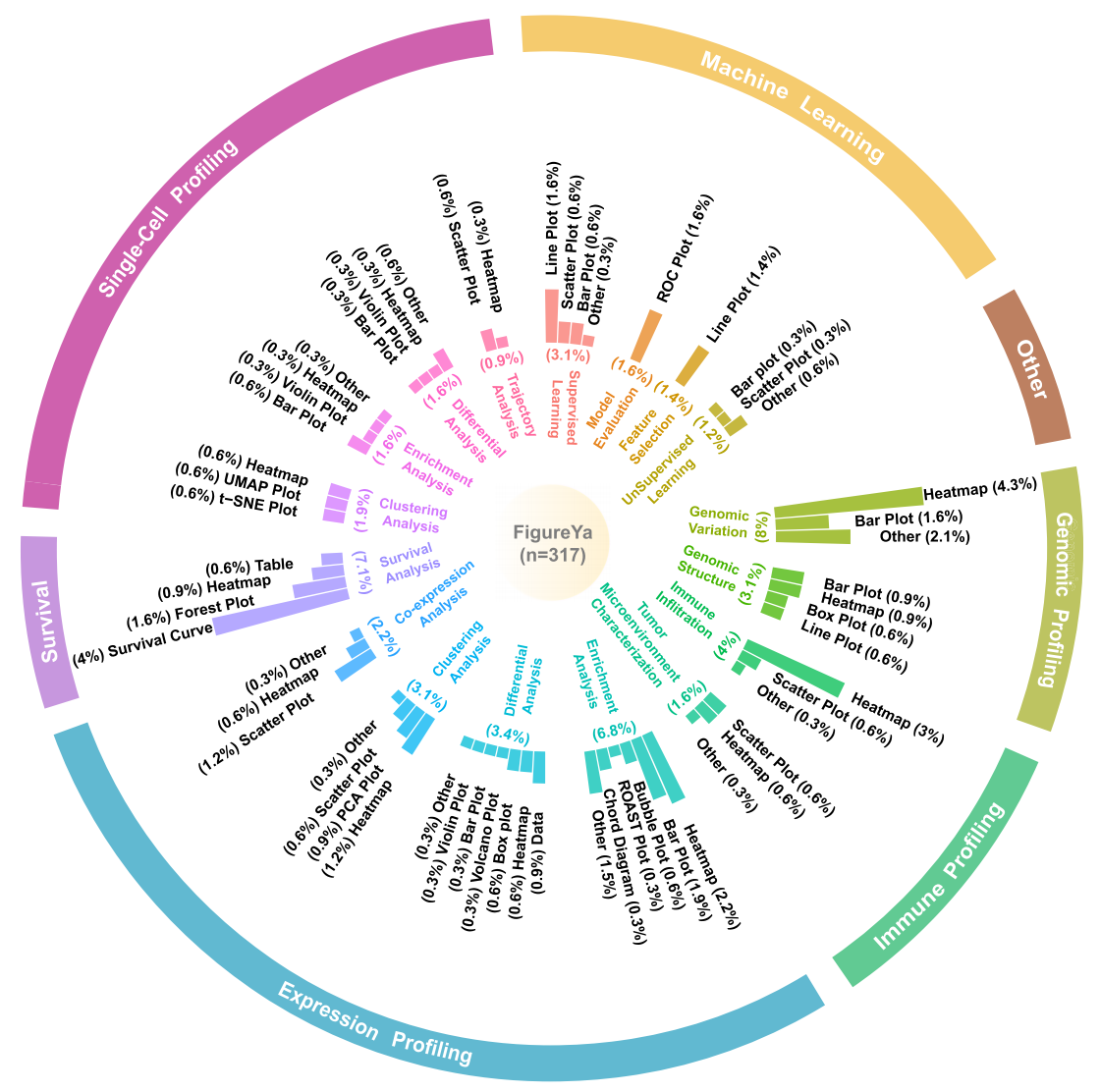
Figure 1. 在 FigureYa 资源软件包中的可视化工具的分布
该饼图展示了可视化工具在三个维度上的分布情况:研究类型(外环)、分析方法(中环)和输出格式(内环)。主要研究类别包括单细胞分析(粉色)、机器学习(黄色)、基因组分析(绿色)、免疫学分析(浅蓝色)、表达谱分析(深蓝色)和生存分析(紫色)。每个扇区显示各类别工具所占比例。最常见的可视化形式包括热图(22.7%)、折线图(15.8%)和散点图(10.2%)。这种多维分类系统使研究人员能够根据具体研究目标和数据特征快速定位合适的工具。
1.2 覆盖分析
FigureYa 代码资源包的覆盖范围体现了"从基础到前沿、从单一到整合"的设计理念。在基础统计可视化方面,该资源包包含标准描述性图表 —— 如 FigureYa12box 和 FigureYa59volcano —— 这些图表经过优化,可自动处理显著性标记和多组比较。针对领域特异性可视化,资源包涵盖了基因组学、转录组学和临床研究等主要生物医学研究领域的关键图表类型。对于新兴技术,FigureYa 提供了单细胞分析、空间转录组学(如FigureYa239ST_PDAC)和多组学整合(如FigureYa258SNF)的端到端可视化工具。此外,该资源包还包含药物敏感性分析、免疫微环境谱分析和表观遗传学研究的专用工具。这种全面的覆盖范围确保研究人员无论处于哪个研究阶段或学科焦点,都能找到合适的可视化工具。
1.3 代码互连
FigureYa 代码资源包构建了一个互联互通、可组合的可视化生态系统。这种连通性体现在三个层面:数据流整合、分析工作流联动和组合式可视化集成。在数据流层面,资源包包含数据预处理与格式转换工具 —— 如 FigureYa21TCGA2table 和 FigureYa22FPKM2TPM —— 形成完整的数据流水线。在分析工作流层面,多个脚本可组合构建完整分析流程。例如在转录组差异表达分析中,可依次使用 FigureYa119Multiclasslimma、FigureYa9heatmap、FigureYa59volcano 和 FigureYa60GSEA_clusterProfiler,形成从差异检验到功能阐释的完整分析链路。在可视化集成层面,多个可视化脚本可组合生成复杂的多图版综合图表,如 Figure 2 所示。例如 FigureYa69cancerSubtype 将聚类热图、生存分析和临床相关性分析整合到统一视觉框架中。
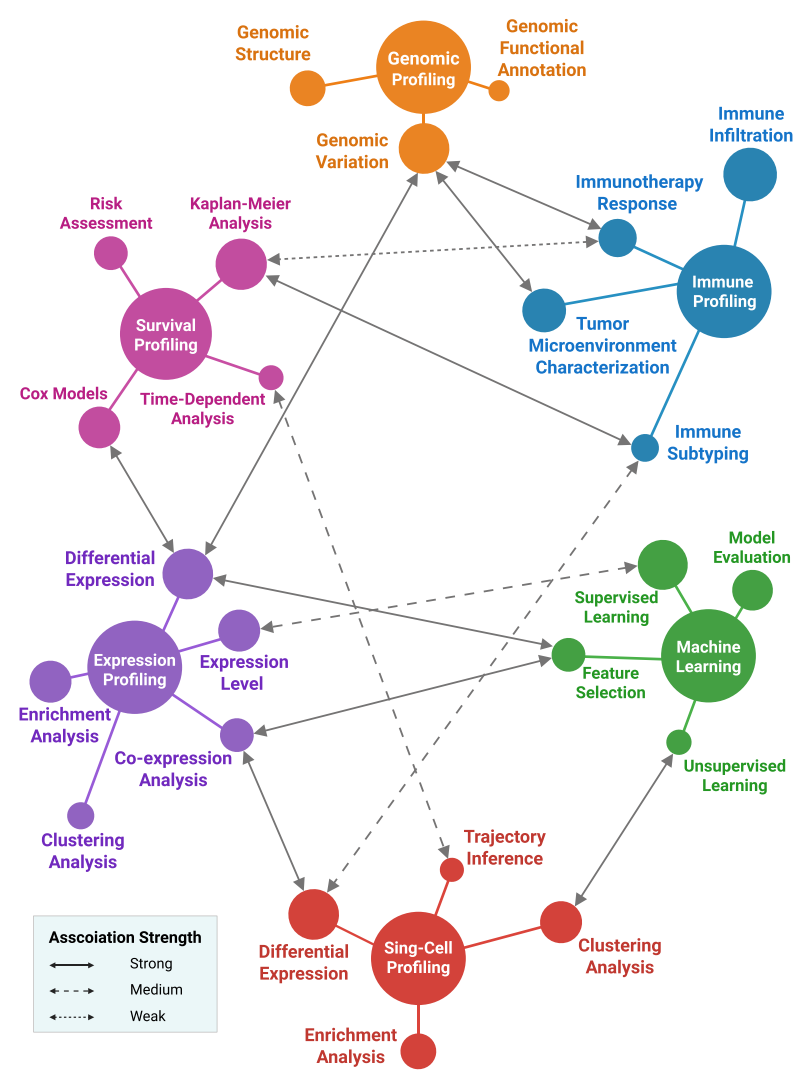
Figure 2. FigureYa 中主要分析模块之间的功能关系
该网络图展示了多组学研究中关键分析模块间的互连关系。主要节点代表核心分析领域:基因组分析(橙色)、免疫分析(蓝色)、生存分析(洋红色)、表达谱分析(紫色)和机器学习(绿色)。实线表示强关联,虚线表示中度或弱关联。该网络凸显了基因组变异如何影响免疫应答通路与生存结局,以及表达谱分析如何衔接生存分析与机器学习方法。这一整合框架体现了 FigureYa 可视化工具如何促进跨生物医学研究领域的多组学数据综合解读。
2. 核心可视化模块功能演示
FigureYa 代码资源包提供了涵盖从基础统计分析到前沿技术多个领域的全面可视化解决方案,如 Figure 3 所示。
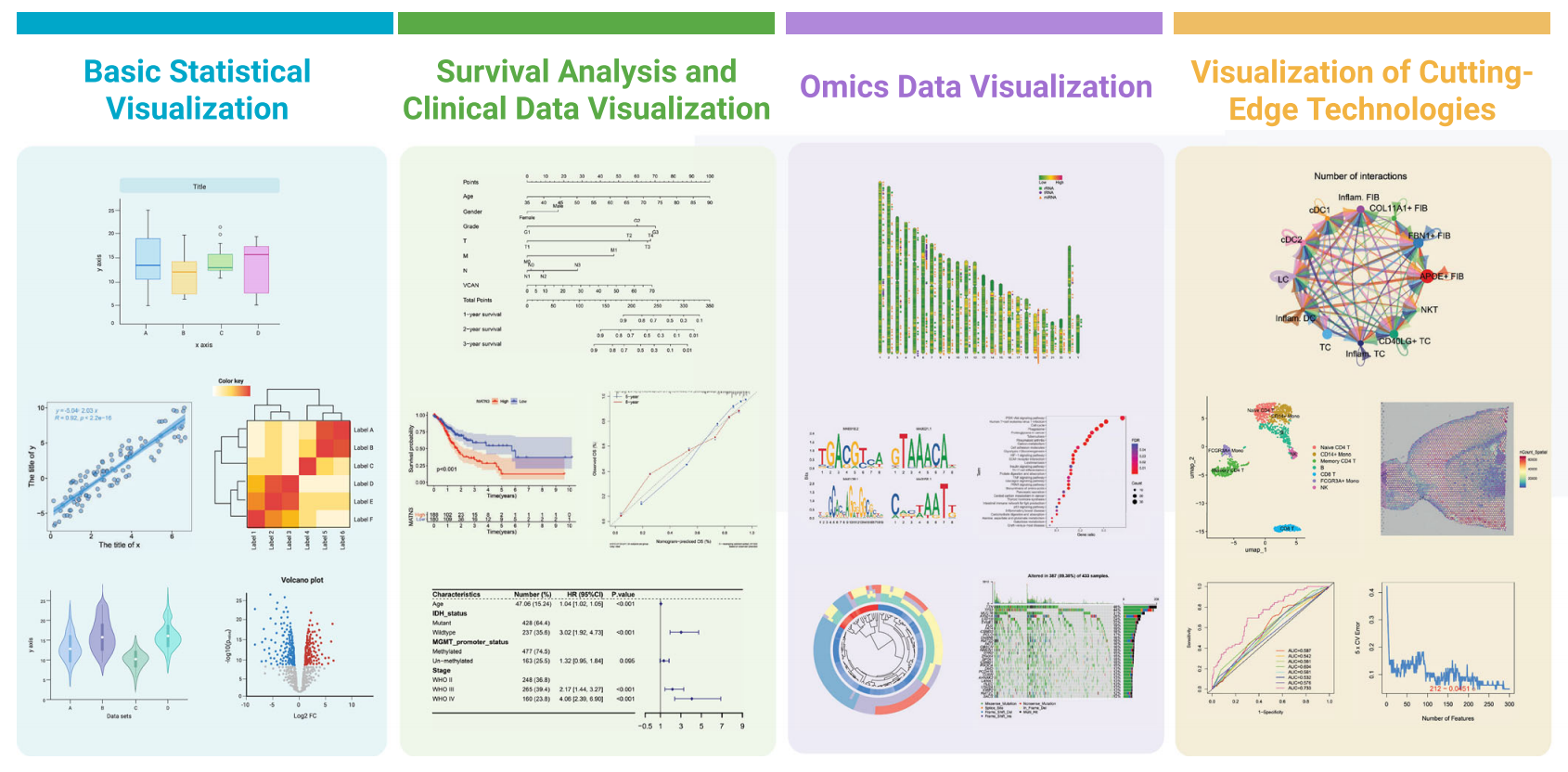
Figure 3. 生物医学研究数据可视化方法的分类
该图展示了 FigureYa 实现的四类主要可视化技术:基础统计可视化(蓝色)、生存与临床数据可视化(绿色)、组学数据可视化(紫色)以及前沿可视化技术(橙色)。每个类别均包含代表性方法及对应示例输出。基础统计方法包括箱线图、散点图和热图;生存与临床可视化涵盖 Kaplan-Meier 曲线和森林图;组学可视化包含序列模体和多维数据表征;前沿技术则以网络分析和空间转录组学为特色。这种结构化分类使研究人员能够根据具体数据类型和分析目标高效选择可视化方法。
2.1 基本统计可视化
2.1.1 箱线图和小提琴图系列
FigureYa 提供高度优化的箱线图和小提琴图工具,如 FigureYa12box 和 FigureYa162boxViolin。这些工具能够自动计算并标注组间统计显著性,支持多重检验方法,并通过简单参数调整实现复杂分组比较。其参数化特性允许研究人员自定义配色方案、调整图形比例、应用预定义主题样式,快速生成符合各期刊版式要求的可视化输出。值得注意的是,其内置数据处理功能包含自动处理缺失值、识别标注异常值,并支持数据转换,从而简化数据预处理流程。
2.1.2 相关分析可视化
FigureYa 提供一系列相关性分析可视化工具,包括 FigureYa37correlation 和 FigureYa126CorrelationHeatmap。这些工具可自动计算相关系数、执行显著性检验并进行层次聚类,同时支持智能处理缺失值,提供成对删除或插值等选项。针对更复杂的多元关联,FigureYa76corrgram 可生成相关矩阵,而 FigureYa152DoubleCorPlot 支持双相关性比较。这些工具能自动筛选显著相关变量对,应用多重检验校正,并根据相关性强度动态调整视觉编码 —— 使研究人员能高效识别数据中的关键关联模式。
2.1.3 参数定制功能
FigureYa 的基础统计可视化工具在四个关键维度提供强大的参数化定制功能:视觉风格、数据处理、统计分析与版式结构。所有脚本支持主题参数,提供符合各期刊格式要求的预定义样式;配色方案参数适配多种专业色彩体系;文字参数可调整字体类型与大小。数据处理方面提供变换、标准化与缩放参数;统计分析支持多重检验方法与校正程序;版式结构允许控制图形比例与元素排布。这种"简单参数,复杂效果"的设计理念,使研究人员能够以最小学习成本生成符合专业出版标准的定制化统计图形。
2.2 生存分析和临床数据可视化
2.2.1 生存曲线系列
FigureYa 提供完整的生存分析可视化工具套件,包括实现标准 Kaplan-Meier 生存曲线的 FigureYa1survivalCurve,可自动计算生存率与置信区间、生成风险表并标注 p 值。针对更复杂的生存分析场景,提供包括 FigureYa4bestSeparation(最佳截断点分析)、FigureYa171subgroupSurv(亚组分析)和 FigureYa183condSurv(条件生存分析)等专用工具。这些工具能智能处理随访数据集,自动识别事件状态与时间变量,并计算复杂生存统计量 —— 显著简化生存分析流程,使生物统计学经验有限的研究人员也能生成达到发表质量的生存分析图。
2.2.2 临床预测模型可视化
FigureYa 提供临床预测模型的专用可视化工具,包括 FigureYa30nomogram 可通过自动将多元回归模型转换为直观的图形化评分系统来构建临床列线图。FigureYa33DCA 执行决策曲线分析,通过计算一系列阈值概率下的净收益来量化预测模型的临床效用。FigureYa138NiceCalibration 生成校准曲线以评估预测概率与观测结果的一致性。这些工具的集成使用显著简化了临床预测模型的开发与评估流程,使缺乏专业统计学背景的医学研究人员也能构建符合方法学标准的模型。
2.2.3 多变量分析结果的介绍
FigureYa 提供多变量分析结果可视化的专用工具,包括 FigureYa6rHRs 可生成标准化森林图,自动计算并展示点估计值、置信区间和 p 值。FigureYa47HRtable 以表格形式呈现多变量分析结果,并自动格式化数值输出。更高级的工具包括 FigureYa193RiskTable(多变量 Cox 回归风险表)和 FigureYa238corRiskMut(风险因素-基因突变关联分析)。这些工具的集成使用提升了临床研究数据分析的效率与标准化程度,确保结果的一致性和可复现性。
2.3 组学数据可视化
2.3.1 基因组和表观基因组可视化
FigureYa 提供基因组与表观基因组数据的专用可视化工具,包括 FigureYa3genomeView 可生成基因组信号轨道图,将基因注释与多种组学信号的多轨道呈现相结合。FigureYa155ATAC 专门针对 ATAC-seq 数据定制可视化方案,而 FigureYa107ChIPheatmap 则为 ChIP-seq 数据集特别设计。针对全基因组分析,FigureYa10chromosome 可生成染色体级别热图,FigureYa14circos 则构建 Circos 风格环形图。这些工具的技术创新点在于能促进数据整合与生物学解读,显著简化基因组学研究中的数据可视化流程。
2.3.2 转录组数据分析图形
FigureYa 提供丰富的转录组数据分析可视化工具,包括 FigureYa9heatmap 可生成基因表达热图并自动进行数据标准化与聚类分析。FigureYa59volcano 提供差异表达分析的火山图,而 FigureYa60GSEA_clusterProfiler 则实现基因集富集分析结果的高质量可视化。更高级的工具包括 FigureYa15WGCNA(加权基因共表达网络分析)和 FigureYa249Regulon(转录调控网络分析)。这些工具的集成使用支持完整的转录组数据分析流程 —— 从原始数据处理到功能阐释再到调控网络分析 —— 从而提升研究效率并确保结果一致性。
2.3.3 多组学整合可视化
FigureYa 提供多组学整合的创新可视化工具,包括 FigureYa14circos 可生成 Circos 风格环形图,能够将多层次数据关联整合至单一图形中。FigureYa45iCluster 执行多组学整合聚类分析,FigureYa258SNF 则实现相似网络融合算法。针对特定多组学整合任务,FigureYa122mut2expr 支持基因突变-表达关联分析,而 FigureYa304MAGIC 可跨多个组学层面进行整合聚类。这些多组学整合工具的应用深化了对复杂生物学问题的研究,使研究人员能够从多维度探索生物系统。
2.4 尖端技术的可视化
2.4.1 单细胞分析的可视化
FigureYa 提供完整的单细胞分析可视化工具套件,包括基于 UMAP 降维的 FigureYa93UMAP 和用于标记基因识别与可视化的 FigureYa224scMarker。针对进阶单细胞分析,FigureYa267scCellChat 支持细胞间通讯网络分析,而 FigureYa306slingshot 可实现轨迹推断。这些工具的集成使用构成完整的单细胞分析平台,使未经专业生物信息学训练的研究人员也能开展深度单细胞研究。
2.4.2 空间转录组数据可视化
FigureYa 提供空间转录组学数据的专用可视化工具,包括用于空间基因表达定位的 FigureYa239ST_PDAC,可将基因表达数据与组织空间坐标对齐。FigureYa323STpathseq 支持空间通路活性分析,而 FigureYa309cell2location 可实现空间细胞类型反卷积。这些工具融合了多项创新技术,包括空间自适应平滑算法与空间差异表达分析,使研究人员能够从空间转录组数据集中获取丰富的生物学见解。
2.4.3 机器学习结果的可视化
FigureYa 提供机器学习结果的专业可视化工具,包括 FigureYa293machineLearning 可通过统一接口支持多种算法,实现模型构建与评估的全面可视化。FigureYa316RF_XGBoost_Boruta 提供特征重要性分析与可视化,实现多种特征相关性评估方法。这些工具显著简化机器学习模型构建与解读流程,使生物医学研究人员能够应用先进算法,并更好地理解模型决策依据。
3. FigureYa 工作流程的技术架构和效率评估
如 Figure 4 所示,FigureYa 通过其"即插即用"工作流和标准化代码架构革新了生物医学数据可视化。本研究开发的工作流将复杂可视化过程简化为四个连贯步骤:选择合适代码模板、替换用户特定数据、执行标准化脚本、生成出版级图表输出。这种设计使无编程经验的研究人员也能高效产出符合发表标准的可视化结果。标准化代码架构遵循六阶段结构,涵盖数据输入、清洗、转换、分析、可视化生成和结果保存,每个阶段均配有详细注释与参数说明,确保代码可读性与可维护性。R 包使用频率分析显示 ggplot2、tidyverse 和 ComplexHeatmap 等工具在系统中的核心地位,这些科研与数据分析领域公认的专业可视化包被精心整合至 FigureYa 框架,构成系统的技术基础。这种集成不仅提升了平台的图形生成能力,更使复杂可视化任务得以简化和标准化,确保最终输出的专业质量与一致性。
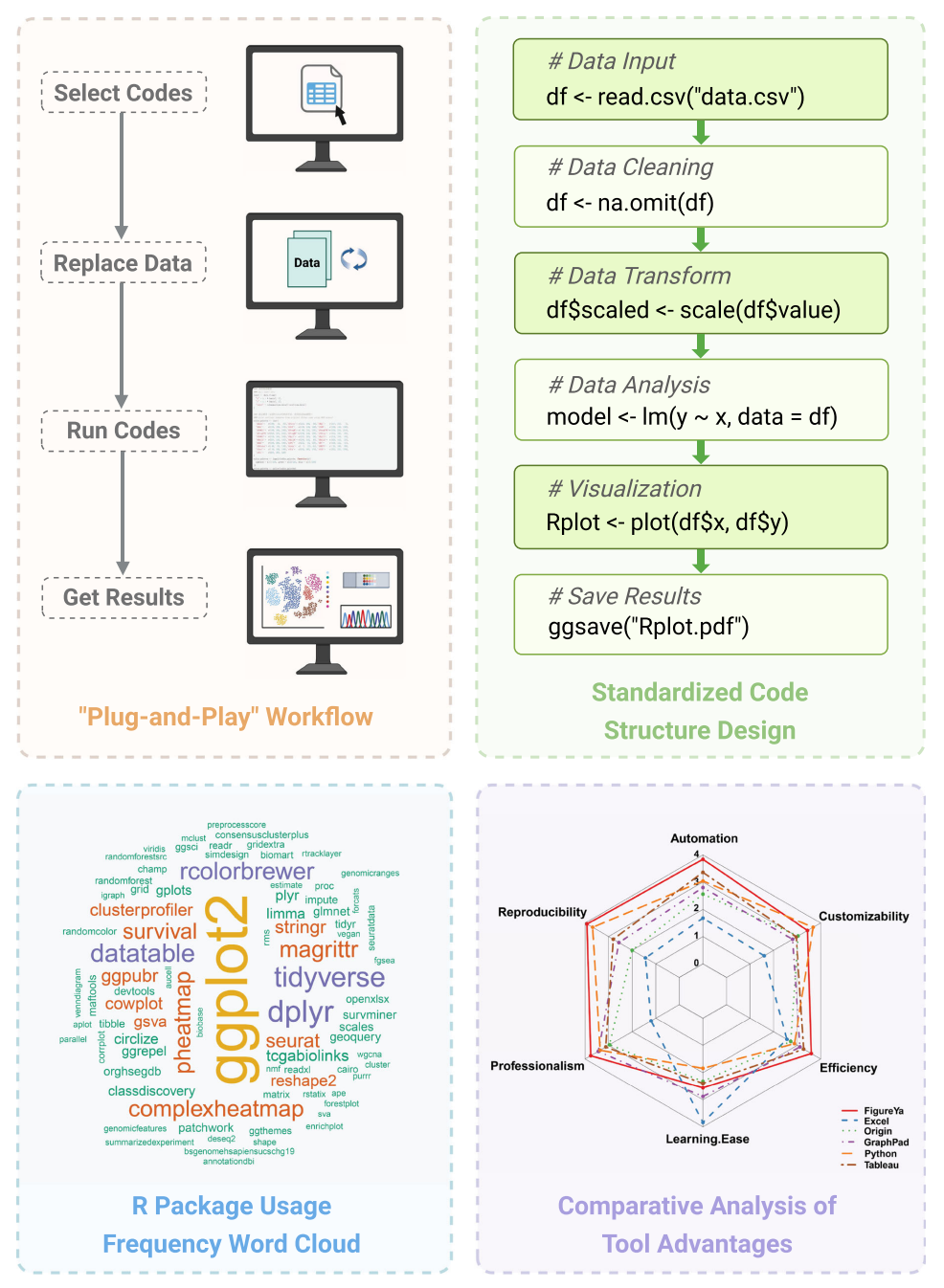
Figure 4. FigureYa 的工作流设计和实施框架
该图展示了 FigureYa 的 "即插即用"工作流程(左面板)与标准化代码架构(右面板)。左面板描绘了四个步骤:代码选择、数据替换、执行和结果可视化。右面板概述了标准化 R 代码框架,包含数据输入、清洗、转换、分析、可视化和结果导出的顺序阶段。底部面板包含 R 包使用频率词云图(左),突显了 ggplot2、tidyverse 和 ComplexHeatmap 等包的核心作用;以及工具优势的对比分析(右),涵盖自动化、可复现性、可定制性、专业性、高效性和易学性六个维度。这种标准化方法显著降低了技术门槛,同时确保生成专业质量的可视化结果。
多维评估结果表明,FigureYa 在六个关键维度实现了最佳平衡:自动化水平、可复现性、专业标准、时间效率、易学性及适度可定制性。该工具特别适用于生物医学研究场景中的跨学科团队协作。这一创新工作流不仅消除了技术壁垒,更重要的是使研究人员能够专注于科学探究而非技术实现细节。
4. 应用案例研究
为更好展示 FigureYa 多模块集成的优势,通过 Figure 5 所示的案例研究说明 FigureYa 系统的应用流程与实践价值。该案例展示了应用 FigureYa 探究肿瘤免疫微环境与患者预后关系的工作流程,凸显其跨模块的整合分析能力。虽然此示例重点呈现多模块在肿瘤免疫微环境分析中的协同作用,但并未涵盖 FigureYa 应用场景的全部范畴。实际上,GitHub 仓库中每个 FigureYa 模块文件夹均为独立用例,包含真实数据、可视化脚本和输出示例,覆盖实体瘤(如结直肠癌、肝癌)、血液肿瘤(如AML、淋巴瘤)等疾病背景,以及蛋白质组学、代谢组学、免疫组学等多组学层面。流程始于数据预处理,包括通过 FigureYa22FPKM2TPM、FigureYa34count2FPKMv2、FigureYa23count2TPM 等模块实现 FPKM 与 TPM、count 等表达矩阵格式的相互转换。本案例中将 FPKM 数据转换为 TPM 后,进行对数转换以优化后续分析结果。
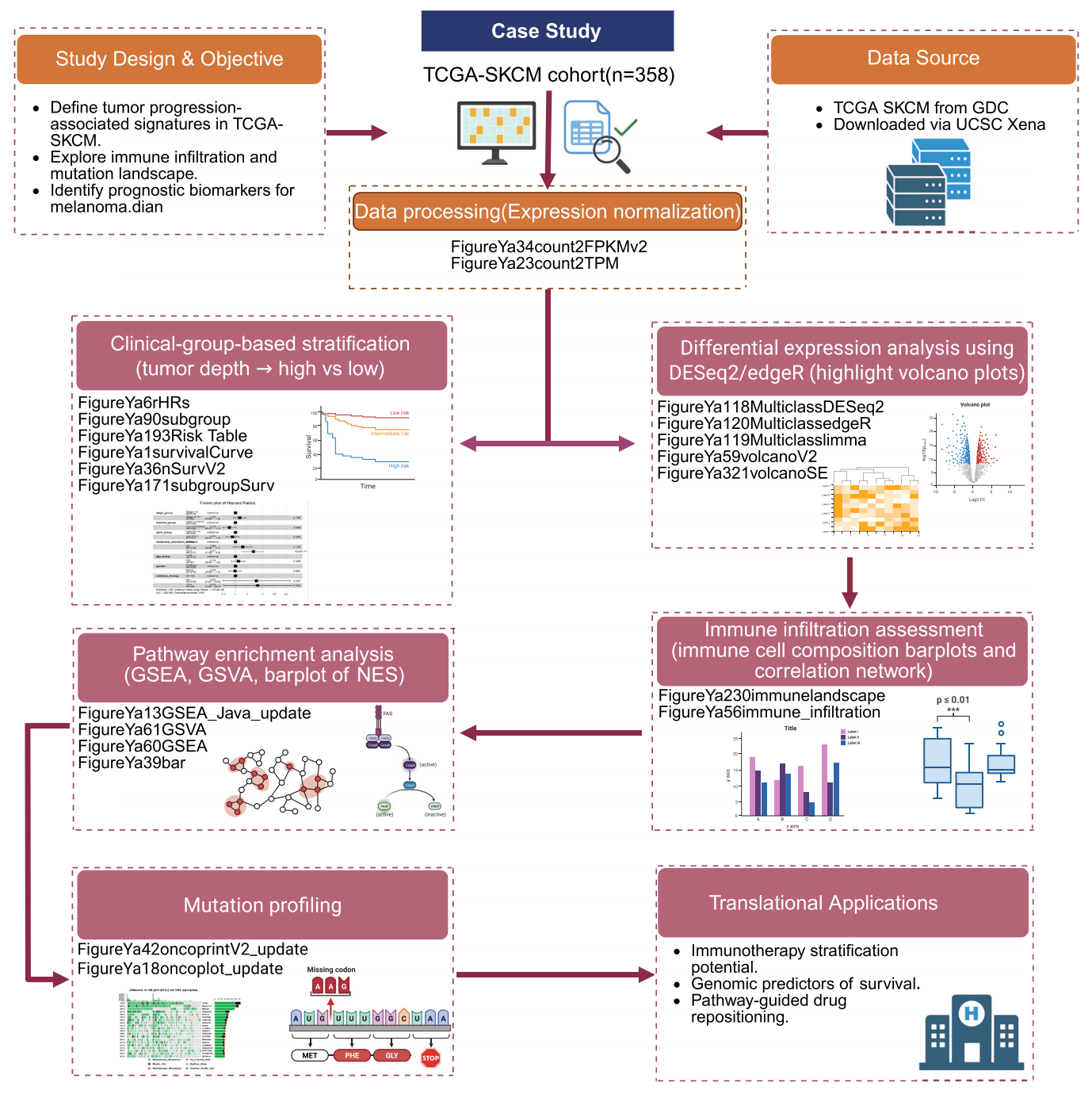
Figure 5. 癌症基因组图谱 - 皮肤黑色素瘤(TCGA-SKCM)队列的多组学分析工作流程使用 FigureYa 系统
该分析涉及 358 个 TCGA-SKCM 样本,首先使用 FigureYa34count2FPKMv2 和 FigureYa23count2TPM 进行标准化处理。随后工作流分为三个主要分支:(1) 基于肿瘤浸润深度(高/低)进行临床分组,开展生存分析与风险评估;(2) 采用 DESeq2 和 edgeR 进行差异表达分析,通过火山图实现可视化;(3) 进行免疫浸润评估,包括免疫细胞组成谱分析和免疫网络可视化。基于上述结果,开展通路富集分析(GSEA、GSVA、NES条形图)与突变景观分析,系统表征肿瘤浸润深度相关分子特征与免疫微环境模式。GSEA:基因集富集分析;GSVA:基因集变异分析;NES:标准化富集得分。
随后,通过结合生存分析与风险评估,作者发现黑色素瘤样本中肿瘤浸润深度与患者预后存在显著相关性。基于此分层结果,使用 FigureYa118MulticlassDESeq.2 和 FigureYa120MulticlassedgeR 等模块进行差异表达分析,并借助 FigureYa59volcanoV2 和 FigureYa321volcanoSE 等可视化模块呈现结果。对差异表达基因进一步使用 FigureYa13GSEA_Java_update、FigureYa61GSVA 和 FigureYa60GSEA 等模块进行分析,并通过 FigureYa39bar 实现可视化。在免疫分析环节,采用 FigureYa234panImmune 评估免疫浸润程度,利用 FigureYa81immune_network 解析免疫细胞相互作用。最后通过 FigureYa42oncoprintV2_update 和 FigureYa18oncoplot_update 模块对 TCGA-SKCM 样本的突变谱进行可视化呈现。
本案例展示了 FigureYa 模块间的灵活互操作性,使该平台高度适用于多类型肿瘤研究的可视化任务。该案例明确了黑色素瘤的关键预后因素并探索其潜在机制。Figure 6A 展示了包括 stage、Breslow depth 和 Clark grade 的多因素 Cox 回归分析结果。多因素 Cox 回归分析表明 Breslow depth(>3.0 mm vs. ≤3.0 mm;HR=1.54,95% CI:0.88-2.7,p<0.001)与溃疡状态是患者预后的关键预测因子(Figure 6B,C),这一发现通过 Kaplan-Meier 生存分析得到进一步验证(p<0.0001)。基于 Breslow depth 的差异表达分析识别出多个显著改变基因,包括角蛋白家族成员(KRT14、KRT5)和黑素细胞标志物(TYRP1)的上调,以及免疫相关基因(IGHV3-7、IGHM)的下调(Figure 6D)。免疫微环境分析显示不同深度组间免疫细胞浸润存在显著差异,尤其在 M1 型巨噬细胞(***p<0.001)、单核细胞(***p<0.001)和活化树突状细胞(***p<0.001)中(Figure 6E)。GSEA 分析进一步揭示差异表达基因主要富集于适应性免疫应答和淋巴细胞介导的免疫相关通路(Figure 6F)。突变谱分析显示 94.23% 样本存在基因组改变,其中 TTN(72%)、MUC16(54%)和 BRAF(54%)为最高频突变基因(Figure 6G)。
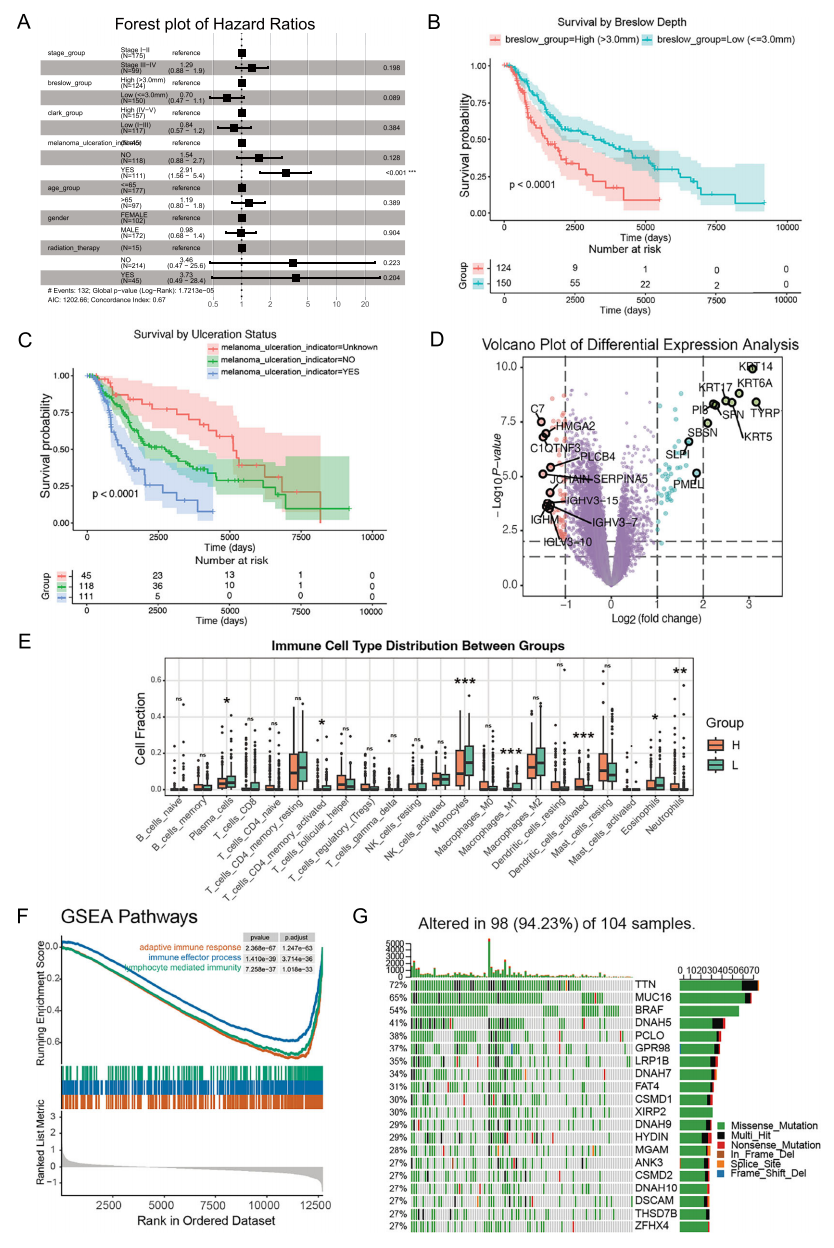
Figure 6. 与黑色素瘤预后相关的临床特征,分子表型和免疫微环境的综合分析
(A) 展示临床特征(分期、Breslow深度、Clark分级)多因素 Cox 回归分析的森林图,包含风险比与95%置信区间。
(B) 按 Breslow 深度分层(>3.0 mm vs. ≤3.0 mm)的 Kaplan-Meier 生存分析,显示对预后的显著影响(p<0.0001)。
(C) 基于溃疡状态(是/否/未知)的生存分析,证实与预后的强相关性(p<0.0001)。
(D) 基于 Breslow 深度分组的差异表达基因火山图,突出显示关键基因。
(E) Breslow 深度组间免疫细胞浸润分布。星号表示显著性水平。
(F) 展示差异表达基因中免疫相关通路富集的GSEA图。
(G) 显示黑色素瘤样本突变景观的瀑布图。不同突变类型用颜色标注:错义突变(蓝色)、无义突变(红色)、移码插入(绿色)、框内缺失(橙色)、剪接位点(紫色)及其他(灰色)。注:统计学显著性标注如下:p<0.05,p<0.01,p<0.001。GSEA:基因集富集分析。
这些发现系统阐明了黑色素瘤浸润深度、分子特征、免疫微环境与突变谱之间的关联,为理解疾病机制及优化治疗策略提供了重要依据。
讨论
在生物医学研究中,数据可视化对识别模式、验证假设及有效呈现结果具有关键作用。由 317 个模块化、任务导向型 R/Python 脚本构成的 FigureYa 框架,通过系统化开发的专业可视化工具组,全面应对生物医学研究中的数据可视化挑战。该平台显著提升研究效率,促进跨学科协作,为生物医学研究的创新推进与临床转化提供有力支撑。
FigureYa 系统为研究人员拓展了数据可视化的可能性,有助于发现原本可能被忽视的数据规律。例如在基因表达分析中,除标准热图外,系统可自动生成带有分类信息的聚类热图、展示基因层级差异的 p 值、并提供多分类体系的对比视图。这种多视角的数据呈现方式,使研究人员能够通过数据发现隐藏的模式与关联,从而促进新发现。
通过高度标准化与参数化设计,FigureYa 从根本上改变了研究人员的时间分配方式。传统上,数据可视化约占用总研究时间的 25%–40%。FigureYa 显著减轻了这一技术负担。与缺乏验证或兼容性的零散在线代码片段不同,每个 FigureYa 模块均经过测试、文档化和优化,可直接投入科研使用,使研究人员能更专注于结果解读。例如使用 FigureYa9heatmap 可视化基因表达数据时,研究人员可聚焦于生物学问题,而无需纠结于技术实现细节。这种优化后的时间分配不仅提升研究效率,更拓展科学探索的深度与广度,使研究人员能够验证更多假设、测试更多模型,最终产出更多原创性发现。
通过降低技术门槛,FigureYa 显著促进了不同背景研究人员之间的跨学科合作。FigureYa 提供的标准化可视化代码作为一种"通用语言",连接了不同学科的专业人员:临床研究人员无需深入理解复杂算法即可应用先进分析方法,而数据科学家能更好地理解生物学问题的可视化需求。与 ChatGPT 和 DeepSeek 等根据用户提示自动生成图表的 AI 辅助可视化平台不同,FigureYa 提供对参数的完全控制、一致的生物学可解释性和可复现性。其开源结构支持透明编辑、同行验证并可与学术工作流集成,这对严谨的科学研究和论文发表至关重要。此外,当前 AI 模型仅能生成需要迭代优化的孤立代码片段,而 FigureYa 提供端到端的模块化框架,每个组件均具有互操作性且专为生物医学数据定制,显著减少了试错成本。例如在肿瘤免疫微环境研究中,临床肿瘤学家可使用 FigureYa56immune_inflitration 分析免疫浸润;免疫学家可应用 FigureYa267scCellChat 探索细胞通讯网络;数据科学家可利用 FigureYa316RF_XGBoost_Boruta 识别关键特征,从而形成完整的研究工作流。这种协作模式不仅加速研究进展,更促进多学科知识的融合与创新。
高质量数据可视化对研究成果的接收率、引用率和传播度产生显著正向影响。FigureYa 提供专业级可视化工具,使研究人员能制作更具说服力和视觉冲击力的展示内容。在高度竞争的研究领域,伴随清晰专业可视化的相同科学发现往往能获得更多认可并产生更广泛影响。例如使用 FigureYa14circos 生成的多组学整合环形图,可将复杂的多维关系直观整合于单一图形中,极大提升研究成果的传播力。此外,FigureYa 的标准化输出提升了研究的可复现性与透明度,进一步强化其学术可信度与长期影响力。
与 Excel 和 Origin 等通用可视化软件相比,FigureYa 在生物医学研究专业可视化方面具有显著优势。自动化方面,FigureYa 可自动完成数据标准化、统计检验和结果标注,而通用工具通常需手动操作。可复现性方面,FigureYa 基于代码的框架确保分析过程完全可追溯与复现。专业化方面,FigureYa 提供高度定制化解决方案,如 FigureYa18oncoplot(肿瘤突变谱分析)和 FigureYa239ST_PDAC(空间转录组可视化)。尽管针对简单可视化任务 FigureYa 可能相对复杂,但其在处理高维数据和复杂分析工作流时的优势尤为突出。因此 FigureYa 与通用工具在许多方面具有互补性,研究人员可根据任务复杂度选择最合适工具,以优化整体科学可视化流程。
FigureYa 通过采用模块化、可互操作的基于脚本的架构,区别于传统的可视化平台或模板工具。每个模块代表一个独立可执行且完全文档化的单元,并通过标准化输入输出结构实现相互兼容。"即插即用"概念并非指图形界面,而是指R脚本在不同组学层析和分析任务间可重用与互连的能力,且仅需最少人工调整。例如 Figure6 所示的黑色素瘤案例中,先使用 FigureYa22FPKM2TPM 转换基因表达数据,随后进行基于生存的分层与差异表达分析(FigureYa118MulticlassDESeq.2)、富集可视化(FigureYa13GSEA_Java_update、FigureYa39bar)以及免疫特征分析(FigureYa234panImmune)。该分析链展示了模块输出如何直接作为另一模块的输入。这种模块化互操作性在保持生物医学数据可视化清晰度与可复现性的同时,支持工作流的灵活构建。
尽管 FigureYa 为生物医学数据可视化提供了灵活且可复现的解决方案,仍需承认若干局限性。首先,该框架主要依赖静态 R 脚本执行,对于需要实时或交互式可视化的场景(如网络仪表盘或临床决策支持系统)可能并非最优选择。其次,虽然模块化结构支持多种组学数据类型分析,但在处理超大规模单细胞数据集时可能面临性能瓶颈,特别是在聚类和降维等内存密集型步骤中。
在后续迭代中,计划通过集成交互式可视化库(如 plotly、shiny)支持,并利用并行化和轻量级数据结构优化计算模块的可扩展性,以扩展 FigureYa 的功能。这些改进旨在提升平台在高通量研究和用户交互场景中的实用性。
总结
FigureYa 代码资源包通过提供 317 种专业可视化工具,填补了生物医学研究可视化领域的空白。基于"替换数据即用"的设计理念,该资源库显著降低了技术门槛,使研究人员能够轻松创建高质量图表。FigureYa 采用标准化代码架构与精准注释,在专业级输出与用户友好操作之间取得平衡,从而加速数据解读进程。通过消除技术壁垒,研究人员能更专注于科学探索,并最大程度减少工作流程中的认知中断。标准化可视化输出提升了研究成果的可解读性与学术传播力,成为跨学科交流的通用语言,促进多学科知识的融合与创新发展。FigureYa 将持续完善平台功能,进一步推动生物医学研究的创新与临床转化。
--------------- 结束 ---------------
注:本文为个人学习笔记,仅供大家参考学习,不得用于任何商业目的。如有侵权,请联系作者删除。

