DiffDock 环境安装和使用教程
写在前面
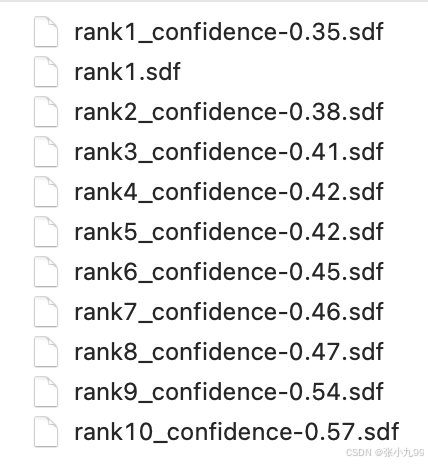
蛋白质的生物学功能可以通过与其结合的小分子配体(如药物)来调节。预测小分子配体化合物与受体蛋白质的结合构象预测对于药物设计至关重要。传统的分子对接方法基于检索和打分函数(score function),首先是搜索配体和受体可能的结合模式(位置、配体在口袋内的取向、配体构象),候选集可能非常大(百万级),然后对候选结合复合物打分。近期把分子对接看作回归问题来处理的深度学习方案逐渐增多,虽然减少了运行时间,但尚未显着提高准确性。研究团队创新性的将分子对接(molecular docking)视为一个生成建模问题,并采用了时下在图像生成等领域相当热门的生成扩散模型,开发了 DiffDock。模型概览如图所示,该模型以单独的配体和蛋白质作为输入文件。随机采样的初始对接位姿在平移、旋转和扭转自由度上反向扩散去噪。被采样的对接位姿按告高置信度模型,产生一系列预测和置信度评分,并按照评分排序,获得最终预测。DiffDock 在 PDBBind 上获得了 38% 的 TOP1 成功率(RMSD<2Å),显著优于传统对接方法(SMINA,QuickVina-W,GLIDE和GNINA,成功率约23%)和最先进的深度学习方法(EquiBind和TANKBind,成功率约20%)。与此同时,速度也有3-12倍提升。对于柔性折叠的复合物结构,此前各种方法最大仅达到10.4%的对接成功率,而DiffDock仍达到了21.7%的成功率水平。
DiffDock特点:
1. 基于扩散模型:DiffDock 采用扩散模型来生成配体的位置、方向和构象,这是一种新颖的方法,有别于传统的基于评分函数的对接方法。
2. 全局搜索:无需预先定义对接区域,DiffDock 能够进行全面的全局搜索,探索更多可能的结合模式。
3. 端到端学习:DiffDock 能够直接从蛋白质和配体的原子坐标学习,无需人工特征工程。
4. 考虑水分子:将水分子纳入对接过程,提高了预测的准确性和真实性。
5. 高效性能:在多个基准测试中,DiffDock 展现出优于传统方法的性能,特别是在预测复杂蛋白质 - 配体相互作用方面。
6. 快速预测:DiffDock 能够在短时间内生成多个可能的对接构象,为药物设计和筛选提供了高效工具。
一、Docker安装和使用教程(更简单方便)
(一)安装教程
1. 获取代码:先把仓库克隆到本地(可选):
git clone https://github.com/gcorso/DiffDock.git
cd DiffDock
2. 使用 Docker 安装环境
方式一:直接用官方预构建镜像(推荐)
直接拉取官方提供的镜像:
docker pull rbgcsail/diffdock方式二:自己构建镜像
在仓库根目录执行:
docker build . -f Dockerfile -t diffdock这样会生成一个名为 diffdock 的镜像。
3. 检查 GPU 是否可用
先用官方 CUDA 镜像测试 GPU 驱动是否连通:启动一个临时的 CUDA 11.7 + Ubuntu 22.04 容器,挂载宿主机所有 GPU,运行 nvidia-smi,然后退出并自动清理容器。如果本地没有该镜像(nvidia/cuda:11.7.1-devel-ubuntu22.04),Docker 就去 Docker Hub 上拉取,随后显示显卡信息;
docker run --rm --gpus all nvidia/cuda:11.7.1-devel-ubuntu22.04 nvidia-smi
--rm:容器在运行完后 自动删除(不会在本地留下痕迹)。适合做一次性的测试命令,比如nvidia-smi。如果不用--rm,容器退出后会处于 stopped 状态,你可以用docker ps -a看到一堆历史容器。
如果能看到显卡信息说明 GPU 正常可用。
4. 启动容器
有 GPU(推荐)
docker run -it --gpus all --entrypoint /bin/bash rbgcsail/diffdock
无 GPU(会很慢)
docker run -it --entrypoint /bin/bash rbgcsail/diffdock5. 容器内部环境激活
进入容器后,激活环境即可进行后续的推理和计算:
micromamba activate diffdock(二)使用教程
1、创建DiffDock的文件目录
# 在个人用户目录下创建新文件夹
mkdir DiffDock_files
mkdir DiffDock_files/diffdock_models# 进入模型权重下载目录
cd DiffDock_files/diffdock_models2、下载DiffDock模型权重
# 下载模型权重文件
curl -L -o ./diffdock_models.zip https://github.com/gcorso/DiffDock/releases/download/v1.1/diffdock_models.zip# 解压权重文件
unzip diffdock_models.zip3、准备结构和小分子文件
# 创建文件目录
mkdir DiffDock_files/1_project # 将 .pdb 和 .mol2 或者 .sdf文件传入# 方案一:修改文件所属用户
#在 Docker 容器里,appuser 默认就是 UID=1000 / GID=1000
sudo chown -R 1000:1000 1_project/ # 把宿主机目录 /data/1_project 的所有者递归改为 UID=1000, GID=1000,避免后续会出现文件# 把整个 torch 缓存目录的 owner 改为 1000:1000
sudo chown -R 1000:1000 ~/.cache/torch
# 注意:果你把这些文件 chown 给 1000:1000,容器能读,但你本机的用户(如果不是 1000)可能就读不了了(除非你用 sudo)。# 方案二:修改文件权限
find 1_project -type d -exec chmod 777 {} \; # 需要docker用户写入
find 1_project -type f -exec chmod 666 {} \;find ~/.cache/torch -type d -exec chmod 755 {} \;
find ~/.cache/torch -type f -exec chmod 644 {} \;
-
目录 (d) →
755 (rwxr-xr-x):
你 (owner) 可读写执行;组用户和其他用户可读执行(能进入目录和读取内容)。 -
文件 (f) →
644 (rw-r--r--):
你可读写;其他人只读。
4、修改配置信息
下载配置文件:
wget https://raw.githubusercontent.com/gcorso/DiffDock/main/default_inference_args.yaml
修改配置文件的confidence_model_dir和model_dir目录为自己下载模型的目录:
confidence_model_dir: ./models/diffdock_models/confidence_modelmodel_dir: ./models/diffdock_models/score_model4、运行docker
docker run -it --gpus all \-v ./diffdock_models:/home/appuser/DiffDock/models/diffdock_models:ro \-v ./1_project:/data/1_project \-v ~/.cache/torch:/home/appuser/.cache/torch \-v ./default_inference_args.yaml:/home/appuser/DiffDock/default_inference_args.yaml:ro \--entrypoint /bin/bash rbgcsail/diffdock
docker run -it
以交互模式启动容器,并分配终端(i=interactive,t=tty)。
--gpus all
把宿主机所有 GPU 映射到容器中,确保 DiffDock 可以调用 CUDA 加速。
-v ./diffdock_models:/home/appuser/DiffDock/models/diffdock_models:ro
将宿主机当前目录下的 模型文件夹diffdock_models挂载到容器路径/home/appuser/DiffDock/models/diffdock_models。
:ro表示 只读,避免容器内部意外修改模型权重。
-v ./1_project:/data/1_project
将宿主机的实验项目目录挂载到容器/data/1_project,便于在容器中直接读取输入 PDB/SMILES 文件并输出结果。
-v ~/.cache/torch:/home/appuser/.cache/torch
把宿主机的 PyTorch 缓存目录挂载到容器,避免每次运行都要重新下载 FAIR-ESM 模型权重(默认会下载到.cache/torch/hub/checkpoints/)。
👉 这样可以大幅减少等待时间。
-v ./default_inference_args.yaml:/home/appuser/DiffDock/default_inference_args.yaml:ro
使用宿主机修改好的配置文件,覆盖容器内部默认的default_inference_args.yaml。
这样就不需要每次进入容器后再sed修改模型路径,配置一次,永久生效。
--entrypoint /bin/bash
指定进入容器后直接启动bash终端,而不是镜像默认的入口程序。
rbgcsail/diffdock
要运行的 DiffDock 官方镜像。
5、运行推理
python -m inference \--config default_inference_args.yaml \--protein_path /data/1_project/test.pdb \--ligand /data/1_project/test.sdf \--out_dir /data/1_project/results
成功运行:
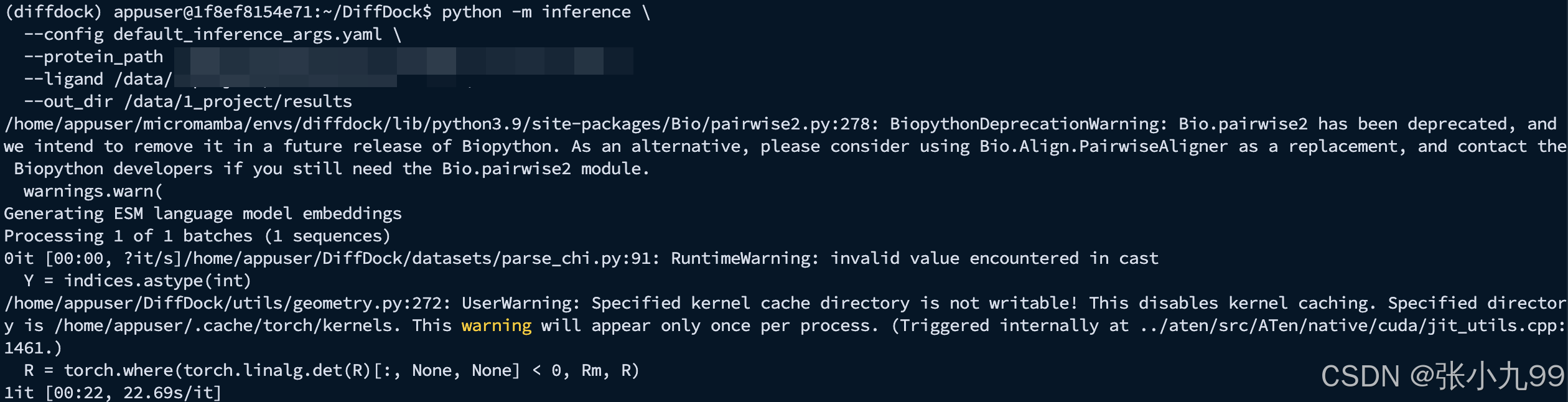
输出文件如下:

二、conda安装和使用教程(更复杂但推荐)
(一)安装教程
1、克隆仓库代码
git clone https://github.com/gcorso/DiffDock.git
cd DiffDock
2、下载权重文件
# 存储模型权重的目录
cd models# 下载模型
curl -L -o ./diffdock_models.zip https://github.com/gcorso/DiffDock/releases/download/v1.1/diffdock_models.zip# 解压文件
unzip diffdock_models.zip
注意:也可以不手动下载,第一次执行命令的时候会自动下载模型权重到目录文件下的!
3、修改默认配置参数
# 查看配置参数文件
vim default_inference_args.yaml将文件中的 confidence_model_dir 和 model_dir 修改为正确的目录,如下:
actual_steps: 19
ckpt: best_ema_inference_epoch_model.pt
confidence_ckpt: best_model_epoch75.pt
confidence_model_dir: ./models/confidence_model
different_schedules: false
inf_sched_alpha: 1
inf_sched_beta: 1
inference_steps: 20
initial_noise_std_proportion: 1.4601642460337794
limit_failures: 5
model_dir: ./models/score_model
no_final_step_noise: true
no_model: false
no_random: false
no_random_pocket: false
ode: false
old_filtering_model: true
old_score_model: false
resample_rdkit: false
samples_per_complex: 10
sigma_schedule: expbeta
temp_psi_rot: 0.9022615585677628
temp_psi_tor: 0.5946212391366862
temp_psi_tr: 0.727287304570729
temp_sampling_rot: 2.06391612594481
temp_sampling_tor: 7.044261621607846
temp_sampling_tr: 1.170050527854316
temp_sigma_data_rot: 0.7464326999906034
temp_sigma_data_tor: 0.6943254174849822
temp_sigma_data_tr: 0.92998025315726724、用 environment.yml 创建基础环境
注意:
由于官方提供的environment.yml里有多个pip:段 ,conda 会按顺序逐段调用pip。只要其中任一段报错(比如openfold那段),conda 仍然会把前面已经成功的部分写成“已完成”,导致你以为都装好了,但其实执行完上指令之后基本上只装好了conda里的依赖,大部分 pip 的深度学习包(torch, pyg 系列, openfold 等)都没装上;所以我们对官方提供的environment.yml做一些修改,并依次安装所需的包。
1)修改environment.yml文件如下
name: diffdock
channels:- conda-forge- pytorch- nvidia- pyg- defaults
dependencies:- python=3.9.18- pip- git- python-devtools- setuptools=69.5.1- prody==2.2.0- scipy==1.12.0- rdkit==2022.03.3
2)创建并环境
conda env create --file environment.yml
conda activate diffdock
5、安装 PyTorch(CUDA 11.7 对应版本)
这一步将安装 torch==1.13.1+cu117。如果你机器 CUDA 驱动足够新(驱动版本 ≥ 470),无需额外装 cudatoolkit。
# 先装 torch(CUDA 11.7)
pip install torch==1.13.1+cu117 torchvision==0.14.1+cu117 torchaudio==0.13.1 --extra-index-url https://download.pytorch.org/whl/cu117验证pytorch
python - <<'PY'
import torch
print("Torch:", torch.__version__, "CUDA:", torch.version.cuda, "GPU available:", torch.cuda.is_available())
PY
# 输出显示:Torch: 1.13.1+cu117 CUDA: 11.7 GPU available: True6、安装 PyTorch Geometric(与 torch 版本严格匹配)
# 再装 PYG 系列(指定 find-links)
pip install \torch-scatter==2.1.0+pt113cu117 \torch-sparse==0.6.16+pt113cu117 \torch-cluster==1.6.0+pt113cu117 \torch-spline-conv==1.2.1+pt113cu117 \torch-geometric==2.2.0 \--find-links https://pytorch-geometric.com/whl/torch-1.13.1+cu117.html注意:需要固定到有 whl 的对应版本,并让 pip 只在 PyG 提供的链接里找,否则可能会报错;
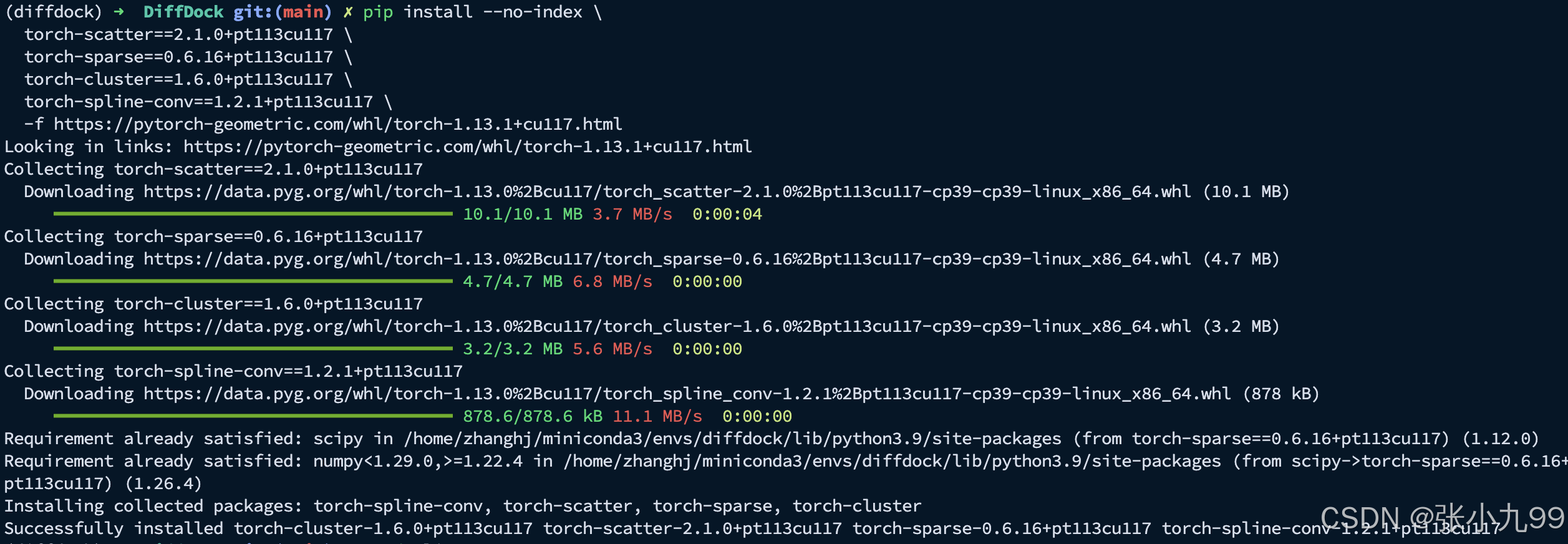
安装完成可以验证一下:
python - <<'PY'
import torch, torch_geometric as tg
import torch_scatter, torch_sparse, torch_cluster, torch_spline_conv
print("Torch:", torch.__version__, "CUDA:", torch.version.cuda, "GPU:", torch.cuda.is_available())
print("PyG:", tg.__version__)
print("scatter:", torch_scatter.__version__)
print("sparse:", torch_sparse.__version__)
print("cluster:", torch_cluster.__version__)
print("spline:", torch_spline_conv.__version__)
PY"""
输出信息如下:
Torch: 1.13.1+cu117 CUDA: 11.7 GPU: True
PyG: 2.2.0
scatter: 2.1.0+pt113cu117
sparse: 0.6.16+pt113cu117
cluster: 1.6.0+pt113cu117
spline: 1.2.1+pt113cu117
"""7、补装相关的Pip包
# 其余 pip 依赖
pip install \"dllogger@git+https://github.com/NVIDIA/dllogger.git" \"e3nn==0.5.1" "fair-esm[esmfold]==2.0.0" \"networkx==2.8.4" "pandas==1.5.1" "pybind11==2.11.1" \"pytorch-lightning==1.9.5" "scikit-learn==1.1.0" "torchmetrics==0.11.0" "requests"
注意:安装到这一步即可正常运行DiffDock的推理任务!
8、安装 CUDA 11.7 编译链(只装到 conda 环境,不动系统 CUDA,可选)
# 在当前环境安装 CUDA 11.7 工具链(含 nvcc)
conda install -y -n diffdock -c "nvidia/label/cuda-11.7.1" cuda-toolkit=11.7.1# 验证
nvcc -V # 看到 release 11.7 即正确which nvcc # 应该在 $CONDA_PREFIX/bin 下,比如/home/username/miniconda3/envs/diffdock/bin/nvcc9、安装 OpenFold(用刚装的 CUDA 11.7 编译,可选)
注意:必须加 --no-build-isolation,否则构建时用不到你环境里已安装的 torch,会报 ModuleNotFoundError: No module named 'torch'。
# 安装(关闭 build isolation,以便用到环境里的 torch/cuda)
pip install --no-build-isolation 'openfold @ git+https://github.com/aqlaboratory/openfold.git@4b41059694619831a7db195b7e0988fc4ff3a307'安装operfold的时候可能也会遇到一些问题,具体解决方案可以参考我之前安装ESMFold的博客:
ESMFold 安装教程-CSDN博客
(1)需要执行这几步操作,修改openfold的version由原来的2.0.0变成了1.0.0;
git clone --filter=blob:none --quiet https://github.com/aqlaboratory/openfold.git
cd ./openfold/
git rev-parse -q --verify 'sha^4b41059694619831a7db195b7e0988fc4ff3a307'
git fetch -q https://github.com/aqlaboratory/openfold.git 4b41059694619831a7db195b7e0988fc4ff3a307
git checkout -q 4b41059694619831a7db195b7e0988fc4ff3a307(2)将setup.py中原本的:
f'arch=compute_{major}{minor},code=sm_{major}{minor}',修改为当前设备nvcc支持的compute构架就可以,比如:
f'arch=compute_86,code=sm_86',以下指令列出当前 CUDA 编译器(nvcc)支持的所有 GPU 架构(compute capability)代码名称:
nvcc --list-gpu-arch # 方法二:直接查看支持的compute构架"""
例如输出如下:
compute_35
compute_37
compute_50
compute_52
compute_53
compute_60
compute_61
compute_62
compute_70
compute_72
compute_75
compute_80
compute_86
compute_87
"""(3)安装
pip install -e .(4)安装成功,显示如下:
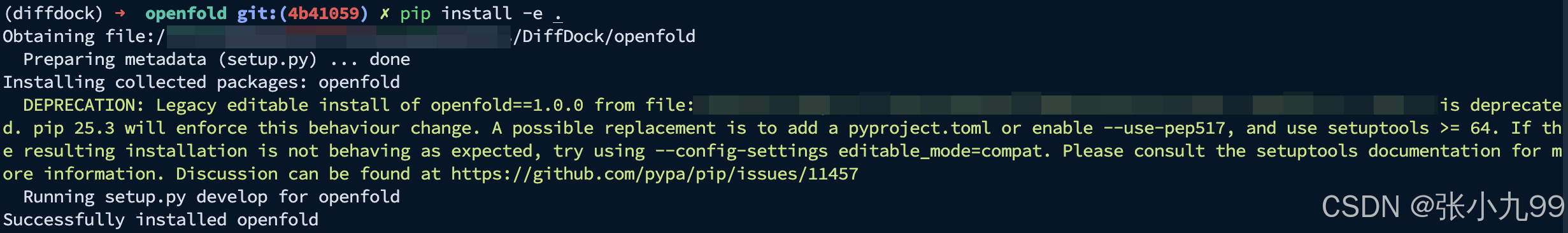
注意:上述第6步和第7步是为了安装OpenFold,可选安装,不安装也可以正常执行DiffDock的推理,安装可能会报错,解决起来比较麻烦。
(二)使用教程
1、输入输入格式与组织方式
1)蛋白输入(两选一)
-
PDB 文件:
--protein_path protein.pdb -
序列字符串:
--protein_sequence GIQSYCTPPYSVLQDPPQPVV
若提供序列,DiffDock 会调用 ESMFold 先折叠出结构,再进行对接。
2)配体输入(两选一)
-
SMILES 字符串:例如
--ligand "COc(cc1)ccc1C#N"用引号包裹,避免 shell 误解析特殊字符。
-
小分子文件(RDKit 可读):
.sdf/.mol2等
例如--ligand ligand.sdf
2、单个复合物预测
只对“一个蛋白 + 一个配体”做预测:
示例 A:PDB + SDF
python -m inference \--config default_inference_args.yaml \--protein_path 0_demo/test.pdb \--ligand 0_demo/test.sdf \--out_dir 0_demo/test_results/示例 B:序列 + SMILES
python -m inference \--config default_inference_args.yaml \--protein_sequence GIQSYCTPPYSVLQDPPQPVV \--ligand "COc(cc1)ccc1C#N" \--out_dir results/single_run_seq
3、批量预测
当你有多组“蛋白—配体”要同时跑时,准备一个 CSV,列名与意义如下:
-
complex_name:结果保存时使用的名称(可留空) -
protein_path:蛋白.pdb路径(若留空则使用protein_sequence) -
ligand_description:SMILES 或小分子文件路径(.sdf/.mol2等) -
protein_sequence:若protein_path为空,则用该序列并通过 ESMFold 折叠
示例 CSV(data/protein_ligand_example.csv):
complex_name,protein_path,ligand_description,protein_sequence
case1,/abs/path/protein1.pdb,/abs/path/lig1.sdf,
case2,, "COc(cc1)ccc1C#N" ,GIQSYCTPPYSVLQDPPQPVV
case3,/abs/path/protein3.pdb,/abs/path/lig3.mol2,
小贴士:
路径建议写绝对路径或相对工程根目录的路径;
SMILES 中含特殊字符,最好加引号;
如果
protein_path留空,务必提供protein_sequence。
python -m inference \--config default_inference_args.yaml \--protein_ligand_csv data/protein_ligand_example.csv \--out_dir results/user_predictions_small
程序会把每个复合物的预测结果写入你指定的 --out_dir 目录。里面通常包含:
-
各复合物的预测构象(对接姿势)相关文件;
-
以及模型的置信度/评分等(具体文件名与格式以版本实现为准)。
目录结构和文件名可能随版本略有差异,但核心思路不变:按
complex_name或输入命名组织,每个复合物都有对应输出。
4、结果分析
这里的连字符 “-” 就是负号。
DiffDock 在保存文件名时直接把数值拼到 confidence 后面:数值为负就会带 “-”,为正就不会带 “+”(例如正 0.12 会写成 confidence0.12.sdf)。
5、常见问题
Q1:没有 GPU,可以跑吗?
可以。提供 .pdb 文件时,CPU 也能跑,但速度明显更慢。
Q2:我只有序列,没有结构怎么办?
用 --protein_sequence,程序会自动通过 ESMFold 折叠,然后继续对接。
Q3:CSV 里到底该用文件还是 SMILES?
两者皆可。字段统一用 ligand_description,RDKit 能读就行;SMILES 记得加引号。
Q4:输出怎么评估?
通常会有置信度/评分等指标;你也可以结合下游分析(如 RMSD、结合位点可视化等)进行更全面的评估。
Q5:如何理解 DiffDock 的置信分数(confidence score)?
-
记
c为最高分姿势(top pose)的置信分数。官方给出一个大致判读区间:-
c > 0:高置信度(模型认为该姿势质量较高)
-
-1.5 < c < 0:中等置信度
-
c < -1.5:低置信度
-
-
这个区间在前提下成立:体系与训练数据分布相似(典型“小分子药样 + 中等大小的单体蛋白/两条链、构象接近复合物的结合态”)。
-
如果是较大的配体、大型蛋白复合体、或者未结合/构象差异大的蛋白,则整体区间需要下调(同样质量的姿势,分数可能更低)。
Q6: DiffDock 会不会给结合亲和力?
不会,DiffDock并不能预测配体与蛋白质的结合亲和力。它预测的是复合物的三维结构,并输出置信度评分。后者是预测质量的衡量指标,即模型对其结合结构预测结果的置信程度。部分合作者观察到该分数与结合亲和力存在相关性(直观而言,若配体未结合则无法形成良好构象),但并非亲和力的直接衡量指标。
研究人员正在开发更优的亲和力预测模型,在此期间建议将DiffDock预测结果与其他工具结合使用,例如对接功能(如GNINA)、分子力学/球形平均势(MM/GBSA)或绝对结合自由能计算。为此,研究人员建议先使用亲和力预测所用的工具/力场对DiffDock的结构预测结果进行松弛处理。
Q7: 适用范围:是否可用于蛋白-蛋白/蛋白-核酸?
-
DiffDock 的设计、训练和测试目标是小分子对蛋白的对接。对于大分子配体(长肽、完整核酸等),模型不推荐使用(即便程序不报错)。
-
如果你是其他类型相互作用:
-
蛋白-蛋白(刚体):看 DiffDock-PP
-
蛋白-蛋白(可柔性):看 AlphaFold-Multimer
-
蛋白-核酸:看 RoseTTAFold2NA
-
参考文档:
https://github.com/gcorso/DiffDock