【数据分析】微生物群落网络构建与模块划分的比较研究:SparCC、Spearman-RAW与Spearman-CLR方法的性能评估
禁止商业或二改转载,仅供自学使用,侵权必究,如需截取部分内容请后台联系作者!
文章目录
-
- 介绍
-
- 背景知识
- 研究方法
- 实验设计
- 加载所需 R 包
- 模拟微生物群落数据
- Ground-truth network from C_pd
- 构建网络(SparCC / Spearman-RAW / Spearman-CLR)
- 划分网络模块(Louvain)
- 网络可视化+网络结构评估指标
-
- 边层面
- 社区层面
- 网络层面
- 网络可视化
- PR / ROC 曲线图
- 桑基图
- 指标条形图
- 打印列联表
- 总结
- 系统信息
介绍
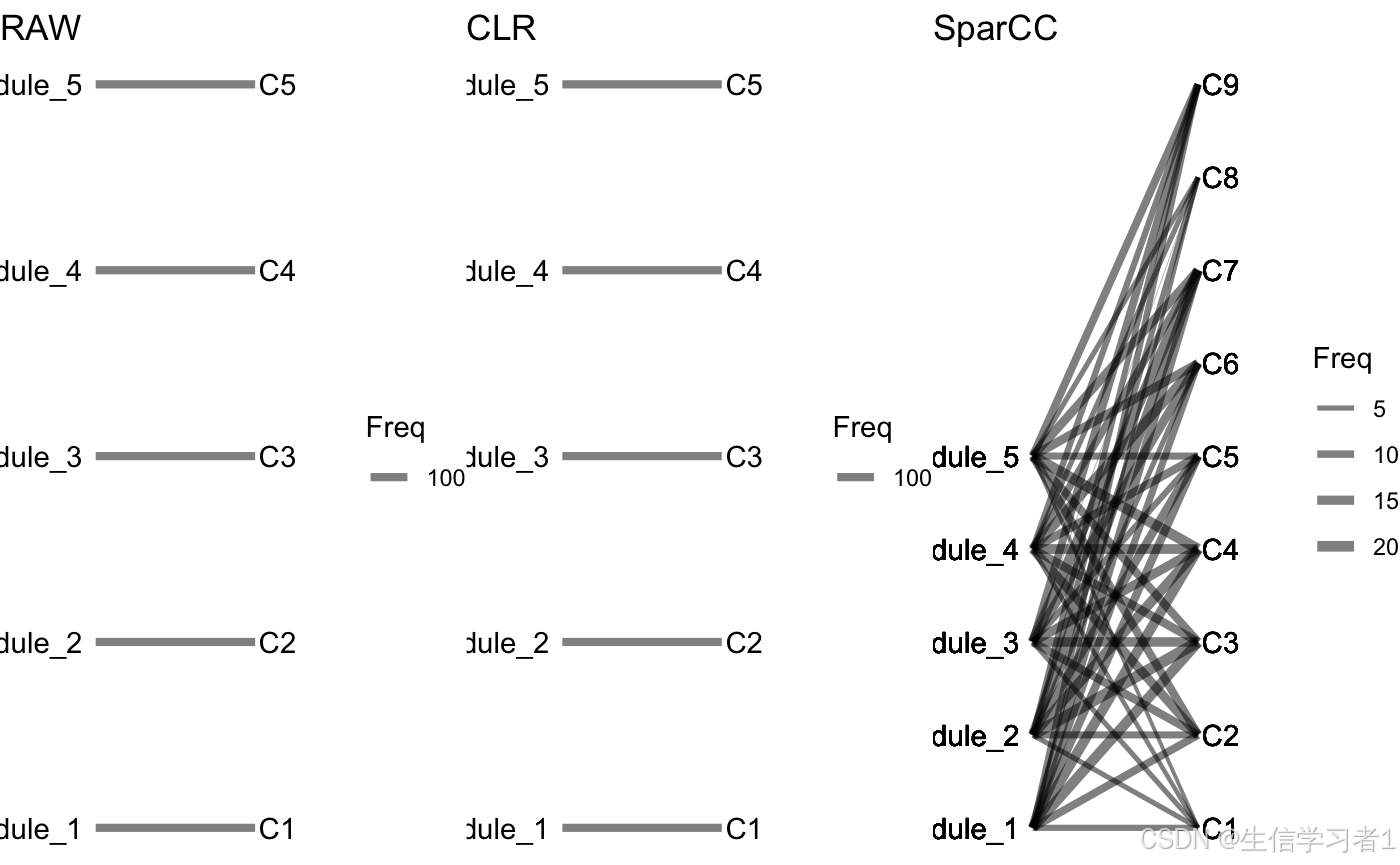
在微生物生态学研究中,微生物群落的网络构建与模块划分是理解微生物相互作用和群落结构的关键步骤。近年来,随着测序技术的发展,微生物群落数据的获取变得更加容易,但如何从这些复杂的数据中提取出有意义的生态信息仍然是一个挑战。本研究旨在通过模拟微生物群落数据,比较三种常用的相关性估计方法——Spearman-RAW、Spearman-CLR和SparCC——在构建微生物群落网络和划分模块时的表现。
背景知识
微生物群落网络构建的核心是估计微生物物种(OTU)之间的相关性。传统的Spearman秩相关系数(Spearman-RAW)直接应用于相对丰度数据,但这种方法可能会受到稀疏性和测序噪声的影响。为了克服这些问题,研究者提出了CLR(Centered Log-Ratio)变换,通过CLR变换后再计算Spearman相关系数(Spearman-CLR),可以更好地处理稀疏数据。此外,SparCC(Sparse Correlations for Compositional data)是一种专门针对组合数据设计的方法,能够更准确地估计微生物之间的稀疏相关性。
研究方法
本研究通过模拟一个具有真实模块结构的微生物群落数据集,生成了包
