R语言使用随机森林对数据进行插补
数据插补的目的是为了恢复数据的完整性,以便后续的数据分析和挖掘工作能够顺利进行。插补方法的选择取决于数据的特点和缺失模式。常见的插补方法包括均值插补、回归插补、多重插补等。均值插补简单易行,但可能会改变数据分布;回归插补考虑了变量之间的关系,但可能引入偏差;多重插补则通过模拟缺失值的不确定性,提供了更合理的统计推断。
既往咱们已经介绍了使用R语言使用mice包多重插补,本期介绍一下如何使用R语言随机森林对数据插补。在R语言中,使用随机森林(Random Forest)进行数据插补是一种有效的处理缺失值的方法,尤其适用于非线性关系和复杂交互的数据,在机器学习中SCI文章插补很常见。最常用的实现方式是通过 missForest 包,它利用随机森林算法迭代地预测每个变量中的缺失值。
咱们先导入R包和数据
library(survival)
library(missForest)
library(VIM)
data<-mgus
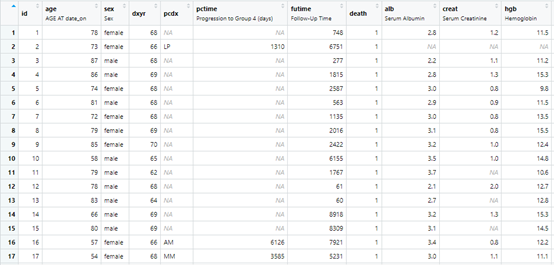
咱们可以看到,数据是存在缺失值的,也可以使用VIM包查看一下
aggr(data, numbers = TRUE, prop = FALSE, sortVar = TRUE)
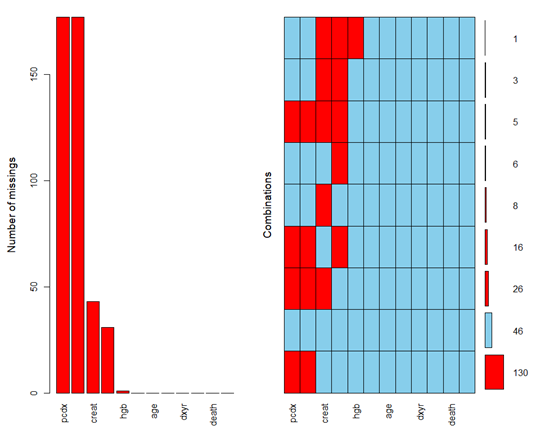
在咱们数据中,sex和pcdx是分类变量,这里要转成因子
data$sex<-as.factor(data$sex)
data$pcdx<-as.factor(data$pcdx)
下面可以正式插补了,就一句话代码
set.seed(123)
data.imp <- missForest(data,ntree = 200, # more trees -> stabler imputationsmaxiter = 5, # outer iterations (default 10; 5 is fine for demo)verbose = FALSE
)
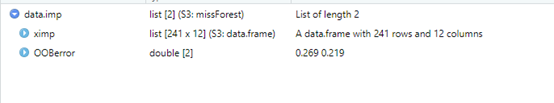
上图可见,只生成一个插补数据,这样就不用纠结用哪个数据插补好了。我们可以把数据提取出来看一下,可以看到已经没有缺失值了。
data.imp2<-data.imp[["ximp"]]
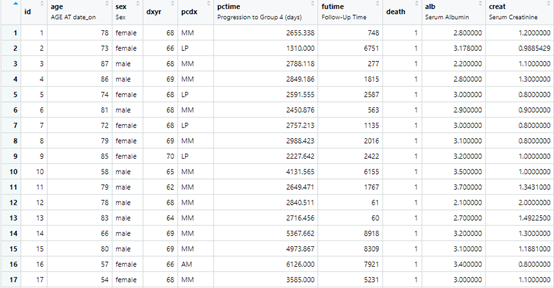
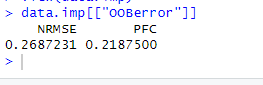
OOBerror这个表示它插补的错误率,自然是越低越好
注意事项
数据类型:missForest 能同时处理数值型和因子型变量。
计算成本:对于大数据集,missForest 可能较慢,建议调整 ntree 和 maxiter。
缺失机制:假设数据为“随机缺失”(MAR)或“完全随机缺失”(MCAR)。
分类变量:确保因子型变量是正确的因子类型,否则会被当作数值处理。
