基于深度学习的胸部 X 光图像肺炎分类系统(一)
本文先重点介绍了过采样的原理是实现。 由于医学数据相对缺乏,过采样是解决数据问题的方法之一。 后续写一篇搭建神经网络的说明
import numpy as np
import matplotlib.pyplot as plt
from sklearn.model_selection import train_test_split
from sklearn.metrics import f1_score, roc_auc_score, roc_curve, confusion_matrix, classification_report
from imblearn.over_sampling import RandomOverSampler
import tensorflow as tf
from keras import layers
from keras import models
# 或者更常用的是直接导入Sequential类
from keras.models import Sequential
from keras.preprocessing.image import ImageDataGenerator
import os
import zipfile
import requests
from tensorflow.python.keras.callbacks import EarlyStopping# 数据加载和预处理
def load_data(train_dir, test_dir, val_dir, img_size=(150, 150), batch_size=32):# 数据增强器 - 仅用于训练集train_datagen = ImageDataGenerator(rescale=1. / 255,rotation_range=10,width_shift_range=0.1,height_shift_range=0.1,shear_range=0.1,zoom_range=0.1,horizontal_flip=True)# 验证集和测试集只需要重新缩放val_test_datagen = ImageDataGenerator(rescale=1. / 255)# 加载训练数据train_generator = train_datagen.flow_from_directory(train_dir,target_size=img_size,batch_size=batch_size,class_mode='binary',classes=['NORMAL', 'PNEUMONIA'],shuffle=True)# 加载验证数据val_generator = val_test_datagen.flow_from_directory(val_dir,target_size=img_size,batch_size=batch_size,class_mode='binary',classes=['NORMAL', 'PNEUMONIA'],shuffle=False)# 加载测试数据test_generator = val_test_datagen.flow_from_directory(test_dir,target_size=img_size,batch_size=batch_size,class_mode='binary',classes=['NORMAL', 'PNEUMONIA'],shuffle=False)return train_generator, val_generator, test_generator# 处理样本不均衡(过采样)
def handle_imbalance(generator):# 提取特征和标签X, y = [], []num_batches = len(generator)# 重置生成器以确保从开始获取数据generator.reset()for i in range(num_batches):batch_x, batch_y = generator.next()X.append(batch_x)y.append(batch_y)X = np.concatenate(X)y = np.concatenate(y)# 打印原始分布print(f"原始样本分布: 正常={np.sum(y == 0)}, 肺炎={np.sum(y == 1)}")# 展平特征用于过采样X_flat = X.reshape(X.shape[0], -1)# 过采样少数类ros = RandomOverSampler(random_state=42)X_resampled, y_resampled = ros.fit_resample(X_flat, y)# 恢复图像形状X_resampled = X_resampled.reshape(-1, *X.shape[1:])print(f"过采样后分布: 正常={np.sum(y_resampled == 0)}, 肺炎={np.sum(y_resampled == 1)}")return X_resampled, y_resampled, y# 构建改进的CNN模型
def build_model(input_shape):model = models.Sequential([# 第一个卷积块layers.Conv2D(32, (3, 3), activation='relu', input_shape=input_shape),layers.BatchNormalization(),layers.MaxPooling2D((2, 2)),layers.Dropout(0.2),# 第二个卷积块layers.Conv2D(64, (3, 3), activation='relu'),layers.BatchNormalization(),layers.MaxPooling2D((2, 2)),layers.Dropout(0.3),# 第三个卷积块layers.Conv2D(128, (3, 3), activation='relu'),layers.BatchNormalization(),layers.MaxPooling2D((2, 2)),layers.Dropout(0.4),# 第四个卷积块layers.Conv2D(256, (3, 3), activation='relu'),layers.BatchNormalization(),layers.MaxPooling2D((2, 2)),layers.Dropout(0.5),# 分类器layers.Flatten(),layers.Dense(512, activation='relu'),layers.BatchNormalization(),layers.Dropout(0.5),layers.Dense(1, activation='sigmoid')])# 使用更稳定的优化器optimizer = tf.keras.optimizers.Adam(learning_rate=0.0001)model.compile(optimizer=optimizer,loss='binary_crossentropy',metrics=['accuracy',tf.keras.metrics.Precision(name='precision'),tf.keras.metrics.Recall(name='recall'),tf.keras.metrics.AUC(name='auc')])return model# 主函数
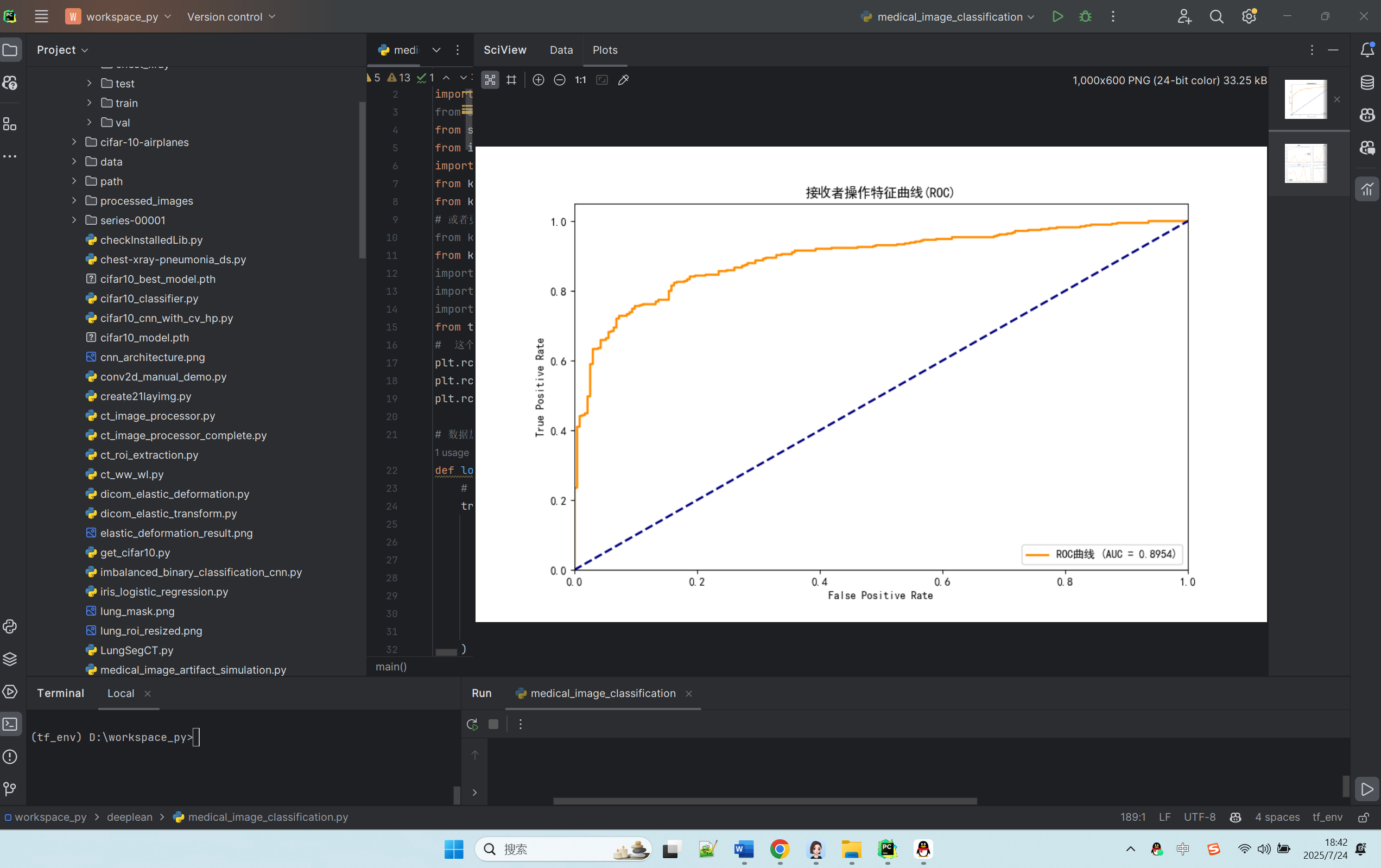
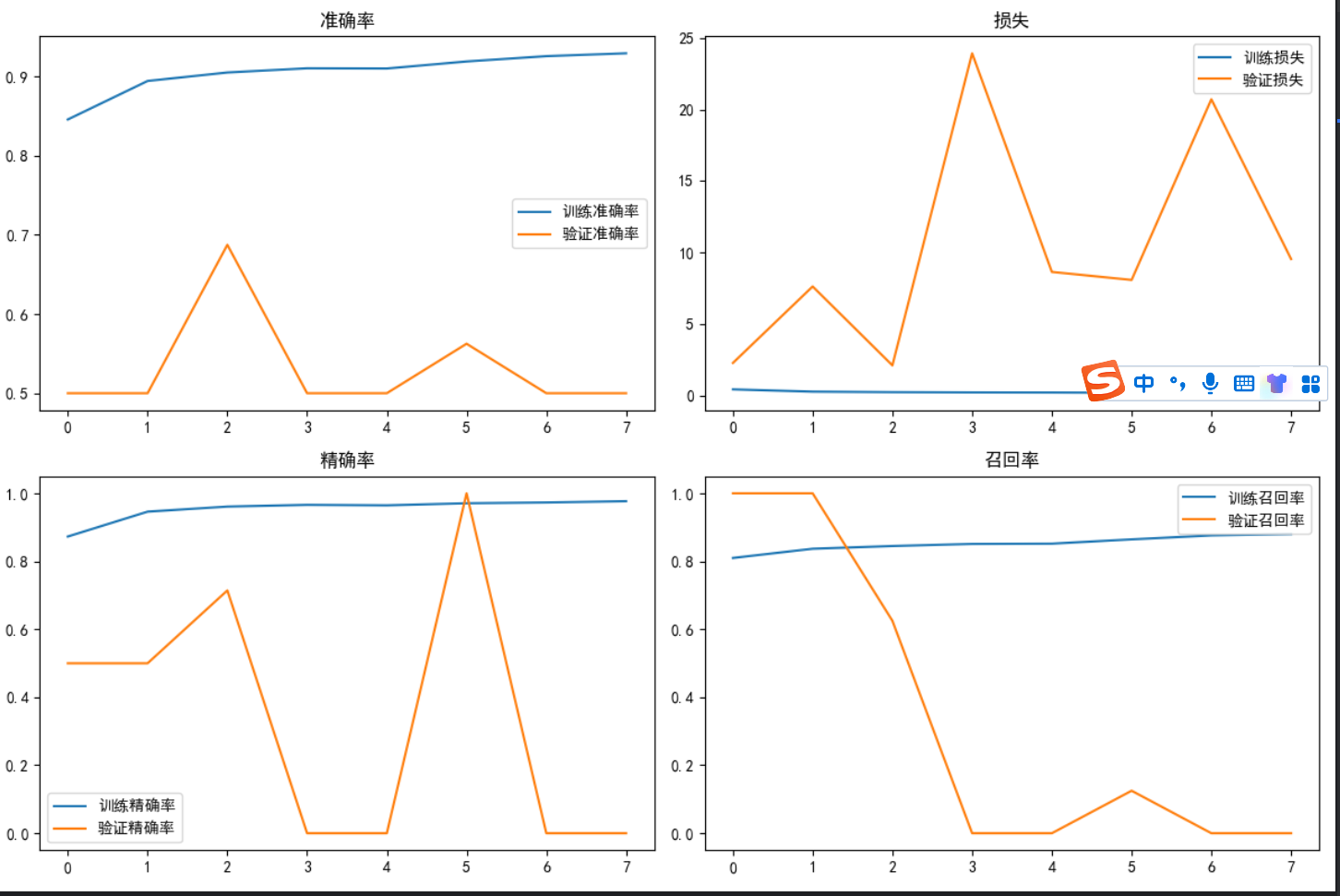
def main():# 假设数据集已经手动下载并解压train_dir = "chest_xray/train"test_dir = "chest_xray/test"val_dir = "chest_xray/val"# 加载数据img_size = (150, 150)batch_size = 32train_generator, val_generator, test_generator = load_data(train_dir, test_dir, val_dir, img_size, batch_size)# 处理样本不均衡X_train, y_train_resampled, y_train_original = handle_imbalance(train_generator)# 计算类别权重(基于原始分布)n_normal = np.sum(y_train_original == 0)n_pneumonia = np.sum(y_train_original == 1)total = n_normal + n_pneumoniaweight_for_normal = (1 / n_normal) * (total / 2.0)weight_for_pneumonia = (1 / n_pneumonia) * (total / 2.0)class_weights = {0: weight_for_normal, 1: weight_for_pneumonia}print(f"类别权重: 正常={weight_for_normal:.2f}, 肺炎={weight_for_pneumonia:.2f}")# 构建模型model = build_model((*img_size, 3))model.summary()# 提前停止回调early_stopping = EarlyStopping(monitor='val_loss',patience=5,restore_best_weights=True,verbose=1)# 训练模型history = model.fit(X_train, y_train_resampled,epochs=30,batch_size=32,validation_data=val_generator,class_weight=class_weights,callbacks=[early_stopping],verbose=1)# 评估模型 - 使用完整测试集test_generator.reset()test_steps = len(test_generator)test_results = model.evaluate(test_generator, steps=test_steps, verbose=1)print("\n测试集评估结果:")print(f"准确率: {test_results[1]:.4f}")print(f"精确率: {test_results[2]:.4f}")print(f"召回率: {test_results[3]:.4f}")print(f"AUC: {test_results[4]:.4f}")# 获取测试集所有预测结果test_generator.reset()y_true = []y_pred_prob = []for i in range(test_steps):batch_x, batch_y = test_generator.next()y_true.extend(batch_y)batch_pred = model.predict(batch_x, verbose=0).ravel()y_pred_prob.extend(batch_pred)y_true = np.array(y_true)y_pred_prob = np.array(y_pred_prob)y_pred = (y_pred_prob > 0.5).astype(int)# 计算额外指标f1 = f1_score(y_true, y_pred)auc = roc_auc_score(y_true, y_pred_prob)print(f"\nF1-score: {f1:.4f}")print(f"AUC-ROC: {auc:.4f}")# 分类报告print("\n分类报告:")print(classification_report(y_true, y_pred, target_names=['NORMAL', 'PNEUMONIA']))# 混淆矩阵cm = confusion_matrix(y_true, y_pred)print("混淆矩阵:")print(cm)# 绘制ROC曲线fpr, tpr, _ = roc_curve(y_true, y_pred_prob)plt.figure(figsize=(10, 6))plt.plot(fpr, tpr, color='darkorange', lw=2, label=f'ROC曲线 (AUC = {auc:.4f})')plt.plot([0, 1], [0, 1], color='navy', lw=2, linestyle='--')plt.xlim([0.0, 1.0])plt.ylim([0.0, 1.05])plt.xlabel('False Positive Rate')plt.ylabel('True Positive Rate')plt.title('接收者操作特征曲线(ROC)')plt.legend(loc="lower right")plt.savefig('roc_curve.png', dpi=300)plt.show()# 绘制训练历史plt.figure(figsize=(12, 8))plt.subplot(2, 2, 1)plt.plot(history.history['accuracy'], label='训练准确率')plt.plot(history.history['val_accuracy'], label='验证准确率')plt.title('准确率')plt.legend()plt.subplot(2, 2, 2)plt.plot(history.history['loss'], label='训练损失')plt.plot(history.history['val_loss'], label='验证损失')plt.title('损失')plt.legend()plt.subplot(2, 2, 3)plt.plot(history.history['precision'], label='训练精确率')plt.plot(history.history['val_precision'], label='验证精确率')plt.title('精确率')plt.legend()plt.subplot(2, 2, 4)plt.plot(history.history['recall'], label='训练召回率')plt.plot(history.history['val_recall'], label='验证召回率')plt.title('召回率')plt.legend()plt.tight_layout()plt.savefig('training_history.png', dpi=300)plt.show()if __name__ == "__main__":main()运行情况:
D:\ProgramData\anaconda3\envs\tf_env\python.exe D:\workspace_py\deeplean\medical_image_classification.py
Found 5216 images belonging to 2 classes.
Found 16 images belonging to 2 classes.
Found 624 images belonging to 2 classes.
原始样本分布: 正常=1341, 肺炎=3875
过采样后分布: 正常=3875, 肺炎=3875
类别权重: 正常=1.94, 肺炎=0.67
2025-07-24 09:21:36.980575: I tensorflow/core/platform/cpu_feature_guard.cc:182] This TensorFlow binary is optimized to use available CPU instructions in performance-critical operations.
To enable the following instructions: SSE SSE2 SSE3 SSE4.1 SSE4.2 AVX AVX2 FMA, in other operations, rebuild TensorFlow with the appropriate compiler flags.
Model: "sequential"
_________________________________________________________________Layer (type) Output Shape Param #
=================================================================conv2d (Conv2D) (None, 148, 148, 32) 896 batch_normalization (Batch (None, 148, 148, 32) 128 Normalization) max_pooling2d (MaxPooling2 (None, 74, 74, 32) 0 D) dropout (Dropout) (None, 74, 74, 32) 0 conv2d_1 (Conv2D) (None, 72, 72, 64) 18496 batch_normalization_1 (Bat (None, 72, 72, 64) 256 chNormalization) max_pooling2d_1 (MaxPoolin (None, 36, 36, 64) 0 g2D) dropout_1 (Dropout) (None, 36, 36, 64) 0 conv2d_2 (Conv2D) (None, 34, 34, 128) 73856 batch_normalization_2 (Bat (None, 34, 34, 128) 512 chNormalization) max_pooling2d_2 (MaxPoolin (None, 17, 17, 128) 0 g2D) dropout_2 (Dropout) (None, 17, 17, 128) 0 conv2d_3 (Conv2D) (None, 15, 15, 256) 295168 batch_normalization_3 (Bat (None, 15, 15, 256) 1024 chNormalization) max_pooling2d_3 (MaxPoolin (None, 7, 7, 256) 0 g2D) dropout_3 (Dropout) (None, 7, 7, 256) 0 flatten (Flatten) (None, 12544) 0 dense (Dense) (None, 512) 6423040 batch_normalization_4 (Bat (None, 512) 2048 chNormalization) dropout_4 (Dropout) (None, 512) 0 dense_1 (Dense) (None, 1) 513 =================================================================
Total params: 6815937 (26.00 MB)
Trainable params: 6813953 (25.99 MB)
Non-trainable params: 1984 (7.75 KB)
_________________________________________________________________
Epoch 1/30
243/243 [==============================] - 134s 541ms/step - loss: 0.4104 - accuracy: 0.8552 - precision: 0.8934 - recall: 0.8067 - auc: 0.9327 - val_loss: 8.2165 - val_accuracy: 0.5000 - val_precision: 0.5000 - val_recall: 1.0000 - val_auc: 0.5000
Epoch 2/30
243/243 [==============================] - 132s 545ms/step - loss: 0.2674 - accuracy: 0.8968 - precision: 0.9505 - recall: 0.8372 - auc: 0.9627 - val_loss: 9.0454 - val_accuracy: 0.5000 - val_precision: 0.5000 - val_recall: 1.0000 - val_auc: 0.5000
Epoch 3/30
243/243 [==============================] - 144s 594ms/step - loss: 0.2277 - accuracy: 0.9112 - precision: 0.9674 - recall: 0.8511 - auc: 0.9713 - val_loss: 2.9472 - val_accuracy: 0.6250 - val_precision: 0.5833 - val_recall: 0.8750 - val_auc: 0.6719
Epoch 4/30
243/243 [==============================] - 133s 545ms/step - loss: 0.2208 - accuracy: 0.9165 - precision: 0.9676 - recall: 0.8619 - auc: 0.9735 - val_loss: 1.9898 - val_accuracy: 0.6250 - val_precision: 0.5833 - val_recall: 0.8750 - val_auc: 0.7188
Epoch 5/30
243/243 [==============================] - 128s 525ms/step - loss: 0.2024 - accuracy: 0.9228 - precision: 0.9688 - recall: 0.8738 - auc: 0.9754 - val_loss: 2.0641 - val_accuracy: 0.6875 - val_precision: 0.6154 - val_recall: 1.0000 - val_auc: 0.6875
Epoch 6/30
243/243 [==============================] - 133s 548ms/step - loss: 0.1924 - accuracy: 0.9227 - precision: 0.9691 - recall: 0.8733 - auc: 0.9783 - val_loss: 12.3177 - val_accuracy: 0.5000 - val_precision: 0.0000e+00 - val_recall: 0.0000e+00 - val_auc: 0.3984
Epoch 7/30
243/243 [==============================] - 128s 528ms/step - loss: 0.1849 - accuracy: 0.9303 - precision: 0.9728 - recall: 0.8854 - auc: 0.9796 - val_loss: 3.6844 - val_accuracy: 0.6250 - val_precision: 0.5714 - val_recall: 1.0000 - val_auc: 0.7500
Epoch 8/30
243/243 [==============================] - 129s 531ms/step - loss: 0.1561 - accuracy: 0.9334 - precision: 0.9784 - recall: 0.8865 - auc: 0.9849 - val_loss: 2.7532 - val_accuracy: 0.5000 - val_precision: 0.0000e+00 - val_recall: 0.0000e+00 - val_auc: 0.3438
Epoch 9/30
243/243 [==============================] - 128s 529ms/step - loss: 0.1646 - accuracy: 0.9356 - precision: 0.9739 - recall: 0.8952 - auc: 0.9839 - val_loss: 1.7896 - val_accuracy: 0.6875 - val_precision: 0.6364 - val_recall: 0.8750 - val_auc: 0.7031
Epoch 10/30
243/243 [==============================] - 125s 515ms/step - loss: 0.1542 - accuracy: 0.9385 - precision: 0.9778 - recall: 0.8973 - auc: 0.9847 - val_loss: 2.2632 - val_accuracy: 0.6250 - val_precision: 1.0000 - val_recall: 0.2500 - val_auc: 0.6797
Epoch 11/30
243/243 [==============================] - 130s 534ms/step - loss: 0.1450 - accuracy: 0.9432 - precision: 0.9810 - recall: 0.9040 - auc: 0.9862 - val_loss: 5.9280 - val_accuracy: 0.5000 - val_precision: 0.0000e+00 - val_recall: 0.0000e+00 - val_auc: 0.3906
Epoch 12/30
243/243 [==============================] - 129s 532ms/step - loss: 0.1435 - accuracy: 0.9422 - precision: 0.9793 - recall: 0.9035 - auc: 0.9866 - val_loss: 2.1806 - val_accuracy: 0.5000 - val_precision: 0.0000e+00 - val_recall: 0.0000e+00 - val_auc: 0.6562
Epoch 13/30
243/243 [==============================] - 132s 543ms/step - loss: 0.1348 - accuracy: 0.9475 - precision: 0.9793 - recall: 0.9143 - auc: 0.9876 - val_loss: 0.9206 - val_accuracy: 0.7500 - val_precision: 0.8333 - val_recall: 0.6250 - val_auc: 0.8203
Epoch 14/30
243/243 [==============================] - 133s 547ms/step - loss: 0.1261 - accuracy: 0.9520 - precision: 0.9853 - recall: 0.9177 - auc: 0.9893 - val_loss: 0.4002 - val_accuracy: 0.8125 - val_precision: 0.7778 - val_recall: 0.8750 - val_auc: 0.9062
Epoch 15/30
243/243 [==============================] - 129s 532ms/step - loss: 0.1259 - accuracy: 0.9507 - precision: 0.9821 - recall: 0.9182 - auc: 0.9890 - val_loss: 0.6035 - val_accuracy: 0.6875 - val_precision: 0.7143 - val_recall: 0.6250 - val_auc: 0.8281
Epoch 16/30
243/243 [==============================] - 128s 527ms/step - loss: 0.1224 - accuracy: 0.9525 - precision: 0.9851 - recall: 0.9190 - auc: 0.9896 - val_loss: 1.0697 - val_accuracy: 0.6875 - val_precision: 1.0000 - val_recall: 0.3750 - val_auc: 0.8750
Epoch 17/30
243/243 [==============================] - 124s 509ms/step - loss: 0.1145 - accuracy: 0.9556 - precision: 0.9838 - recall: 0.9265 - auc: 0.9907 - val_loss: 0.3882 - val_accuracy: 0.8125 - val_precision: 0.7778 - val_recall: 0.8750 - val_auc: 0.9062
Epoch 18/30
243/243 [==============================] - 122s 503ms/step - loss: 0.1106 - accuracy: 0.9583 - precision: 0.9863 - recall: 0.9295 - auc: 0.9911 - val_loss: 1.0384 - val_accuracy: 0.6250 - val_precision: 1.0000 - val_recall: 0.2500 - val_auc: 0.8594
Epoch 19/30
243/243 [==============================] - 124s 508ms/step - loss: 0.1084 - accuracy: 0.9561 - precision: 0.9852 - recall: 0.9262 - auc: 0.9923 - val_loss: 2.7370 - val_accuracy: 0.5000 - val_precision: 0.0000e+00 - val_recall: 0.0000e+00 - val_auc: 0.8125
Epoch 20/30
243/243 [==============================] - 124s 508ms/step - loss: 0.1044 - accuracy: 0.9574 - precision: 0.9863 - recall: 0.9277 - auc: 0.9920 - val_loss: 1.6090 - val_accuracy: 0.6250 - val_precision: 0.5714 - val_recall: 1.0000 - val_auc: 0.8828
Epoch 21/30
243/243 [==============================] - 123s 508ms/step - loss: 0.0952 - accuracy: 0.9634 - precision: 0.9875 - recall: 0.9386 - auc: 0.9932 - val_loss: 2.9180 - val_accuracy: 0.5625 - val_precision: 0.5333 - val_recall: 1.0000 - val_auc: 0.6875
Epoch 22/30
243/243 [==============================] - ETA: 0s - loss: 0.0913 - accuracy: 0.9635 - precision: 0.9891 - recall: 0.9373 - auc: 0.9937Restoring model weights from the end of the best epoch.
243/243 [==============================] - 124s 508ms/step - loss: 0.0913 - accuracy: 0.9635 - precision: 0.9891 - recall: 0.9373 - auc: 0.9937 - val_loss: 1.3448 - val_accuracy: 0.5625 - val_precision: 1.0000 - val_recall: 0.1250 - val_auc: 0.8281
Epoch 00022: early stopping
20/20 [==============================] - 5s 221ms/step - loss: 0.2983 - accuracy: 0.8990 - precision: 0.9247 - recall: 0.9128 - auc: 0.9554测试集评估结果:
准确率: 0.8990
精确率: 0.9247
召回率: 0.9128
AUC: 0.9554F1-score: 0.9187
AUC-ROC: 0.9568分类报告:precision recall f1-score supportNORMAL 0.86 0.88 0.87 234PNEUMONIA 0.92 0.91 0.92 390accuracy 0.90 624macro avg 0.89 0.89 0.89 624
weighted avg 0.90 0.90 0.90 624混淆矩阵:
[[205 29][ 34 356]]概述
这段代码是一个用于胸部 X 光图像肺炎分类的深度学习项目。
导入必要的库
import numpy as np import matplotlib.pyplot as plt from sklearn.model_selection import train_test_split from sklearn.metrics import f1_score, roc_auc_score, roc_curve, confusion_matrix, classification_report from imblearn.over_sampling import RandomOverSampler import tensorflow as tf from keras import layers from keras import models from keras.models import Sequential from keras.preprocessing.image import ImageDataGenerator import os import zipfile import requests from tensorflow.python.keras.callbacks import EarlyStopping |
- numpy 和 matplotlib.pyplot:用于数值计算和数据可视化。
- sklearn 相关模块:用于数据划分、模型评估等机器学习任务。
- imblearn 模块:用于处理不平衡数据集的过采样。
- tensorflow 和 keras 相关模块:用于构建和训练深度学习模型。
- os、zipfile、requests:用于文件操作和网络请求
可以这样理解:
- numpy和matplotlib:计算器和画图板,用来算数据、画结果图。
- sklearn:机器学习小助手,帮着评估电脑学得好不好。
- tensorflow和keras:深度学习的 “大脑”,负责让电脑学会识别图片。
- 其他工具:帮着读图片、处理文件的小帮手。
数据加载和预处理函数
def load_data(train_dir, test_dir, val_dir, img_size=(150, 150), batch_size=32): train_datagen = ImageDataGenerator( rescale=1. / 255, rotation_range=10, width_shift_range=0.1, height_shift_range=0.1, shear_range=0.1, zoom_range=0.1, horizontal_flip=True ) val_test_datagen = ImageDataGenerator(rescale=1. / 255) train_generator = train_datagen.flow_from_directory( train_dir, target_size=img_size, batch_size=batch_size, class_mode='binary', classes=['NORMAL', 'PNEUMONIA'], shuffle=True ) val_generator = val_test_datagen.flow_from_directory( val_dir, target_size=img_size, batch_size=batch_size, class_mode='binary', classes=['NORMAL', 'PNEUMONIA'], shuffle=False ) test_generator = val_test_datagen.flow_from_directory( test_dir, target_size=img_size, batch_size=batch_size, class_mode='binary', classes=['NORMAL', 'PNEUMONIA'], shuffle=False ) return train_generator, val_generator, test_generator |
- 定义了一个 load_data 函数,用于加载和预处理胸部 X 光图像数据。
- 使用 ImageDataGenerator 进行数据增强,包括旋转、平移、缩放等操作,以增加训练数据的多样性。
- 分别为训练集、验证集和测试集创建数据生成器,从指定目录加载图像数据,并将图像大小调整为 img_size,批量大小为 batch_size,类别模式为二分类('NORMAL' 和 'PNEUMONIA')。
可以这样说:
- 数据分三类:train(训练用,让电脑学的)、test(考试用,最后打分的)、val(练习用,学的时候随时纠错的)。
- 给图片做 “预处理”:比如把图片统一改成 150x150 大小(方便电脑处理),训练时还会故意旋转、缩放图片(让电脑见多识广,别认死理)。
- 告诉电脑:“NORMAL” 是正常,“PNEUMONIA” 是肺炎,这是两类。
处理样本不均衡函数
现实中,肺炎的片子可能比正常的多很多(比如 100 张肺炎 vs 20 张正常),电脑会学偏。这个函数就是 “找平”:
- 把少的那类(比如正常片)复制一些,让两类数量差不多(比如都变成 100 张)。
这样电脑学的时候,不会因为某类片子多看就偏心。
def handle_imbalance(generator): X, y = [], [] num_batches = len(generator) generator.reset() for i in range(num_batches): batch_x, batch_y = generator.next() X.append(batch_x) y.append(batch_y) X = np.concatenate(X) y = np.concatenate(y) print(f"原始样本分布: 正常={np.sum(y == 0)}, 肺炎={np.sum(y == 1)}") X_flat = X.reshape(X.shape[0], -1) ros = RandomOverSampler(random_state=42) X_resampled, y_resampled = ros.fit_resample(X_flat, y) X_resampled = X_resampled.reshape(-1, *X.shape[1:]) print(f"过采样后分布: 正常={np.sum(y_resampled == 0)}, 肺炎={np.sum(y_resampled == 1)}") return X_resampled, y_resampled, y |
- 定义了一个 handle_imbalance 函数,用于处理样本不均衡问题。
- 从数据生成器中提取特征和标签,并将其展平用于过采样。
- 使用 RandomOverSampler 对少数类进行过采样,以平衡数据集。
构建改进的 CNN 模型函数
def build_model(input_shape): model = models.Sequential([ layers.Conv2D(32, (3, 3), activation='relu', input_shape=input_shape), layers.BatchNormalization(), layers.MaxPooling2D((2, 2)), layers.Dropout(0.2), layers.Conv2D(64, (3, 3), activation='relu'), layers.BatchNormalization(), layers.MaxPooling2D((2, 2)), layers.Dropout(0.3), layers.Conv2D(128, (3, 3), activation='relu'), layers.BatchNormalization(), layers.MaxPooling2D((2, 2)), layers.Dropout(0.4), layers.Conv2D(256, (3, 3), activation='relu'), layers.BatchNormalization(), layers.MaxPooling2D((2, 2)), layers.Dropout(0.5), layers.Flatten(), layers.Dense(512, activation='relu'), layers.BatchNormalization(), layers.Dropout(0.5), layers.Dense(1, activation='sigmoid') ]) optimizer = tf.keras.optimizers.Adam(learning_rate=0.0001) model.compile( optimizer=optimizer, loss='binary_crossentropy', metrics=[ 'accuracy', tf.keras.metrics.Precision(name='precision'), tf.keras.metrics.Recall(name='recall'), tf.keras.metrics.AUC(name='auc') ] ) return model |
- 定义了一个 build_model 函数,用于构建一个改进的卷积神经网络(CNN)模型。
- 模型包含多个卷积块,每个卷积块由卷积层、批量归一化层、最大池化层和 dropout 层组成。
- 最后是全连接层和输出层,使用 sigmoid 激活函数进行二分类。
- 模型使用 Adam 优化器,学习率为 0.0001,损失函数为 binary_crossentropy,评估指标包括准确率、精确率、召回率和 AUC。
主函数
def main(): train_dir = "chest_xray/train" test_dir = "chest_xray/test" val_dir = "chest_xray/val" img_size = (150, 150) batch_size = 32 train_generator, val_generator, test_generator = load_data(train_dir, test_dir, val_dir, img_size, batch_size) X_train, y_train_resampled, y_train_original = handle_imbalance(train_generator) n_normal = np.sum(y_train_original == 0) n_pneumonia = np.sum(y_train_original == 1) total = n_normal + n_pneumonia weight_for_normal = (1 / n_normal) * (total / 2.0) weight_for_pneumonia = (1 / n_pneumonia) * (total / 2.0) class_weights = {0: weight_for_normal, 1: weight_for_pneumonia} print(f"类别权重: 正常={weight_for_normal:.2f}, 肺炎={weight_for_pneumonia:.2f}") model = build_model((*img_size, 3)) model.summary() early_stopping = EarlyStopping( monitor='val_loss', patience=5, restore_best_weights=True, verbose=1 ) history = model.fit( X_train, y_train_resampled, epochs=30, batch_size=32, validation_data=val_generator, class_weight=class_weights, callbacks=[early_stopping], verbose=1 ) test_generator.reset() test_steps = len(test_generator) test_results = model.evaluate(test_generator, steps=test_steps, verbose=1) print("\n测试集评估结果:") print(f"准确率: {test_results[1]:.4f}") print(f"精确率: {test_results[2]:.4f}") print(f"召回率: {test_results[3]:.4f}") print(f"AUC: {test_results[4]:.4f}") test_generator.reset() y_true = [] y_pred_prob = [] for i in range(test_steps): batch_x, batch_y = test_generator.next() y_true.extend(batch_y) batch_pred = model.predict(batch_x, verbose=0).ravel() y_pred_prob.extend(batch_pred) y_true = np.array(y_true) y_pred_prob = np.array(y_pred_prob) y_pred = (y_pred_prob > 0.5).astype(int) f1 = f1_score(y_true, y_pred) auc = roc_auc_score(y_true, y_pred_prob) print(f"\nF1-score: {f1:.4f}") print(f"AUC-ROC: {auc:.4f}") print("\n分类报告:") print(classification_report(y_true, y_pred, target_names=['NORMAL', 'PNEUMONIA'])) cm = confusion_matrix(y_true, y_pred) print("混淆矩阵:") print(cm) fpr, tpr, _ = roc_curve(y_true, y_pred_prob) plt.figure(figsize=(10, 6)) plt.plot(fpr, tpr, color='darkorange', lw=2, label=f'ROC曲线 (AUC = {auc:.4f})') plt.plot([0, 1], [0, 1], color='navy', lw=2, linestyle='--') plt.xlim([0.0, 1.0]) plt.ylim([0.0, 1.05]) plt.xlabel('False Positive Rate') plt.ylabel('True Positive Rate') plt.title('接收者操作特征曲线(ROC)') plt.legend(loc="lower right") plt.savefig('roc_curve.png', dpi=300) plt.show() plt.figure(figsize=(12, 8)) plt.subplot(2, 2, 1) plt.plot(history.history['accuracy'], label='训练准确率') plt.plot(history.history['val_accuracy'], label='验证准确率') plt.title('准确率') plt.legend() plt.subplot(2, 2, 2) plt.plot(history.history['loss'], label='训练损失') plt.plot(history.history['val_loss'], label='验证损失') plt.title('损失') plt.legend() plt.subplot(2, 2, 3) plt.plot(history.history['precision'], label='训练精确率') plt.plot(history.history['val_precision'], label='验证精确率') plt.title('精确率') plt.legend() plt.subplot(2, 2, 4) plt.plot(history.history['recall'], label='训练召回率') plt.plot(history.history['val_recall'], label='验证召回率') plt.title('召回率') plt.legend() plt.tight_layout() plt.savefig('training_history.png', dpi=300) plt.show() if __name__ == "__main__": main() |
- 定义了一个 main 函数,作为程序的入口。
- 指定了训练集、测试集和验证集的目录。
- 调用 load_data 函数加载数据,调用 handle_imbalance 函数处理样本不均衡问题。
- 计算类别权重,用于处理样本不均衡问题。
- 调用 build_model 函数构建模型,并打印模型摘要。
- 使用 EarlyStopping 回调函数,在验证损失不再下降时停止训练,并恢复最佳权重。
- 训练模型,评估模型在测试集上的性能,并计算各种评估指标。
- 绘制 ROC 曲线和训练历史图表,以可视化模型的性能。
总的来说,这段代码实现了一个完整的胸部 X 光图像肺炎分类系统,包括数据加载、预处理、模型构建、训练和评估等步骤。
数据生成器generator:高效助力电脑学习X光片
数据生成器(generator)你可以理解成一个 “自动上菜的服务员”,专门给电脑 “喂” 数据的。咱们用大白话讲:
为啥需要这个 “服务员”?
如果你的电脑要学 10000 张 X 光片,这些片子加起来可能有几个 G 大。如果一下子全塞进电脑内存(相当于 “一口气把所有菜都端上桌”),内存可能装不下,电脑会变慢甚至卡死。
这时候就需要 “数据生成器” 这个服务员:它不一次性把所有片子都拿出来,而是一批一批地给(比如一次给 32 张),电脑学完这 32 张,再给下 32 张,循环往复,直到学完所有。
数据生成器具体干了啥?
- 按批次取数据:比如你设定 “一批 32 张”,它就每次从文件夹里挑 32 张 X 光片。
- 顺便做预处理:拿片子的时候,自动把它们改成统一大小(比如 150x150),或者旋转、缩放一下(增加数据多样性,让电脑学得更灵活)。
- 给片子贴标签:每张片子对应的 “正常” 或 “肺炎” 标签,它也会一起拿给电脑,不用你手动对应。
def load_data(train_dir, test_dir, val_dir, img_size=(150, 150), batch_size=32)
def load_data(train_dir, test_dir, val_dir, img_size=(150, 150), batch_size=32):# 数据增强器 - 仅用于训练集train_datagen = ImageDataGenerator(rescale=1. / 255,rotation_range=10,width_shift_range=0.1,height_shift_range=0.1,shear_range=0.1,zoom_range=0.1,horizontal_flip=True)# 验证集和测试集只需要重新缩放val_test_datagen = ImageDataGenerator(rescale=1. / 255)# 加载训练数据train_generator = train_datagen.flow_from_directory(train_dir,target_size=img_size,batch_size=batch_size,class_mode='binary',classes=['NORMAL', 'PNEUMONIA'],shuffle=True)# 加载验证数据val_generator = val_test_datagen.flow_from_directory(val_dir,target_size=img_size,batch_size=batch_size,class_mode='binary',classes=['NORMAL', 'PNEUMONIA'],shuffle=False)# 加载测试数据test_generator = val_test_datagen.flow_from_directory(test_dir,target_size=img_size,batch_size=batch_size,class_mode='binary',classes=['NORMAL', 'PNEUMONIA'],shuffle=False)return train_generator, val_generator, test_generator在 load_data 函数里,我们创建了 train_generator(训练用的服务员)、val_generator(验证用的服务员):
train_generator = train_datagen.flow_from_directory(...) |
这里的 flow_from_directory 就是 “从文件夹里取数据” 的意思,这个函数会生成一个 “服务员”,你调用它的 .next() 方法,它就给你一批处理好的片子和标签:
batch_x, batch_y = generator.next() # 服务员,来一批32张片子和标签~ |
数据生成器就像:
- 一个 “自助餐服务员”,每次给你端一小盘菜(一批数据),吃完再端,不浪费空间。
- 一个 “预处理小助手”,端菜前还会帮你把菜切好、摆盘(统一尺寸、增强数据)。
有了它,电脑就能高效地 “吃” 数据、学知识,不会因为数据太多噎着(内存不足)。
Python 中对 X 光片标签的统计操作
print(f"原始样本分布: 正常={np.sum(y == 0)}, 肺炎={np.sum(y == 1)}")
这里的 y 不是集合,而是一个 “标签列表”(更准确地说,是 NumPy 数组),里面装的全是 0 和 1:0 代表 “正常” 的 X 光片,1 代表 “肺炎” 的 X 光片。
咱们用大白话解释这两行代码:
1. 先理解 y 是什么
假设我们有 10 张 X 光片,对应的标签 y 可能是这样的:
y = [0, 1, 1, 0, 1, 1, 1, 0, 1, 1]
(意思是:第 1、4、8 张是正常片,其余 7 张是肺炎片)
2. np.sum(y == 0) 是什么意思?
- y == 0:先检查 y 里的每个元素是不是 0,会得到一个 “真假列表”。
比如上面的 y 会变成:
[True, False, False, True, False, False, False, True, False, False]
(True 表示这个位置是 0,False 表示不是)
- np.sum(...):把 “真假列表” 里的 True 算成 1,False 算成 0,然后求和。
上面的例子里,True 有 3 个,所以 np.sum(y == 0) = 3
(意思是:正常的片子有 3 张)
3. np.sum(y == 1) 同理
- y == 1:检查每个元素是不是 1,得到另一个 “真假列表”:
[False, True, True, False, True, True, True, False, True, True]
- 求和后 np.sum(y == 1) = 7
(意思是:肺炎的片子有 7 张)
总结
这两行代码就是在 “数数量”:
np.sum(y == 0) → 数清楚 “正常” 的片子有多少张;
np.sum(y == 1) → 数清楚 “肺炎” 的片子有多少张。
最后用 print 显示出来,就能直观看到两类片子的数量是否平衡啦~
将 X 中的 X 光片从正方形重塑为长条形以适配处理工具
这句话的作用是把所有 X 光片从 “正方形” 变成 “长条形”,方便后面的工具处理。咱们用大白话解释:
先看 X 是什么
X 里装的是一堆 X 光 X 光片,假设是 3200 张,每张都是 150x150 像素的彩色图(3 个颜色通道)。
它的形状可以理解为:(3200, 150, 150, 3)
翻译过来就是:3200 张图,每张图高 150、宽 150、3 个颜色层。
X.reshape(...) 是在 “重塑形状”
X.reshape(X.shape[0], -1) 里的参数:
- X.shape[0]:表示 “保持样本数量不变”(还是 3200 张)。
- -1:表示 “自动计算剩下的长度”(不用我们手动算)。
变成 “长条形” 后是什么样?
原来每张图是 150x150x3 的 “方块”,展开成一条线的长度是:150×150×3 = 67500。
所以重塑后,X_flat 的形状是 (3200, 67500):
- 3200 张图不变;
- 每张图从 “150x150x3 的方块” 变成了 “67500 个数字排成的长条”。
为啥要这么做?
因为后面用来 “复制样本” 的工具(RandomOverSampler)比较 “死板”,只认这种 “一行代表一个样本” 的长条形数据,不认原来的 “方块形” 图片。
这一步就相当于把 “魔方” 拆成 “一条直线”,方便工具操作,后面用完了还会再拼回去。
总结
这行代码就是:把所有图片从二维的 “方块” 展开成一维的 “长条”,目的是适配后面的处理工具,就像把衣服叠成特定形状才能放进收纳盒一样。
随机过采样器
ros = RandomOverSampler(random_state=42)
这句话是在创建一个 “复制机”,专门用来复制少的那类数据,让两类数据数量一样多。咱们用大白话解释:
ros = RandomOverSampler(...) 是啥?
- RandomOverSampler 翻译过来是 “随机过采样器”,你可以理解成一个 “智能复制机”。
- 它的唯一任务:发现哪类数据少,就随机复制少的那类,直到两类数量一样多(比如正常片少就复制正常片,肺炎片少就复制肺炎片)。
- ros 是给这个复制机起的 “小名”,方便后面调用它干活。
参数 random_state=42 是啥意思?
- 这个参数是给复制机设定 “复制规则”,保证每次复制的结果都一样。
举个例子:
如果没有 random_state=42,复制机第一次可能复制第 1、3、5 张正常片,第二次可能复制第 2、4、6 张 —— 两次结果不一样,电脑学习效果也会波动。
加上 random_state=42 后,就像给复制机定了个 “固定菜谱”,每次都会按同样的规则选要复制的片子,结果完全一样。这样实验结果能重复,方便调试。
(为啥是 42?这是个常用的 “随机种子” 数字,用其他数字比如 100、2024 也可以,只要固定就行。)
总结
- RandomOverSampler 是个 “复制机”,负责把少的样本复制到和多的一样多。
- random_state=42 是为了让复制结果固定不变,保证实验能重复。
就像你按同一个食谱做饭,每次味道都一样,不会忽咸忽淡~
复制机平衡数据
X_resampled, y_resampled = ros.fit_resample(X_flat, y)
这句话的作用是:让 “复制机” 开始工作,把少的那类 X 光片复制到和多的那类数量一样多。咱们用大白话讲:
先回忆一下角色
- ros 是之前初始化的 “复制机”(RandomOverSampler),专门负责复制少的样本。
- X_flat 是 “压平成条” 的 X 光片(长条形,方便复制机操作)。
- y 是这些片子的标签(0 = 正常,1 = 肺炎)。
这行代码干了啥?
- 复制机先 “看” 数据:
复制机(ros)会先检查 y 里的标签,数数 0 和 1 各有多少。
比如发现:正常片(0)有 500 张,肺炎片(1)有 2500 张 —— 正常片太少了。
- 自动复制少的那类:
复制机只复制少的(这里是正常片),一直复制到两类数量一样多。
上面的例子里,会把正常片从 500 张复制到 2500 张,和肺炎片数量相同。
- 返回复制后的结果:
- X_resampled:复制后的 “长条形” X 光片(现在正常和肺炎各 2500 张,共 5000 张)。
- y_resampled:对应的标签(也是 5000 个,0 和 1 各 2500 个)。
举个生活例子
就像你有 5 颗草莓糖和 25 颗巧克力糖,想让两种糖数量一样多:
- 复制机看到草莓糖少,就会复制 20 颗草莓糖(总共 25 颗)。
- 最后得到 25 颗草莓糖(X_resampled里的正常片)和 25 颗巧克力糖(X_resampled里的肺炎片),标签也对应上。
总结
这行代码就是启动 “复制机”,自动把少的样本复制到和多的样本数量相同,让两类数据平衡,方便电脑公平学习。
使用过采样解决训练数据样本不平衡问题
这个 handle_imbalance 函数的核心作用是解决训练数据中不同类别的样本数量不平衡问题(比如肺炎样本远多于正常样本),通过 “过采样” 让两类样本数量趋于均衡,避免模型学习时偏向数量多的类别。
下面分步骤详细解释:
1. 初始化变量
X, y = [], [] num_batches = len(generator) generator.reset() |
- X 用来存储所有图像数据,y 用来存储对应的标签(0 表示正常,1 表示肺炎)。
- num_batches 获取数据生成器(generator)中的批次数量(比如每次生成 32 张图,共 100 批,就是 3200 张图)。
- generator.reset() 重置生成器,确保从第一批数据开始读取,避免漏读或重复。
2. 提取所有数据和标签
for i in range(num_batches): batch_x, batch_y = generator.next() # 获取一批数据(图像+标签) X.append(batch_x) # 把这批图像加入X列表 y.append(batch_y) # 把这批标签加入y列表 X = np.concatenate(X) # 把列表中的所有批次图像合并成一个大数组 y = np.concatenate(y) # 把列表中的所有批次标签合并成一个大数组 |
- 数据生成器是 “分批” 提供数据的(比如一次给 32 张图),这里通过循环把所有批次的数据合并成一个完整的数据集。
- 例如:原来分 10 批,每批 32 张图,合并后 X 就是一个形状为 (320, 150, 150, 3) 的数组(320 张图,每张 150x150 像素,3 通道 RGB)。
3. 查看原始样本分布
print(f"原始样本分布: 正常={np.sum(y == 0)}, 肺炎={np.sum(y == 1)}") |
- 统计并打印原始数据中 “正常”(标签 0)和 “肺炎”(标签 1)的样本数量。
- 假设输出为 原始样本分布: 正常=1000, 肺炎=4000,说明两类样本比例是 1:4,不平衡问题明显。
4. 数据展平(为过采样做准备)
X_flat = X.reshape(X.shape[0], -1) |
- 过采样工具(RandomOverSampler)要求输入的特征是 “二维数组”(样本数 × 特征数),而图像是四维数组(样本数 × 高 × 宽 × 通道)。
- 这一步将每张图像 “展平”:比如 150x150x3 的图像会变成一个长度为 150×150×3=67500 的一维数组,最终 X_flat 形状为 (样本数, 67500)。
5. 过采样少数类
ros = RandomOverSampler(random_state=42) # 初始化过采样器(固定随机种子,结果可重复) X_resampled, y_resampled = ros.fit_resample(X_flat, y) # 对少数类进行过采样 |
- RandomOverSampler 的作用是:复制少数类的样本,让两类样本数量相同。
- 以上面的例子(1000 正常 vs 4000 肺炎),过采样后会变成 4000 正常 vs 4000 肺炎(通过复制 3000 个正常样本实现)。
6. 恢复图像形状
X_resampled = X_resampled.reshape(-1, *X.shape[1:]) |
- 过采样后的 X_resampled 是展平的一维数组,需要恢复成图像的原始形状(高 × 宽 × 通道),方便后续输入模型训练。
- 例如:从 (8000, 67500) 恢复为 (8000, 150, 150, 3)。
7. 查看过采样后的分布
print(f"过采样后分布: 正常={np.sum(y_resampled == 0)}, 肺炎={np.sum(y_resampled == 1)}") |
- 打印过采样后的样本数量,确认两类已平衡(比如 正常=4000, 肺炎=4000)。
8. 返回处理后的数据
return X_resampled, y_resampled, y |
- 返回 3 个结果:
- X_resampled:过采样后的图像数据(平衡后)。
- y_resampled:过采样后的标签(平衡后)。
- 原始标签 y:用于后续计算类别权重等。
总结
这个函数的核心逻辑是:
- 从数据生成器中提取所有原始数据。
- 用过采样方法(复制少数类)平衡两类样本数量。
- 恢复图像形状,方便模型使用。
通过这一步处理,模型在训练时不会因为某类样本多就 “偏爱” 它,能更公平地学习两类特征,提高分类准确性。
