R语言基础| 下载、安装
在此前的单细胞教程中,许多小伙伴都曾因为R语言基础不足而十分苦恼。R语言是一种开源的编程语言和软件环境,专门用于统计分析、图形表示和数据挖掘。它最初由Ross Ihaka和Robert Gentleman在1993年创建,旨在为统计学家和数据分析师提供一个广泛的统计(线性和非线性建模、经典统计测试、时间序列分析、分类、聚类等)和图形技术的集成环境。R语言以其强大的数据处理能力、图形系统以及在统计计算和图形显示方面的灵活性而闻名。此外,R语言具有一个庞大的用户社区,提供了大量的包(libraries)和工具,用于扩展其基本功能。
在生物信息学领域,R语言被广泛应用于基因组学、蛋白质组学、代谢组学等生物数据分析中,原因包括:
1.统计分析能力:生物信息学涉及大量的统计分析,如差异表达分析、基因组关联研究、群体遗传学分析等。R语言提供了强大的统计测试、模型建立和数据分析工具,能有效处理和分析生物学数据。
2.生物信息学专用包:R社区开发了数以千计的软件包,专门针对生物信息学的需求。例如,Bioconductor项目提供了一系列用于基因组学、转录组学、表观遗传学等领域的分析工具和数据集,大大简化了生物信息学的数据处理和分析过程。
3.数据可视化:R语言的强大图形功能使得生物信息学家可以创建高质量的数据可视化图表,如热图、聚类图和基因表达图,这对于数据解释和结果展示至关重要。
4.灵活性和可扩展性:R语言允许用户编写自定义函数和软件包,以满足特定的分析需求。这种灵活性对于应对生物信息学中的复杂和不断变化的数据分析挑战尤为重要。
5.广泛的社区支持:R语言拥有一个庞大的用户和开发者社区,为生物信息学家提供了丰富的资源、教程和论坛,便于学习交流和问题解决。
综上,从本篇推文开始,我们将开展一系列R语言基础的教程,更多学习资料可见:R语言基础学习手册
1.1 R下载与安装
https://cran.r-project.org/
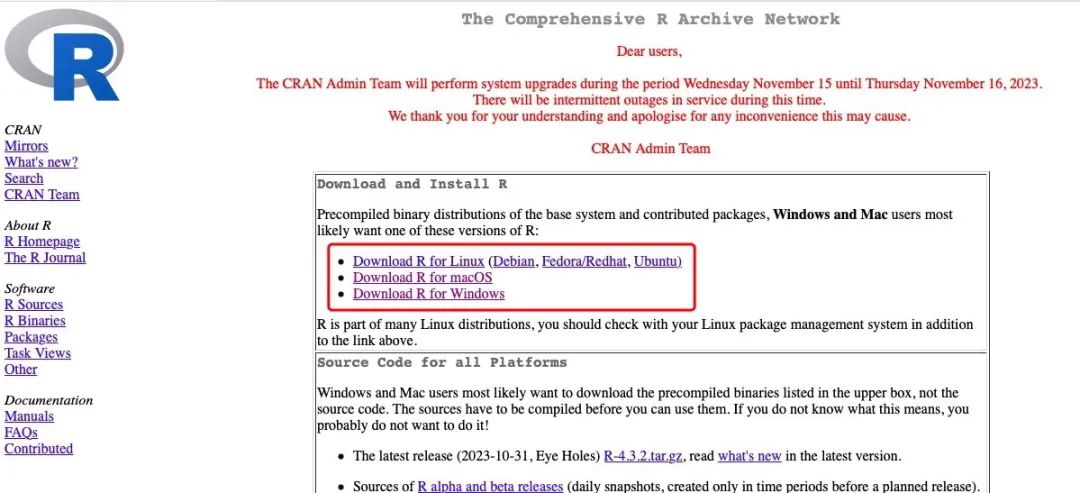
根据电脑系统选择不同的R版本
1.mac系统的则根据macOS版本号选择合适的安装包,下载并安装即可。

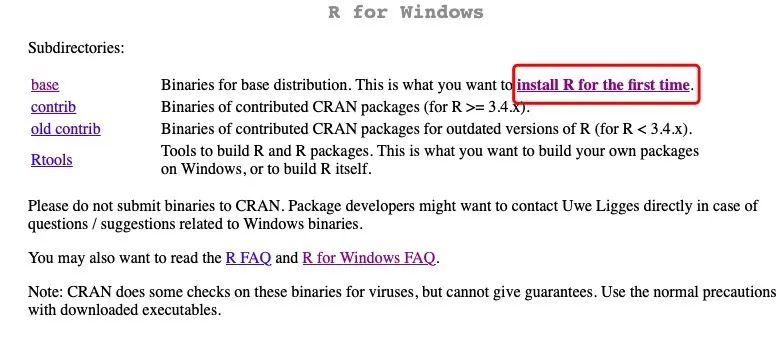
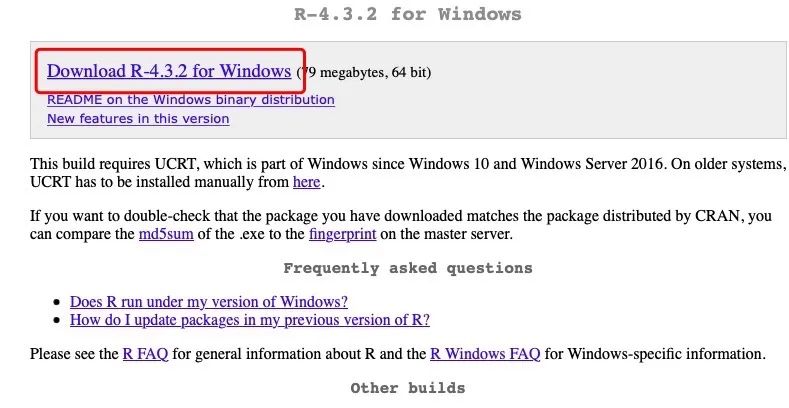
2.windows系统的则按以下步骤下载并安装即可。
3.Linux系统
Linux系统参考我们以前的教程:Rstudio-server的安装、配置、维护
1.1.1 RStudio
RStudio是一款集成开发环境(IDE)。R自带的环境操作起来可能不是很方便,而Rstudio很好地解决了这个问题,且具有调试、可视化等功能,支持纯R脚本、Rmarkdown、Bookdown、Shiny等。
1.1.1.1 Rstudio的下载与安装
下载网址:
https://posit.co/download/rstudio-desktop/
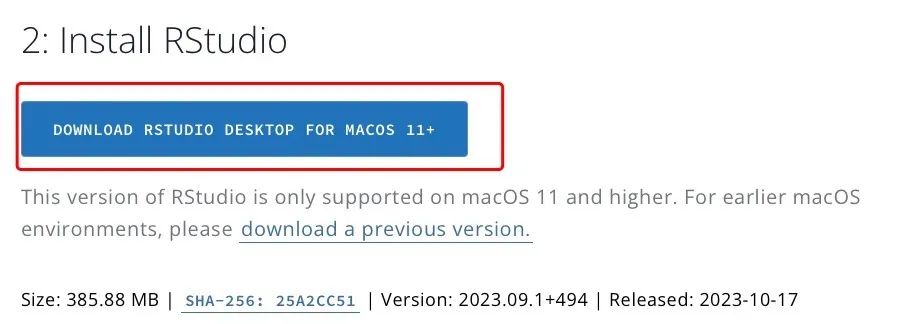
会跳出适合您电脑系统的Rstudio,下载安装即可。
1.1.2 R的帮助函数
| 函数 | 操作 |
|---|---|
| help.start() | 输出通用的帮助信息 |
| help(“foo”)或?foo | 输出函数foo()的帮助信息 |
| help(package =“foo”) | 输出foo包的帮助信息 |
| help.search(“foo”)或??foo | 在帮助系统中查找名称中带有字符串foo的实例(包、类、函数等)的帮助信息 |
| example(“foo”) | 输出foo()的示例信息(可以省略引号) |
| data() | 列出当前已加载的包中的所有可用的示例数据集 |
| vignette() | 列出当前已加载的所有可用的简介信息 |
| vignette(“foo”) | 输出主题foo的简介信息 |
1.1.3 用于管理R工作区的函数
| 函数 | 功能 |
|---|---|
| getwd() | 显示当前的工作目录 |
| setwd(“路径”) | 设置或修改当前的工作目录 |
| Is() | 列出当前工作区中的对象 |
| rm(objectlist) | 移除(删除)一个或多个对象 |
| help(options) | 显示可用选项的说明 |
| options() | 显示或设置当前选项 |
| save.image(“myfile”) | 保存工作区到文件myfile中(默认值为.RData) |
| save(objectlist,file=“myfile”) | 保存指定对象到一个文件中 |
| load(“myfile”) | 读取一个工作区到当前会话中(默认值为.RData) |
| dir.create() | 创建新目录 |
1.2 包(Packages)
1.2.1 包的基本函数
这里涉及需要包名的地方用 tidyr 举例,实操中需要其他包则改为具体的包名;
| 函数 | 操作 |
|---|---|
| .libPaths() | 显示库(库即存储包的目录)所在的位置 |
| library() | 显示库中有哪些包 |
| search() | 显示哪些包已被加载并可以使用 |
| install.packages(“tidyr”) | 安装tidyr包,注意双引号必须有 |
| update.packages(“tidyr”) | 更新包,注意双引号必须有 |
| library(tidyr) | 加载包,无需引号 |
| installed.packages() | 列出已安装的包 |
1.2.2 包处理大型数据集的示例展示
help.start()## 打开httpd帮助服务器… 好了## 如果什么都不发生的话,你应该自己打开'http://127.0.0.1:14344/doc/html/index.html'## Error in shell.exec(url): 无法同'http://127.0.0.1:14344/doc/html/index.html'文件关联help(package="vcd")
library(vcd)## Warning: 程辑包'vcd'是用R版本4.3.2 来建造的## 载入需要的程辑包:gridhelp(Arthritis)## Error in shell.exec(url): 无法同'http://127.0.0.1:14344/library/vcd/html/Arthritis.html'文件关联Arthritis## ID Treatment Sex Age Improved
## 1 57 Treated Male 27 Some
## 2 46 Treated Male 29 None
## 3 77 Treated Male 30 None
## 4 17 Treated Male 32 Marked
## 5 36 Treated Male 46 Marked
## 6 23 Treated Male 58 Marked
## 7 75 Treated Male 59 None
## 8 39 Treated Male 59 Marked
## 9 33 Treated Male 63 None
## 10 55 Treated Male 63 None
## 11 30 Treated Male 64 None
## 12 5 Treated Male 64 Some
## 13 63 Treated Male 69 None
## 14 83 Treated Male 70 Marked
## 15 66 Treated Female 23 None
## 16 40 Treated Female 32 None
## 17 6 Treated Female 37 Some
## 18 7 Treated Female 41 None
## 19 72 Treated Female 41 Marked
## 20 37 Treated Female 48 None
## 21 82 Treated Female 48 Marked
## 22 53 Treated Female 55 Marked
## 23 79 Treated Female 55 Marked
## 24 26 Treated Female 56 Marked
## 25 28 Treated Female 57 Marked
## 26 60 Treated Female 57 Marked
## 27 22 Treated Female 57 Marked
## 28 27 Treated Female 58 None
## 29 2 Treated Female 59 Marked
## 30 59 Treated Female 59 Marked
## 31 62 Treated Female 60 Marked
## 32 84 Treated Female 61 Marked
## 33 64 Treated Female 62 Some
## 34 34 Treated Female 62 Marked
## 35 58 Treated Female 66 Marked
## 36 13 Treated Female 67 Marked
## 37 61 Treated Female 68 Some
## 38 65 Treated Female 68 Marked
## 39 11 Treated Female 69 None
## 40 56 Treated Female 69 Some
## 41 43 Treated Female 70 Some
## 42 9 Placebo Male 37 None
## 43 14 Placebo Male 44 None
## 44 73 Placebo Male 50 None
## 45 74 Placebo Male 51 None
## 46 25 Placebo Male 52 None
## 47 18 Placebo Male 53 None
## 48 21 Placebo Male 59 None
## 49 52 Placebo Male 59 None
## 50 45 Placebo Male 62 None
## 51 41 Placebo Male 62 None
## 52 8 Placebo Male 63 Marked
## 53 80 Placebo Female 23 None
## 54 12 Placebo Female 30 None
## 55 29 Placebo Female 30 None
## 56 50 Placebo Female 31 Some
## 57 38 Placebo Female 32 None
## 58 35 Placebo Female 33 Marked
## 59 51 Placebo Female 37 None
## 60 54 Placebo Female 44 None
## 61 76 Placebo Female 45 None
## 62 16 Placebo Female 46 None
## 63 69 Placebo Female 48 None
## 64 31 Placebo Female 49 None
## 65 20 Placebo Female 51 None
## 66 68 Placebo Female 53 None
## 67 81 Placebo Female 54 None
## 68 4 Placebo Female 54 None
## 69 78 Placebo Female 54 Marked
## 70 70 Placebo Female 55 Marked
## 71 49 Placebo Female 57 None
## 72 10 Placebo Female 57 Some
## 73 47 Placebo Female 58 Some
## 74 44 Placebo Female 59 Some
## 75 24 Placebo Female 59 Marked
## 76 48 Placebo Female 61 None
## 77 19 Placebo Female 63 Some
## 78 3 Placebo Female 64 None
## 79 67 Placebo Female 65 Marked
## 80 32 Placebo Female 66 None
## 81 42 Placebo Female 66 None
## 82 15 Placebo Female 66 Some
## 83 71 Placebo Female 68 Some
## 84 1 Placebo Female 74 Markedexample(Arthritis)##
## Arthrt> data("Arthritis")
##
## Arthrt> art <- xtabs(~ Treatment + Improved, data = Arthritis, subset = Sex == "Female")
##
## Arthrt> art
## Improved
## Treatment None Some Marked
## Placebo 19 7 6
## Treated 6 5 16
##
## Arthrt> mosaic(art, gp = shading_Friendly)
##
## Arthrt> mosaic(art, gp = shading_max)
