【医学影像 AI】视网膜图像多类别分割中的“段内误分类”
更多内容请关注【医学影像 AI by youcans@Xidian 专栏】
【医学影像 AI】学习解决视网膜成像中的段内误分类
- 0. 论文简介
- 0.1 基本信息
- 0.2 论文概览
- 0.3 摘要
- 1. 引言
- 2. 方法
- 2.1 对抗性分割网络
- 2.2 二值转多类别融合网络
- 3. 实验
- 3.1 实验设置
- 3.2 实验结果
- 4. 讨论
- 5. Github 项目介绍
- 6.1 AVSegmentation 项目介绍
- 6.2 AVSegmentation快速入门
0. 论文简介
0.1 基本信息
2022年,Zhou Y 等 在 MICCAI 2021 发布论文 【视网膜图像多类别分割中的“段内误分类”】(Learning to Address Intra-segment Misclassification in Retinal Imaging)。
本文针对视网膜成像中多类别血管分割(区分动脉、静脉与背景)存在的段内误分类问题(动脉与静脉交叉处易相互误判,而二值分割的误差则低的多),提出一种将多类别分割分解为多个二值分割、再通过含对抗性分割网络和深度监督的二值转多类别融合网络进行处理的方法,有效缓解了段内误分类问题。
论文下载: springer,arxiv
项目地址:Github
引用说明: Zhou, Y. et al. (2021). Learning to Address Intra-segment Misclassification in Retinal Imaging. In: MICCAI 2021. Lecture Notes in Computer Science(), vol 12901. Springer, Cham. https://doi.org/10.1007/978-3-030-87193-2_46
0.2 论文概览
- 研究背景与核心问题
- 临床需求:
视网膜眼底图像的血管分割是获取血管形态特征的关键,而血管形态测量对糖尿病、高血压、动脉硬化等眼科及心血管疾病的计算机辅助诊断至关重要;手动标注耗时,需自动化多类别分割(区分动脉、静脉与背景)。 - 现有方法局限:
- 传统方法:分为图基法(利用血管拓扑结构分类,如文献 [3,27])和特征基法(设计特征提取工具鉴别类别,如文献 [11,17]),性能有限。
- 深度学习方法:端到端模型(如 U-Net、TR-GAN 等)虽达当前最优,但存在段内误分类问题—— 动脉与静脉交叉处易相互误判(如图 1 绿色框所示),而二值分割(仅提取单一类别,如动脉)可正确识别,因二值分割标签不确定性更低。
- 研究方法设计
研究提出 “二值分割→特征融合→多类别分割” 的策略,模型含两大核心组件:
- 对抗性分割网络(Adversarial Segmentation Network)
- 二值转多类别融合网络(Binary-to-multi-class Fusion Network)
-
结论:
验证核心假设:二值分割分支可为多类别分割提供交叉处特征增强,有效缓解段内误分类。
提出的二值转多类别融合网络在三个临床数据集上均达当前最优性能,差异具有统计显著性。 -
研究贡献:
- 发现多类别分割中不同类别血管交叉处会出现段内误分类,而二值分割中不存在这一问题;
- 遵循二值转多类别融合策略,提出了一种深度学习模型来解决段内误分类问题;
- 验证所提模型的有效性。
0.3 摘要
准确的多类别分割一直是医学成像中的长期挑战,特别是在类别之间存在高度相似性的情况下。在视网膜照片中分割视网膜血管就是这样一个场景,其中需要识别并区分动脉和静脉以及背景。当动脉和静脉相交时,类别内误分类(即静脉被误分类为动脉或反之)经常发生,而在二元视网膜血管分割中,错误率则要低得多。
因此,我们提出了一种新方法,将多类别分割分解为多个二元分割,然后通过一个二元到多类别的融合网络进行整合。该网络将动脉、静脉和多类别特征图的表示进行合并,每个特征图都通过对抗训练中的专家血管注释进行监督。基于跳跃连接的合并过程明确地保持了类别特定的梯度,以避免深层中的梯度消失,从而有利于区分性特征。
结果显示,与三种最先进的基于深度学习的方法相比,我们的模型在 DRIVE-AV、LES-AV 和 HRF-AV 数据集上分别提高了 4.4%、5.1% 和 4.2% 的 F1 分数。
代码:https://github.com/rmaphoh/Learning-AVSegmentation
1. 引言
视网膜血管形态测量对于糖尿病、高血压和动脉硬化等眼科及心血管疾病的计算机辅助诊断至关重要 [6,28]。眼底图像的视网膜血管分割在视网膜血管特征描述中起着关键作用,它能提供对疾病诊断敏感的丰富形态学特征。然而,视网膜血管的人工标注耗时极长,因此,无论是否借助计算机辅助诊断各类疾病,都对自动化血管分割有着强烈需求,以提高诊断效率。
在视网膜血管自动多类别分割(即从眼底图像中分割出静脉和动脉)领域,主要存在两类方法:基于图的方法和基于特征的方法。基于图的方法利用血管的拓扑结构知识搜索血管树结构,并将其分类为动脉和静脉 [3,27,4,24,30,32];基于特征的方法则设计各类特征提取工具来区分血管类别 [11,17]。目前,达到最先进性能的方法来自端到端的深度学习模型 [9,31,29,7,20,8,16,26]。然而,如图 1 所示,在所有基于深度学习的模型中,类别间血管交叉处出现的段内误分类(即静脉被误分为动脉或反之)是一个公认的普遍问题。
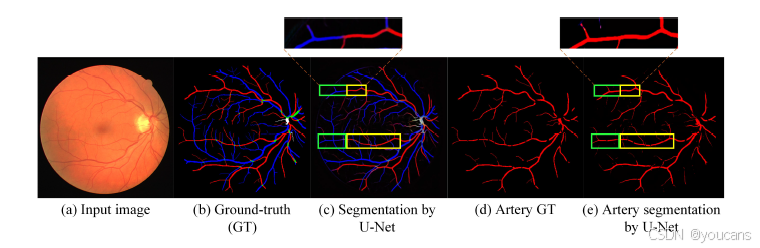
图1. 视网膜血管分割图示意图,突出显示血管交叉处常见错误。在©中,绿色框标记了两条部分动脉在多类别分割算法中被误分类为静脉,而在二值分割任务(e)中被正确分类为动脉。
本文聚焦于视网膜血管多类别分割中的段内误分类问题 [13]。更具体地说,我们观察并发现,如图 1 © 所示,当不同类别血管交叉时,一种段内误分类现象频繁发生,这一问题在文献 [8,15,9,31,16,29,26,18] 中也有报道。但二值分割(例如仅从图像中提取动脉,如图 1 (e) 所示)能够正确识别单一类别的动脉,这可能是因为二值分割任务更简单,像类别间血管交叉处这类像素标签的不确定性更低。这一发现启发我们提出了一种新的多类别分割算法:将多类别分割问题分解为多个二值分割任务,随后通过二值转多类别融合步骤完成分割,如图 2 所示。这种方法可避免在类别间血管交叉这一模糊区域直接进行学习。因此,我们推测,这种专门设计的网络将显著提升在类别间血管交叉导致多类别分类混淆的局部区域的分割性能。
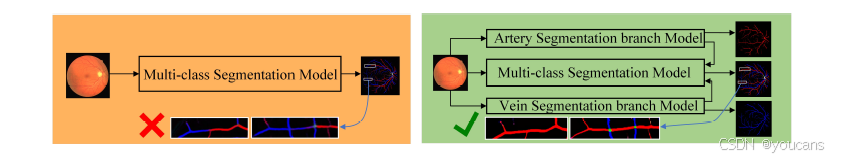
图2. 二值到多类别的融合策略。
我们的研究贡献总结如下:
- 首先,我们发现多类别分割中不同类别血管交叉处会出现段内误分类,而二值分割中不存在这一问题;
- 其次,我们遵循二值转多类别融合策略,提出了一种深度学习模型来解决段内误分类问题,其流程如图 3 所示;
- 最后,我们在三个真实临床数据集上进行了包括消融实验在内的实验,以验证所提模型的有效性。
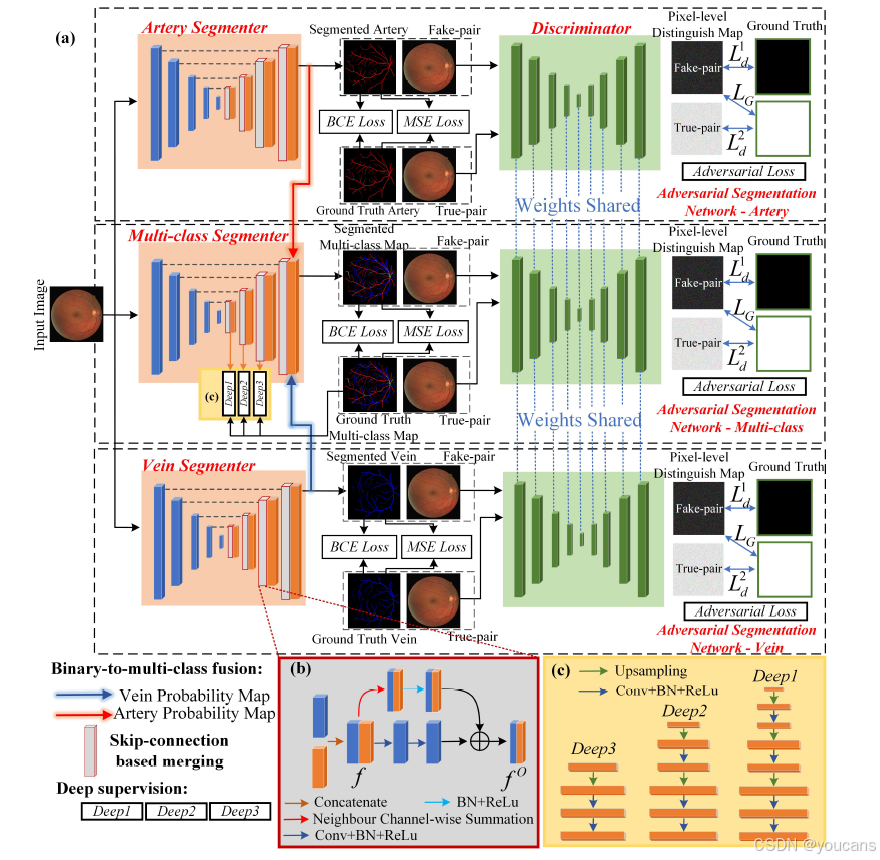
图 3. 二元到多类别融合网络的流程图。基于跳跃连接的合并过程在 (b) 中突出显示。深度监督结构在 © 中展示。Ld1L^1_dLd1、Ld2L^2_dLd2 和 LGL_GLG 是对抗损失中的三个损失目标。
2. 方法
我们设计了一种基于深度学习的模型,该模型包含两个组件:作为融合网络骨干的对抗性分割网络(Adversarial Segmentation Network)和二值转多类别融合网络(Binary-to-multi-class Fusion Network)。
其中,对抗性分割网络负责检测并修正专家标注图与生成器(本文中称为分割器)输出结果之间的高阶不一致性 [14,23];二值转多类别融合网络则将从单一血管类型中学习到的特征表示融合为多类别特征,以补充类别间交叉区域周围各类别独有的判别信息。
2.1 对抗性分割网络
对抗性分割网络由分割器和像素级鉴别器组成。
分割器是 U-Net 的一种变体,而鉴别器采用标准 U-Net 结构 [20]。分割器的输入为视网膜眼底图像,输出为多类别分割图;对于鉴别器,我们将分割图与眼底图像拼接后作为输入,得到像素级判别图作为输出,具体结构如图 3 所示。
分割器中的基于跳跃连接的合并过程
该结构旨在缓解梯度消失问题,同时保留高分辨率信息,其具体形式如图 3 (b) 所示。
首先,我们将同一层级的上采样特征图与下采样特征图拼接,得到组合特征 f∈RW×H×Nf \in \mathbb{R}^{W \times H \times N}f∈RW×H×N(其中W、H、N分别表示空间特征的宽度、高度和通道数,且N可被输出通道数C整除)。
合并过程包含主路径和跳跃路径:主路径中的操作 ϕ:RW×H×N→RW×H×C\phi: \mathbb{R}^{W \times H \times N} \to \mathbb{R}^{W \times H \times C}ϕ:RW×H×N→RW×H×C 由两个模块构成,其中一个模块包含卷积操作、批量归一化(batch normalisation)和激活函数(本文采用 ReLU);跳跃路径中的操作 ψ\psiψ 包含批量归一化和 ReLU。最终输出特征 fOf^OfO的第k个通道(记为 fkOf_k^OfkO)可表示为:
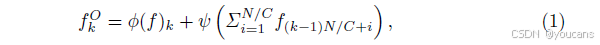
式中,∑i=1N/Cf(k−1)N/C+i\sum_{i=1}^{N / C} f_{(k-1) N / C+i}∑i=1N/Cf(k−1)N/C+i 表示邻域通道求和,该操作分别对上采样特征和下采样特征独立执行。
对抗性训练
为提升分割性能,我们引入了对抗性训练 [22,2]。选择 U-Net 作为鉴别器主要基于以下两点原因:首先,对抗损失是像素级的,因此分割图中每个像素值都能模仿专家标注图中的对应像素值,从而获得边缘清晰、分辨率高的分割结果 [22];其次,与鉴别器中采用下采样卷积块组合的结构相比,U-Net 的鲁棒性更强 [21],能更好地收敛到纳什均衡 [5]。
鉴别器的输入是眼底图像与分割图的拼接结果。为实现收敛,分割器需生成接近真实的分割图,以最小化Ex∼px(x)[log(1−D(x,G(x)))]\mathbb{E}_{x \sim p_{x}(x)}[\log(1-D(x, G(x)))]Ex∼px(x)[log(1−D(x,G(x)))](对应图 3 (a) 中的 LGL_GLG);而像素级鉴别器则需通过最大化 Ey∼pdata(y)[logD(x,y)]+Ex∼px(x)[log(1−D(x,G(x)))]\mathbb{E}_{y \sim p_{data}(y)}[\log D(x, y)] + \mathbb{E}_{x \sim p_{x}(x)}[\log(1-D(x, G(x)))]Ey∼pdata(y)[logD(x,y)]+Ex∼px(x)[log(1−D(x,G(x)))] 来优化性能,即最小化 Ld1L_d^1Ld1 和 Ld2L_d^2Ld2(其中x表示眼底图像,y表示专家标注图)。分割网络的损失函数为:
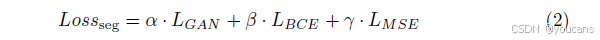
该函数融合了对抗损失(LGANL_{GAN}LGAN)、二值交叉熵损失(LBCEL_{BCE}LBCE)和均方误差损失(LMSEL_{MSE}LMSE),其中 α\alphaα、β\betaβ、γ\gammaγ为权重超参数。正如图 3 (a) 所示,二值交叉熵损失和均方误差损失用于衡量生成器输出G(x)G(x)G(x)与专家标注y 之间的差异。
2.2 二值转多类别融合网络
考虑到二值分割(例如图 1 (e) 所示的动脉分割)具有较高的准确性,我们设计了二值转多类别融合网络,通过融合动脉、静脉和多类别特征的表示,提升交叉区域的多类别分割性能,其整体结构如图 3 (a) 所示。
主分割器输出多类别血管图,且所有三个分割器(动脉分割器、静脉分割器、多类别分割器)共享同一个鉴别器。我们将动脉分割图faf_afa、多类别特征图fmf_mfm和静脉分割图fvf_vfv进行拼接,生成融合特征图,再通过最终的卷积操作得到多类别分割图。
此外,为避免深层网络中的梯度消失问题 [12],我们在上采样阶段引入深度监督机制,以增强判别信息,具体结构如图 3 © 所示。将每个上采样阶段的特征图{fi,i=1,2,3}\{f^i, i=1,2,3\}{fi,i=1,2,3}输入该结构,分别得到fs1f_{s1}fs1、fs2f_{s2}fs2、fs3f_{s3}fs3,并计算对应的深度监督损失{LBCEi,LMSEi∣i∈(s1,s2,s3)}\{L_{BCE_i}, L_{MSE_i} \mid i \in (s1, s2, s3)\}{LBCEi,LMSEi∣i∈(s1,s2,s3)}。结合公式 (2),融合网络的总损失函数为:
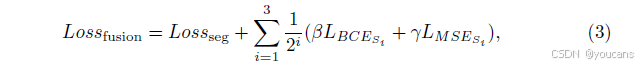
式中,12i\frac{1}{2^i}2i1为三个侧输出的深度监督权重。由于深层网络的梯度消失风险更高,因此为更深层分配了更大的权重。
3. 实验
3.1 实验设置
本研究的实验包含三个临床数据集,分别为 DRIVE-AV [25,10]、LES-AV [19] 和 HRF-AV [1,9]。其中,DRIVE-AV 数据集包含 40 张彩色视网膜眼底图像,图像尺寸为 (565, 584),标注信息涵盖动脉、静脉、交叉区域及不确定区域;LES-AV 数据集包含 22 张图像,尺寸为 (1620, 1444);HRF-AV 数据集包含 45 张图像,尺寸为 (3504, 2336)。
在数据划分方面,DRIVE-AV 数据集中的 20 张图像用于训练,20 张用于测试;LES-AV 数据集中,我们抽取 11 张用于训练,剩余 11 张用于测试;HRF-AV 数据集中,24 张图像用于训练,21 张用于测试。同时,训练集中 10% 的图像被用作验证集。
在实现与训练细节上,出于计算效率考虑 [8,13],训练阶段将 DRIVE-AV 数据集的图像进行零填充处理,使其尺寸变为 (592, 592);将 LES-AV 和 HRF-AV 数据集的图像分别 resize(调整尺寸)为 (800, 720) 和 (880, 592)。而在验证阶段,会将图像恢复至原始尺寸以计算评价指标。所有实验的学习率均设为 0.0008,批次大小(batch size)设为 2。公式 (2) 中的超参数 α、β、γ 分别设置为 0.08、1.1 和 0.5,超参数的搜索过程详见补充材料第 1 节。在单张 Tesla T4 显卡上,模型训练 1500 个 epoch(轮次)所需的总时间约为 12 小时。
在评价指标选择上,视网膜血管多任务分割属于非平衡任务,准确率(accuracy)、特异性(specificity)等指标往往数值较高,缺乏实际参考价值。因此,本研究采用 ROC 曲线下面积(AUC-ROC,Receiver Operating Characteristics)、灵敏度(sensitivity)、F1 分数(F1-score)、PR 曲线下面积(AUC-PR,Precision-Recall)和均方误差(MSE)来全面评估模型性能。以 F1 分数的计算为例,其公式为:
F=(na⋅Fa+nv⋅Fv+nu⋅Fu)/(na+nv+nu)F=(n_{a} \cdot F_{a}+n_{v} \cdot F_{v}+n_{u} \cdot F_{u}) /(n_{a}+n_{v}+n_{u})F=(na⋅Fa+nv⋅Fv+nu⋅Fu)/(na+nv+nu)
其中,nan_{a}na、nvn_{v}nv、nun_{u}nu分别代表标注中属于动脉、静脉和不确定区域的像素数量;FaF_{a}Fa、FvF_{v}Fv、FuF_{u}Fu 分别代表动脉、静脉和不确定区域在二值分割任务(如动脉像素与其他所有像素的区分)中的 F1 分数。此外,本研究采用 Mann-Whitney U 检验(Mann–Whitney U test)来验证不同方法间性能差异的统计显著性。为保证对比的公平性,所有基准模型(baselines)均采用相同的预处理流程、输入尺寸和评价方法进行实现。
3.2 实验结果
与最新方法的性能对比
本研究选取的对比方法包括近期相关研究中的 CS-Net [18]、MNNSA [15]、TR-GAN [2],并以 U-Net 作为基准模型。表 1 列出了所有方法在多类别血管分割任务上的整体性能。曼 - 惠特尼 U 检验结果显示,各方法间性能差异的 P 值均小于 0.05,表明差异具有统计显著性。DRIVE-AV 测试集上的 ROC 曲线和 PR 曲线如图 4 所示。
从实验结果可见,所提方法的性能得到显著提升。例如,在 F1 分数指标上,该方法在 DRIVE-AV、LES-AV 和 HRF-AV 三个数据集上分别比对比方法提升了 4.4%、5.09% 和 4.2%。更多分割结果图可参考补充材料第 2 节。
表1. 在DRIVE-AV、LES-AV与HRF-AV数据集上的多类别血管分割结果,并与最新方法对比。由于TR-GAN为最具竞争力的对比方法,故计算TR-GAN与所提方法之间的Mann–Whitney U检验P值,以显示统计显著性。
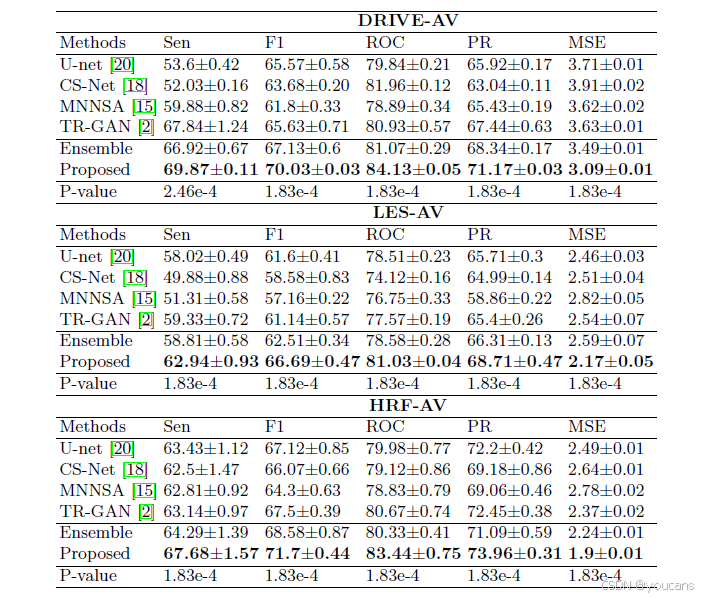
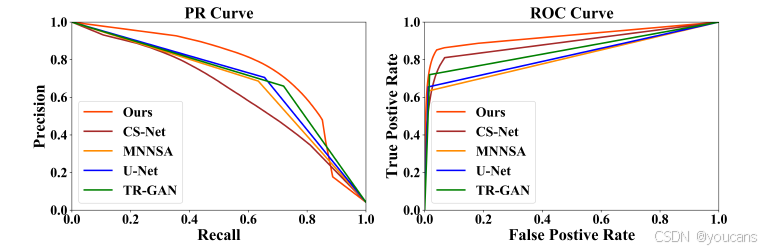
图4. 多种多类别分割方法的ROC与PR曲线。
消融实验
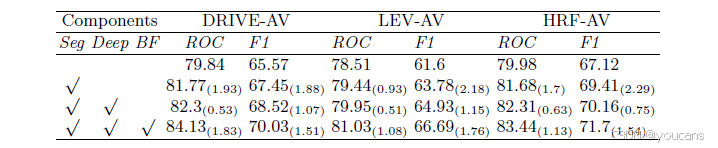
为验证二值转多类别融合网络中各组件的作用,本研究设计了消融实验,分别评估包含 / 不包含对抗性分割网络、深度监督和二值转多类别融合这三个组件时模型的性能,实验结果如表 2 所示。
对比表 2 中第一行的基准模型(标准 U-Net [20])可知,由于采用了基于跳跃连接的合并过程和像素级对抗学习,对抗性分割网络的性能更优。二值转多类别融合网络对整体指标有进一步提升,其中 AUC-ROC 指标最大提升 1.83%,F1 分数最大提升 1.76%。这一结果凸显了引入动脉和静脉二值分割分支的优势。
此外,为验证本研究提出的基于学习的融合策略的有效性,我们在表 1 中设置了 “Ensemble”(集成)方法作为对比:分别训练两个基于对抗性分割的二值分割网络(分别以动脉和静脉标注为监督),然后将动脉分割图与静脉分割图拼接,得到多类别分割结果。
表2. 二值到多类别融合网络的消融实验。对抗分割网络(Seg)、深度监督(Deep)与二值到多类别融合(BF)。评价指标为AUC-ROC(ROC)与F1-score(F1)。括号内显示提升幅度。
4. 讨论
基于消融实验的结果可知,对抗性分割网络与二值转多类别融合网络是模型性能提升的主要贡献者,这两个组件专门针对段内误分类问题设计,能够有效缓解该问题。深度监督机制同样对性能有一定提升作用,但其在 F1 分数上的提升幅度不超过 1.15%。
尽管从定性和定量结果来看,段内误分类问题已得到显著改善,但如图 5 中黄色圆圈所示,仍存在部分未完全解决的情况,只是问题发生的程度大幅降低。具体而言,细小血管分支区域通常更容易出现段内误分类现象。
未来研究将围绕以下两个方向展开探索:第一,可通过多种途径将拓扑正则化嵌入模型,例如采用后处理、定制化卷积滤波器,或是将拓扑结构与表征结构的特征进行融合等方式;第二,探究低对比度背景下的血管分割问题具有重要意义。此外,还可将所提方法嵌入下游临床应用场景(如高血压、糖尿病及动脉硬化的早期诊断与分级)中进一步验证其有效性。
本研究首先明确了在真实临床应用的多类别分割任务中,一个可用于性能提升的关键问题 —— 由类别间血管交叉引发的段内误分类。基于 “二值分割分支能为多类别主分割器提供特征支持,从而提升类别间交叉区域的分割性能” 这一假设,我们提出了二值转多类别融合网络。在三个临床数据集上进行的实验结果验证了该假设的合理性,所提网络也因此取得了当前最优的分割性能。
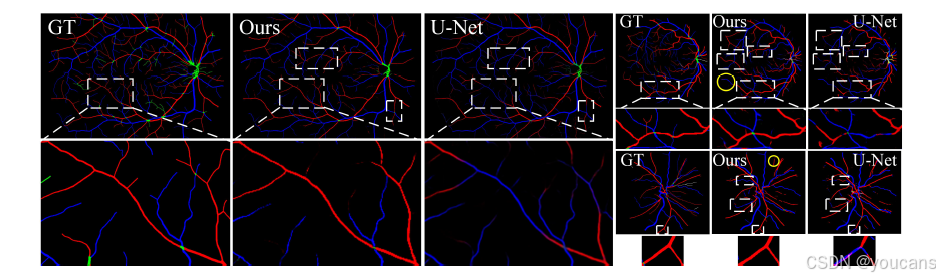
图5. 所提方法与U-Net的可视化结果。白色虚线框示已解决的段内误分类,黄色圆圈示部分残留问题。放大后效果更佳。
5. Github 项目介绍
项目地址:
github
6.1 AVSegmentation 项目介绍
本仓库旨在通过减轻交叉点周围的类别内误分类,提高视网膜眼底照片中多类别血管分割的性能。实验中使用的研究数据集包括 DRIVE-AV 1,2、LES-AV 3 和 HRF-AV 4,5。
本工作有以下几大优势:
- 我们严格以多类别分割的方式评估方法性能,而不仅仅是考虑分类准确率(之前的评估方式)。我们计算了平均值和标准差,以展示测试中的稳健性能。
- 基于生成对抗网络(GAN)的分割框架是在一种最先进的血管分割方法的基础上修订而来的。
- 二元到多类别的融合网络避免了直接学习交叉点带来的模糊像素标签,从而在多类别血管分割方面达到了最先进的性能。
- 代码和算法可以轻松地迁移到其他医学或自然线性分割领域。
6.2 AVSegmentation快速入门
- 安装
需求
- 本项目支持 Linux 和 Windows 系统,但建议使用 Linux 以复现报告的性能。
- 本项目基于以下环境:
- pytorch==1.6.0
- torchvision==0.7.0
- CUDAToolkit==10.1(10.2-11.3 也可支持)
- GPU 是必需的。在我们的工作中,我们使用了一块具有 15 GB 显存的 Tesla T4 GPU。如果使用性能较弱的 GPU,建议在 scripts.utils.py 中更改图像尺寸设置。
安装相关包:
pip install -r requirements.txt
absl-py==0.9.0
art==5.1
cachetools==4.1.1
certifi==2020.6.20
chardet==3.0.4
cycler==0.10.0
Cython==0.29.21
decorator==4.4.2
future==0.18.2
google-auth==1.20.1
google-auth-oauthlib==0.4.1
grpcio==1.31.0
h5py==2.10.0
idna==2.10
imageio==2.9.0
importlib-metadata==1.7.0
jieba==0.42.1
joblib==0.16.0
kiwisolver==1.2.0
Markdown==3.2.2
matplotlib==3.3.4
networkx==2.4
numpy==1.19.1
oauthlib==3.1.0
pandas==1.1.0
Pillow==7.2.0
POT==0.7.0
protobuf==3.12.4
pyasn1==0.4.8
pyasn1-modules==0.2.8
pycm==3.0
pyparsing==2.4.7
python-dateutil==2.8.1
pytz==2020.1
PyWavelets==1.1.1
requests==2.24.0
requests-oauthlib==1.3.0
rsa==4.6
scikit-image==0.17.2
scikit-learn==0.23.2
scipy==1.5.2
seaborn==0.11.1
six==1.15.0
sklearn==0.0
tensorboard==2.3.0
tensorboard-plugin-wit==1.7.0
tensorboardX==2.1
threadpoolctl==2.1.0
tifffile==2020.7.24
torch==1.6.0
torchvision==0.7.0
tqdm==4.48.2
urllib3==1.25.10
Werkzeug==1.0.1
zipp==3.1.0
-
预训练模型(Pretrained Model):
预训练模型已上传至 Google 云端硬盘 Google DRIVE。请下载这些模型,并直接在项目文件夹中解压。 -
训练(Train):
开始训练时,数据集可设置为 DRIVE-AV、LES-AV 或 HRF-AV。
python train.py --e=500 \--batch-size=2 \--learning-rate=8e-4 \--v=10.0 \--alpha=0.5 \--beta=1.1 \--gama=0.08 \--dataset=DRIVE_AV \--discriminator=unet \--job_name=DRIVE_AV_randomseed_42 \--uniform=True \--seed_num=42
if __name__ == '__main__':logging.basicConfig(level=logging.INFO, format='%(levelname)s: %(message)s')args = get_args()device = torch.device('cuda' if torch.cuda.is_available() else 'cpu')logging.info(f'Using device {device}')img_size = Define_image_size(args.uniform, args.dataset)net_G = Generator_main(input_channels=3, n_filters = 32, n_classes=4, bilinear=False)net_D = Discriminator(input_channels=7, n_filters = 32, n_classes=4, bilinear=False)net_G_A = Generator_branch(input_channels=3, n_filters = 32, n_classes=4, bilinear=False)net_G_V = Generator_branch(input_channels=3, n_filters = 32, n_classes=4, bilinear=False)logging.info(f'Network_G:\n'f'\t{net_G.n_channels} input channels\n'f'\t{net_G.n_classes} output channels (classes)\n'f'\t{"Bilinear" if net_G.bilinear else "Transposed conv"} upscaling')logging.info(f'Network_D:\n'f'\t{net_D.n_channels} input channels\n'f'\t{net_D.n_classes} output channels (classes)\n'f'\t{"Bilinear" if net_D.bilinear else "Transposed conv"} upscaling')if args.load:net_G.load_state_dict(torch.load(args.load, map_location=device))logging.info(f'Model loaded from {args.load}')net_D.load_state_dict(torch.load(args.load, map_location=device))logging.info(f'Model loaded from {args.load}')net_G.to(device=device)net_D.to(device=device)net_G_A.to(device=device)net_G_V.to(device=device)train_net(net_G=net_G,net_D=net_D,net_G_A=net_G_A,net_G_V=net_G_V,epochs=args.epochs,batch_size=args.batchsize,alpha_hyper=args.alpha,beta_hyper=args.beta,gama_hyper=args.gama,lr=args.lr,device=device,val_percent=args.val / 100,image_size=img_size)
- 测试(Test):
测试训练后的模型时,需遵循与训练阶段一致的图像预处理逻辑以确保结果准确性:对于 DRIVE-AV 数据集,需将测试图像恢复至原始尺寸(565, 584)(训练时曾零填充至 592, 592);LES-AV 和 HRF-AV 数据集的测试图像也需从训练时的缩放尺寸(分别为 800, 720 和 880, 592)恢复为原始尺寸(LES-AV 为 1620, 1444,HRF-AV 为 3504, 2336)。
python test.py --batch-size=1 \--dataset=DRIVE_AV \--job_name=DRIVE_AV_randomseed \--uniform=True
if __name__ == '__main__':logging.basicConfig(level=logging.INFO, format='%(levelname)s: %(message)s')args = get_args()device = torch.device('cuda' if torch.cuda.is_available() else 'cpu')logging.info(f'Using device {device}')img_size = Define_image_size(args.uniform, args.dataset)dataset_name = args.datasetcheckpoint_saved = dataset_name + '/' +args.jn + '/Discriminator_unet/'test_dir= "./data/{}/test/images/".format(dataset_name)test_label = "./data/{}/test/1st_manual/".format(dataset_name)test_mask = "./data/{}/test/mask/".format(dataset_name)mode = 'whole'acc_total_a = []sensitivity_total_a = []specificity_total_a = []precision_total_a = []G_total_a = []F1_score_2_total_a = []mse_total_a = []iou_total_a = []acc_total_v = []sensitivity_total_v = []specificity_total_v = []precision_total_v = []G_total_v = []F1_score_2_total_v = []mse_total_v = []iou_total_v = []acc_total_u = []sensitivity_total_u = []specificity_total_u = []precision_total_u = []G_total_u = []F1_score_2_total_u = []mse_total_u = []iou_total_u = []acc_total = []sensitivity_total = []specificity_total = []precision_total = []G_total = []F1_score_2_total = []mse_total = []iou_total = []dataset = LearningAVSegData(test_dir, test_label, test_mask, img_size, dataset_name=dataset_name, train_or=False)test_loader = DataLoader(dataset, batch_size=args.batchsize, shuffle=False, num_workers=2, pin_memory=True, drop_last=True)net_G = Generator_main(input_channels=3, n_filters = 32, n_classes=4, bilinear=False)net_G_A = Generator_branch(input_channels=3, n_filters = 32, n_classes=4, bilinear=False)net_G_V = Generator_branch(input_channels=3, n_filters = 32, n_classes=4, bilinear=False)for i in range(1):checkpoint_path_ = "./{}/{}_{}/Discriminator_unet/".format(dataset_name, args.jn,42-2*i)net_G.load_state_dict(torch.load( checkpoint_path_ + 'CP_best_F1_all.pth'))net_G_A.load_state_dict(torch.load( checkpoint_path_ + 'CP_best_F1_A.pth'))net_G_V.load_state_dict(torch.load(checkpoint_path_ + 'CP_best_F1_V.pth'))net_G.eval()net_G_A.eval()net_G_V.eval()net_G.to(device=device)net_G_A.to(device=device)net_G_V.to(device=device)if mode != 'vessel':acc, sent, spet, pret, G_t, F1t, mset, iout, \acc_a, sent_a, spet_a, pret_a, G_t_a, F1t_a, mset_a, iout_a, \acc_v, sent_v, spet_v, pret_v, G_t_v, F1t_v, mset_v, iout_v, \acc_u, sent_u, spet_u, pret_u, G_t_u, F1t_u, mset_u, iout_u = test_net(net_all=net_G, net_a=net_G_A, net_v=net_G_V, loader=test_loader, device=device, mode=mode, dataset_train=dataset_name)#########################################3acc_total_a.append(acc_a)sensitivity_total_a.append(sent_a)specificity_total_a.append(spet_a)precision_total_a.append(pret_a)G_total_a.append(G_t_a)F1_score_2_total_a.append(F1t_a)mse_total_a.append(mset_a)iou_total_a.append(iout_a)###########################################acc_total_v.append(acc_v)sensitivity_total_v.append(sent_v)specificity_total_v.append(spet_v)precision_total_v.append(pret_v)G_total_v.append(G_t_v)F1_score_2_total_v.append(F1t_v)mse_total_v.append(mset_v)iou_total_v.append(iout_v)############################################acc_total_u.append(acc_u)sensitivity_total_u.append(sent_u)specificity_total_u.append(spet_u)precision_total_u.append(pret_u)G_total_u.append(G_t_u)F1_score_2_total_u.append(F1t_u)mse_total_u.append(mset_u)iou_total_u.append(iout_u)###########################################acc_total.append(acc)sensitivity_total.append(sent)specificity_total.append(spet)precision_total.append(pret)G_total.append(G_t)F1_score_2_total.append(F1t)mse_total.append(mset)iou_total.append(iout)print('########################################3')
print('ARTERY')
print('#########################################')print('Accuracy: ', np.mean(acc_total_a))
print('Sensitivity: ', np.mean(sensitivity_total_a))
print('specificity: ', np.mean(specificity_total_a))
print('precision: ', np.mean(precision_total_a))
print('G: ', np.mean(G_total_a))
print('F1_score_2: ', np.mean(F1_score_2_total_a))
print('MSE: ', np.mean(mse_total_a))
print('iou: ', np.mean(iou_total_a))#############################################3
print('########################################3')
print('VEIN')
print('#########################################')
#############################################3
print('Accuracy: ', np.mean(acc_total_v))
print('Sensitivity: ', np.mean(sensitivity_total_v))
print('specificity: ', np.mean(specificity_total_v))
print('precision: ', np.mean(precision_total_v))
print('G: ', np.mean(G_total_v))
print('F1_score_2: ', np.mean(F1_score_2_total_v))
print('MSE: ', np.mean(mse_total_v))
print('iou: ', np.mean(iou_total_v))###########################################
print('########################################3')
print('UNCERTAIN')
print('#########################################')
################################################
print('Accuracy: ', np.mean(acc_total_u))
print('Sensitivity: ', np.mean(sensitivity_total_u))
print('specificity: ', np.mean(specificity_total_u))
print('precision: ', np.mean(precision_total_u))
print('G: ', np.mean(G_total_u))
print('F1_score_2: ', np.mean(F1_score_2_total_u))
print('MSE: ', np.mean(mse_total_u))
print('iou: ', np.mean(iou_total_u))##########################################
print('########################################3')
print('AVERAGE')
print('#########################################')
##########################################
print('Accuracy: ', np.mean(acc_total))
print('Sensitivity: ', np.mean(sensitivity_total))
print('specificity: ', np.mean(specificity_total))
print('precision: ', np.mean(precision_total))
print('G: ', np.mean(G_total))
print('F1_score_2: ', np.mean(F1_score_2_total))
print('MSE: ', np.mean(mse_total))
print('iou: ', np.mean(iou_total))```5. **性能(Performance):**
将最终的激活图(activation map)从 sigmoid 函数替换为 softmax 函数。| Test dataset | Sensitivity | AUC-ROC | F1-score | AUC-PR | MSE |
|--------------|-------------|------------|------------|------------|------------|
| DRIVE-AV | 70.8 ± 0.1 | 84.7 ± 0.05| 71.99 ± 0.04| 73.06 ± 0.03| 2.85 ± 0.01|
| LES-AV | 64.41 ± 0.09| 81.72 ± 0.04| 67.22 ± 0.06| 69.08 ± 0.06| 2.22 ± 0.01|
| HRF-AV | 71.85 ± 0.29| 85.38 ± 0.13| 71.92 ± 0.03| 73.23 ± 0.03| 2 ± 0.01 |<br>## 6. 参考文献```bash
Budai, A., Bock, R., Maier, A., Hornegger, J., Michelson, G.: Robust vessel segmentation in fundus images. Int. J. Biomed. Imaging 2013 (2013)
Chen, W., et al.: TR-GAN: topology ranking GAN with triplet loss for retinal artery/vein classification. In: Martel, A.L., et al. (eds.) MICCAI 2020. LNCS, vol. 12265, pp. 616–625. Springer, Cham (2020). https://doi.org/10.1007/978-3-030-59722-1_59
Dashtbozorg, B., Mendonça, A.M., Campilho, A.: An automatic graph-based approach for artery/Vein classification in retinal images. IEEE Trans. Image Process. 23(3), 1073–1083 (2013)
Estrada, R., Allingham, M.J., Mettu, P.S., Cousins, S.W., Tomasi, C., Farsiu, S.: Retinal artery-vein classification via topology estimation. IEEE Trans. Med. Imaging 34(12), 2518–2534 (2015)
Fedus, W., Rosca, M., Lakshminarayanan, B., Dai, A.M., Mohamed, S., Goodfellow, I.: Many paths to equilibrium: Gans do not need to decrease a divergence at every step. In: International Conference on Learning Representations (2018)
Fraz, M.M., et al.: Blood vessel segmentation methodologies in retinal images-a survey. Comput. Methods Programs Biomed. 108(1), 407–433 (2012)
Fu, H., Xu, Y., Lin, S., Kee Wong, D.W., Liu, J.: DeepVessel: retinal vessel segmentation via deep learning and conditional random field. In: Ourselin, S., Joskowicz, L., Sabuncu, M.R., Unal, G., Wells, W. (eds.) MICCAI 2016. LNCS, vol. 9901, pp. 132–139. Springer, Cham (2016). https://doi.org/10.1007/978-3-319-46723-8_16
Galdran, A., Meyer, M., Costa, P., Campilho, A., et al.: Uncertainty-aware artery/vein classification on retinal images. In: 2019 IEEE 16th International Symposium on Biomedical Imaging (ISBI 2019), pp. 556–560. IEEE (2019)
Hemelings, R., Elen, B., Stalmans, I., Van Keer, K., De Boever, P., Blaschko, M.B.: Artery-vein segmentation in fundus images using a fully convolutional network. Comput. Med. Imaging Graph. 76, 101636 (2019)
Hu, Q., Abràmoff, M.D., Garvin, M.K.: Automated separation of binary overlapping trees in low-contrast color retinal images. In: Mori, K., Sakuma, I., Sato, Y., Barillot, C., Navab, N. (eds.) MICCAI 2013. LNCS, vol. 8150, pp. 436–443. Springer, Heidelberg (2013). https://doi.org/10.1007/978-3-642-40763-5_54
Huang, F., Dashtbozorg, B., ter Haar Romeny, B.M.: Artery/vein classification using reflection features in retina fundus images. Mach. Vis. Appl. 29(1), 23–34 (2018)
Lee, C.Y., Xie, S., Gallagher, P., Zhang, Z., Tu, Z.: Deeply-supervised nets. In: Artificial intelligence and statistics, pp. 562–570. PMLR (2015)
Li, L., Verma, M., Nakashima, Y., Kawasaki, R., Nagahara, H.: Joint learning of vessel segmentation and artery/vein classification with post-processing. In: Medical Imaging with Deep Learning (2020)
Luc, P., Couprie, C., Chintala, S., Verbeek, J.: Semantic segmentation using adversarial networks. arXiv preprint arXiv:1611.08408 (2016)
Ma, W., Yu, S., Ma, K., Wang, J., Ding, X., Zheng, Y.: Multi-task Neural Networks with Spatial Activation for Retinal Vessel Segmentation and Artery/Vein Classification. In: Shen, D., et al. (eds.) MICCAI 2019. LNCS, vol. 11764, pp. 769–778. Springer, Cham (2019). https://doi.org/10.1007/978-3-030-32239-7_85
Meyer, M.I., Galdran, A., Costa, P., Mendonça, A.M., Campilho, A.: Deep convolutional artery/Vein classification of retinal vessels. In: Campilho, A., Karray, F., ter Haar Romeny, B. (eds.) ICIAR 2018. LNCS, vol. 10882, pp. 622–630. Springer, Cham (2018). https://doi.org/10.1007/978-3-319-93000-8_71
Mirsharif, Q., Tajeripour, F., Pourreza, H.: Automated characterization of blood vessels as arteries and veins in retinal images. Comput. Med. Imaging Graph. 37(7–8), 607–617 (2013)
Mou, L., et al.: CS-Net: channel and spatial attention network for curvilinear structure segmentation. In: Shen, D., et al. (eds.) MICCAI 2019. LNCS, vol. 11764, pp. 721–730. Springer, Cham (2019). https://doi.org/10.1007/978-3-030-32239-7_80
Orlando, J.I., Barbosa Breda, J., van Keer, K., Blaschko, M.B., Blanco, P.J., Bulant, C.A.: Towards a glaucoma risk index based on simulated hemodynamics from fundus images. In: Frangi, A.F., Schnabel, J.A., Davatzikos, C., Alberola-López, C., Fichtinger, G. (eds.) MICCAI 2018. LNCS, vol. 11071, pp. 65–73. Springer, Cham (2018). https://doi.org/10.1007/978-3-030-00934-2_8
Ronneberger, O., Fischer, P., Brox, T.: U-Net: convolutional networks for biomedical image segmentation. In: Navab, N., Hornegger, J., Wells, W.M., Frangi, A.F. (eds.) MICCAI 2015. LNCS, vol. 9351, pp. 234–241. Springer, Cham (2015). https://doi.org/10.1007/978-3-319-24574-4_28
Schonfeld, E., Schiele, B., Khoreva, A.: A u-net based discriminator for generative adversarial networks. In: Proceedings of the IEEE/CVF Conference on Computer Vision and Pattern Recognition, pp. 8207–8216 (2020)
Son, J., Park, S.J., Jung, K.H.: Retinal vessel segmentation in fundoscopic images with generative adversarial networks. arXiv preprint arXiv:1706.09318 (2017)
Son, J., Park, S.J., Jung, K.H.: Towards accurate segmentation of retinal vessels and the optic disc in fundoscopic images with generative adversarial networks. J. Digit. Imaging 32(3), 499–512 (2019)
Srinidhi, C.L., Aparna, P., Rajan, J.: Automated method for retinal artery/vein separation via graph search metaheuristic approach. IEEE Trans. Image Process. 28(6), 2705–2718 (2019)
Staal, J., Abràmoff, M.D., Niemeijer, M., Viergever, M.A., Van Ginneken, B.: Ridge-based vessel segmentation in color images of the retina. IEEE Trans. Med. Imaging 23(4), 501–509 (2004)
Wang, Z., Lin, J., Wang, R., Zheng, W.: Retinal artery/vein classification via rotation augmentation and deeply supervised u-net segmentation. In: Proceedings of the 2019 4th International Conference on Biomedical Signal and Image Processing (ICBIP 2019), pp. 71–76 (2019)
Wang, Z., Jiang, X., Liu, J., Cheng, K.T., Yang, X.: Multi-task siamese network for retinal artery/vein separation via deep convolution along vessel. IEEE Trans. Med. Imaging 39(9), 2904–2919 (2020)
Wong, T.Y., Klein, R., Klein, B.E., Tielsch, J.M., Hubbard, L., Nieto, F.J.: Retinal microvascular abnormalities and their relationship with hypertension, cardiovascular disease, and mortality. Surv. Ophthalmol. 46(1), 59–80 (2001)
Wu, Y., Xia, Y., Song, Y., Zhang, Y., Cai, W.: Nfn+: a novel network followed network for retinal vessel segmentation. Neural Netw. 126, 153–162 (2020)
Xie, J.: Classification of retinal vessels into artery-vein in OCT angiography guided by fundus images. In: Martel, A.L., et al. (eds.) MICCAI 2020. LNCS, vol. 12266, pp. 117–127. Springer, Cham (2020). https://doi.org/10.1007/978-3-030-59725-2_12
Xu, X., et al.: Simultaneous arteriole and venule segmentation with domain-specific loss function on a new public database. Biomed. Opt. Express 9(7), 3153–3166 (2018)
Zhao, Y., et al.: Retinal vascular network topology reconstruction and artery/vein classification via dominant set clustering. IEEE Trans. Med. Imaging 39(2), 341–356 (2019)引用说明: Zhou, Y. et al. (2021). Learning to Address Intra-segment Misclassification in Retinal Imaging. In: MICCAI 2021. Lecture Notes in Computer Science(), vol 12901. Springer, Cham. https://doi.org/10.1007/978-3-030-87193-2_46
版权说明:
youcans@xidian 作品,转载必须标注原文链接:
【医学影像 AI】视网膜图像多类别分割中的“段内误分类”(https://youcans.blog.csdn.net/article/details/154194820)
Crated:2025-11
