【医学影像 AI】一种用于生成逼真的3D血管的分层部件生成模型
更多内容请关注【医学影像 AI by youcans@Xidian 专栏】
【医学影像 AI】一种用于生成逼真的3D血管的分层部件生成模型
- 0. 论文简介
- 0.1 基本信息
- 0.2 论文速览
- 0.3 摘要
- 1. 引言
- 2. 相关工作
- 2.1 血管生成
- 2.2 3D 基于部件的形状建模
- 3. 方法
- 3.1 模型概述
- 3.2 阶段 1:关键图生成
- 3.2.1 编码阶段
- 3.2.2 解码阶段
- 3.2.3 损失函数
- 3.3 阶段 2:血管段生成
- 3.3.1 编码与解码
- 3.3.2 损失函数
- 3.4 阶段 3:分层血管组装
- 4. 实验
- 5. 结论
- 6. 代码下载与使用
- 6.1 项目下载
- 6.2 项目应用
- 7. 参考文献
0. 论文简介
0.1 基本信息
2025 年 清华大学 Siqi Chen 等在 MICCAI2025 发表论文 “一种用于生成逼真的3D血管的分层部件生成模型(Hierarchical Part-based Generative Model for Realistic 3D Blood Vessel)”。
本文提出了一种 三阶段、分层式 的3D 血管生成框架。该方法通过将复杂的血管生成任务分解为“通过 RVAE 构建全局关键图”、“Transformer-VAE 生成局部血管段”和“分层组装”三个步骤,成功地解决了现有方法难以兼顾全局拓扑正确性和局部几何细节的难题,在多个真实血管数据集上取得了SOTA性能,为医学领域复杂血管网络生成提供高精度、高鲁棒性的解决方案。
论文下载: arxiv
项目下载: github
引用格式:
S. Chen, G. Zhang, J. Lai, et al. (2026). Hierarchical Part-Based Generative Model for Realistic 3D Blood Vessel. In: Gee, J.C., et al. MICCAI 2025. Lecture Notes in Computer Science, vol 15962. Springer, Cham. https://doi.org/10.1007/978-3-032-04947-6_25

0.2 论文速览
解决的问题
- 3D 血管建模的固有挑战:血管具有复杂的分支模式、不规则曲率及形状,其全局二叉树状拓扑与局部几何细节难以同时精准表征,传统 3D 建模方法(针对桌椅等规则物体)无法适配。
- 现有方法的局限性:
- 点云类方法(如 PointDiffusion):因离散特性难以捕捉管状结构几何,重建网格易出现孔洞,且生成质量与拓扑准确性不足。
- 血管专用模型:VesselVAE 仅能处理少分支血管,多分支场景下保真度下降;TreeDiffusion(基于隐式神经场)虽能建模解剖树,但复杂血管几何的灵活性与准确性受限,易生成不规则、不连续结构。
- 缺乏全局与局部的协同建模:现有方法未明确分离血管全局拓扑与局部细节,难以兼顾结构完整性与几何精细度。
提出的方法
论文提出分层部件式 3D 血管生成框架,通过三阶段流程分离全局拓扑与局部几何建模。
- 阶段 1:关键图生成(全局拓扑建模)
- 阶段 2:血管段生成(局部几何建模)
- 阶段 3:分层血管组装(全局与局部融合)
主要贡献
- 方法创新性:首次将基于部件的生成方法成功应用于 3D 血管建模,打破传统整体建模思路,为复杂管状结构生成提供新范式。
- 建模精度提升:明确分离全局关键图(拓扑)与局部序列曲线(几何),既保证血管分支的层级完整性,又精准捕捉半径、曲率等局部细节,解决现有方法 “顾全局失局部” 或 “顾局部失全局” 的问题。
0.3 摘要
随着三维视觉技术的发展,血管建模在医学应用中的影响力不断提升。然而,由于血管具有复杂的分支模式、曲率及不规则形状,精准表征其复杂的几何结构与拓扑关系仍是一项挑战。
本研究提出一种分层部件式三维血管生成框架,将血管的全局二叉树状拓扑与局部几何细节进行分离处理。该方法通过三个阶段实现:
- 生成关键图以构建整体层级结构模型;
- 基于几何属性生成血管段;
- 依据全局关键图整合局部血管段,完成分层血管组装。
我们在真实数据集上对该框架进行了验证,结果表明,其在复杂血管网络建模方面的性能优于现有方法。本研究首次成功将基于部件的生成方法应用于三维血管建模,为血管数据生成设立了新基准。
相关代码可在以下链接获取:https://github.com/CybercatChen/PartVessel.git
1. 引言
随着计算机图形学的快速发展,3D 视觉技术大幅提升了医学数据精准建模的能力 [21,22,24]。其中,3D 血管生成技术对于精准模拟血管系统的复杂结构至关重要,可支持从诊断评估 [8] 到治疗方案制定 [12] 等一系列医学应用。该技术还能为术前模拟 [14]、医学影像分析 [31] 等关键任务提供支撑,助力更精准、高效的医疗决策制定。此外,3D 血管生成技术可生成详细的数据集,应用于血管分割 [5,29]、标注 [27] 等下游任务,进一步提升自动化分析的效果。
与针对椅子、桌子、飞机等具有固定且可预测结构的规则形状物体的常规 3D 建模方法 [3] 不同,血管建模面临独特挑战。那些通常为刚性、均匀物体设计的传统方法 [11,20,25],并不适用于血管建模。如图 1 所示,血管系统具有高度复杂性,不仅分支点的数量和位置存在差异,血管本身还呈现出复杂的曲率和不规则、非均匀的特征。真实数据集中血管结构的形态多样性,进一步加剧了这些挑战。这种复杂性要求模型既能有效捕捉精细细节,又能精准表征血管几何形态的多样性。
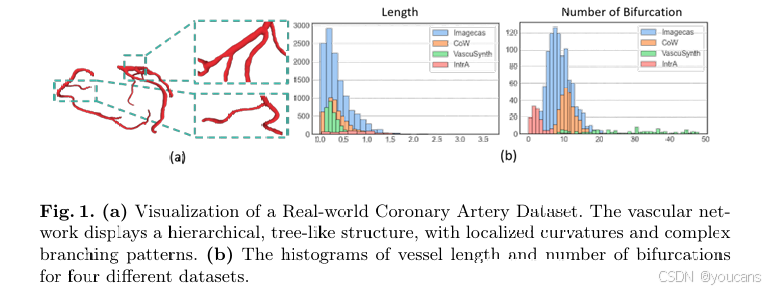
图1. (a) 实际冠状动脉数据集的可视化。血管网络呈现出分层的树状结构,具有局部弯曲和复杂的分支模式。
(b) 四个不同数据集中的血管长度直方图和分叉数直方图。
为实现血管结构的精细化建模,研究人员已提出多种方法。针对普通物体设计的基于点云的方法 [11] 虽能有效表征 3D 物体,但由于其离散特性,难以捕捉管状延伸结构的几何形态。TreeDiffusion [17] 采用隐式神经场建模解剖树,但在捕捉复杂血管几何形态时,灵活性和准确性均受限。VesselVAE [6] 利用骨架图有效捕捉血管结构,但其在生成整个血管网络时,未明确处理各分支的独特特征,因此在分支较少的简单血管建模中表现较好,而在分支较多的血管建模中,保真度会显著下降。
尽管这些方法已考虑血管的几何属性,但一个有效的血管建模方法必须同时捕捉全局和局部特征。从全局来看,大多数血管遵循树状层级结构,其组织方式在很大程度上由端点和分支点决定;从局部来看,血管虽在半径和长度上存在差异,但均呈现出相似的几何形态,可视为管状曲线的片段。
基于这些观察,我们提出一种 “分层部件式血管生成框架”:通过基于血管骨架的树状关键图表征全局血管结构,将局部血管段建模为有序曲线。这种分解方式能够自然地同时捕捉血管系统的分支拓扑结构和局部几何细节。
本研究的贡献如下:
(1)据我们所知,本研究首次将基于部件的方法应用于 3D 血管生成;
(2)明确将血管的全局结构表征为关键图,将局部血管段表征为有序曲线,显著提升了血管建模的细节度;
(3)在真实数据集上对所提方法进行验证,证明其在复杂血管网络建模方面的性能优于现有方法。
2. 相关工作
2.1 血管生成
尽管深度学习已应用于血管生成领域,但该领域的研究仍较为有限。Wolterink 等人 [23] 首次采用生成对抗网络(GANs),以序贯方式生成单条血管的表征。随后,Feldman 等人 [6] 提出了 VesselVAE 模型,用于对具有分支结构的血管进行建模,但该模型仅能处理分支数量较少的血管。Sinha 等人 [17] 探索了将隐式神经表示(INRs)与扩散模型结合用于血管生成,但在建模保真度方面仍面临挑战。尽管这些方法提供了有价值的研究思路,但血管生成任务本身的复杂性仍对实际应用构成限制,这也凸显了进一步提升建模保真度与结构准确性的必要性。
2.2 3D 基于部件的形状建模
深度学习中的基于部件的方法会将复杂的 3D 形状分解为语义上有意义的组件,对这些组件分别进行学习后,再将其组装成完整的结构 [4,9]。其中,GRASS [10] 与 StructureNet [13] 首次强调了层级结构在 3D 形状建模中的重要性,而 CompoNet [16] 则通过部件的变换及其组合方式,提升了生成形状的多样性。借鉴这些方法的思路,我们注意到其与血管结构存在相似性 —— 血管既具有全局树状结构,又在局部呈现管状几何形态,这为我们将基于部件的方法应用于血管建模提供了依据。
3. 方法
3.1 模型概述
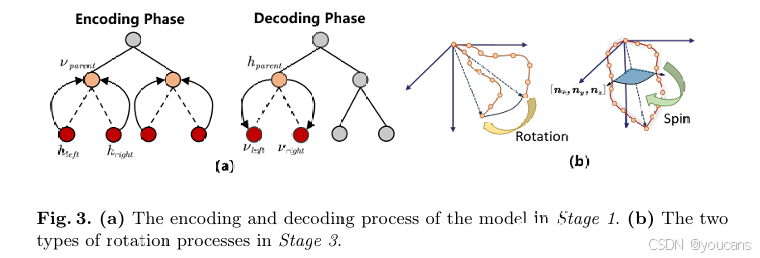
我们采用骨架表示方式,并基于骨架的分叉点与端点构建二叉树结构的关键图。在该关键图中,每条边对应一个独立的血管段。如图 2 所示,我们提出的基于部件的生成模型包含三个阶段:第一阶段学习血管的全局二叉树结构;第二阶段对每个血管段进行建模;最后在第三阶段,根据关键图将生成的各个血管段组装起来,重建完整的血管结构。下文将详细阐述每个阶段所采用的具体方法。
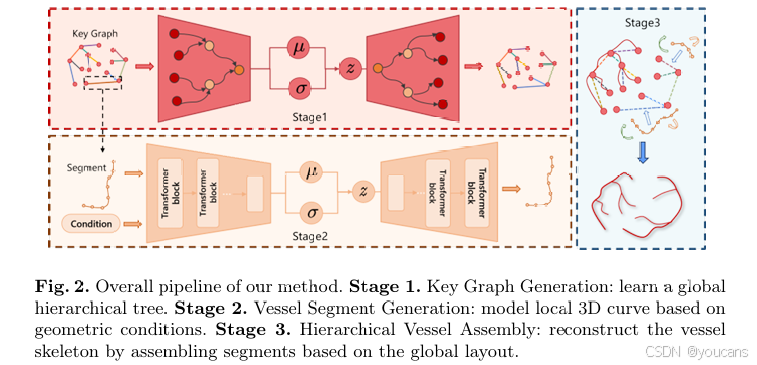
图2. 本方法的整体流程。
阶段1. 关键图生成:学习全局分层树状结构。
阶段2. 血管段生成:基于几何条件建模局部3D曲线。
阶段3. 分层血管组装:基于全局布局组装段以重建血管骨架。
3.2 阶段 1:关键图生成
递归自编码器(Recursive Autoencoders, RAE)最初由文献 [18,19] 提出,随后文献 [10] 将其应用于物体建模,文献 [6] 则将其用于血管建模。为将递归自编码器扩展为生成框架,我们采用递归变分自编码器(Recursive Variational Autoencoder, RVAE)对血管网络的关键图表示进行建模与生成。
每个节点的属性包含三部分:(1)三维空间坐标 [x,y,z];(2)局部血管段的方向 [nx,ny,nz][n_x, n_y, n_z][nx,ny,nz];(3)描述局部血管段属性的几何描述符 C=[ℓ,δ,κ,ρ]C=[ℓ,δ,κ,ρ]C=[ℓ,δ,κ,ρ],该描述符的具体含义将在后续阶段中说明。
3.2.1 编码阶段
编码过程从叶节点开始,逐层向上将子节点的特征聚合到父节点中。设vparentv_{parent}vparent 为父节点的属性,hlefth_{left}hleft 和 hrighth_{right}hright 分别为其左、右子节点的隐藏状态,则父节点的隐藏状态 hparenth_{parent}hparent 按以下公式计算:
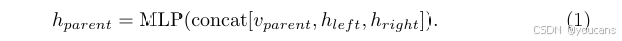
通过在树结构中重复执行上述操作,最终可得到根节点 ZrootZ_{root}Zroot ,该节点作为整个关键图的全局潜在嵌入。
3.2.2 解码阶段
解码过程与编码过程相反,从根节点开始逐层向下重建各节点的属性。对于隐藏状态为 hparenth_{parent}hparent 的父节点,首先通过分类器
y=NodeCLS(h^parent)y=NodeCLS(\hat{h}_{parent})y=NodeCLS(h^parent) 判断其是否存在左子节点和 / 或右子节点。若预测存在左子节点,则左子节点的属性按以下公式计算:
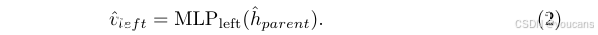
随后,左子节点的隐藏表示按以下公式得到:
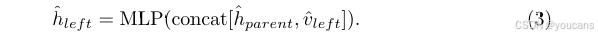
同理,若预测存在右子节点,则采用相同方式计算 v^right\hat{v}_{right}v^right 和 h^right\hat{h}_{right}h^right。上述递归过程持续进行,直至所有节点均完成重建。
3.2.3 损失函数
总损失包含三项:节点属性重建的均方误差 MSE(v,v^)MSE(v, \hat{v})MSE(v,v^)、节点级分类的交叉熵 CrossEntropy(y^,y)CrossEntropy(\hat{y}, y)CrossEntropy(y^,y),以及用于正则化潜在空间的 KL 散度。这三项共同构成最终的损失函数:
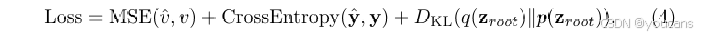
3.3 阶段 2:血管段生成
对于关键图中确定的每个血管段,我们将其骨架表示为三维空间中的有序序列。序列中的每个点用 x=[x,y,z,r]x=[x,y,z,r]x=[x,y,z,r] 描述,其中
r 为血管半径。
为捕捉血管段的形状特征,我们引入几何描述符 C=[ℓ,δ,κ,ρ]C=[ℓ,δ,κ,ρ]C=[ℓ,δ,κ,ρ] 作为条件变量,该描述符与关键图的属性相对应:具体而言,ℓ 表示血管段的长度, δ 表示血管段端点间的直线距离,κ 用于量化血管段的曲率, ρ 表示血管段在二叉树中的深度(以适配不同分支层级的差异)。这些特征共同捕捉了血管段的局部几何信息与整体结构属性,为血管段的精准生成建模提供支持。
3.3.1 编码与解码
我们采用基于 Transformer 的变分框架,将序列中的每个点视为一个 “令牌”(token)。通过以几何描述符 C 为条件,该 Transformer 能够生成更贴合真实血管形态的血管段。编码器将输入序列映射为潜在表示,解码器则从该潜在空间中生成新的序列。
3.3.2 损失函数
总损失包含三项:衡量序列重建误差的均方误差 MSE(x,x^)MSE(x, \hat{x})MSE(x,x^)、确保序列长度准确性的交叉熵 CrossEntropy(ℓ,ℓ^)CrossEntropy(ℓ, \hat{ℓ})CrossEntropy(ℓ,ℓ^),以及 用于生成过程的 KL 散度。因此,损失函数定义如下:
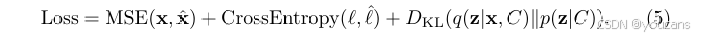
3.4 阶段 3:分层血管组装
在这一最终阶段,我们首先从阶段 1 的潜在空间中采样一个潜在向量,并将其解码为关键图;随后,将该关键图与阶段 2 生成的各个独立血管段进行组装,在同一坐标系下构建完整的血管骨架。
具体而言,我们从关键图的根节点开始,采用深度优先搜索遍历方式进行组装:在遍历的每一步中,首先通过缩放与平移操作对接相应的血管段,确保其空间位置对齐与方向匹配;接着旋转该血管段,使其局部方向与从关键图中获取的方向 [nx,ny,nz][n_x, n_y, n_z][nx,ny,nz] 保持一致(图 3 (b) 展示了两种旋转过程)。这些旋转操作确保了血管段方向与整体血管几何结构的一致性。
通过上述变换逐步对接每个血管段,即可得到完整的血管骨架。最后,参考文献 [30] 中的网格重建方法,利用每个血管段的预测半径,从骨架中重建出最终的三维血管表面。
4. 实验
-
数据集和数据准备。
我们在两个真实世界的数据集和一个合成数据集上进行了实验,所有这些数据集都是公开可用的。 (a) ImageCAS [28]:1,000个真实的冠状动脉3D CCTA扫描,呈现出显著的解剖学变异性。我们是第一个研究这个具有挑战性的数据集的团队。 (b) VascuSynth [7]:一个合成数据集,包含120个生成的3D血管树,具有不同数量的分叉。 © 处理后的CoW [15,26]:300个处理后的颅内动脉3D血管网格。所有数据集均使用90%用于训练,10%用于测试。
我们对3D体积执行一系列预处理步骤,以提取带有半径信息的骨架并推导出关键图。首先,我们对二进制标签体积应用形态学操作,以获得骨架及其对应的表面。接下来,我们采用自适应映射[30]方法构建关键图并构建最大生成树,其中根节点、叶节点和分支节点被识别为关键图的顶点。 -
基线和指标。
为了评估我们提出的方法,我们选择了三种基线模型进行比较。首先,我们包括了一个最先进的点云生成模型[11]。同时,我们纳入了两个专门用于血管生成的模型:TreeDiffusion [17](D=128,L=5)和VesselVAE [6]。
为了全面评估血管重建和生成,我们使用基于点云和图表示的指标。对于从网格采样的点云,我们报告Jensen-Shannon散度(JSD)和Chamfer距离(CD)[1,25],分别用于评估生成和重建质量。对于组装的骨架图,我们专注于检查骨架图的几何和拓扑属性,包括度分布的最大均值差异(Deg.)和拉普拉斯谱(Spec.)。为了准确评估骨架图的重建性能,这些本质上是3D几何图,我们采用了[2]中的图Wasserstein距离(GWD)。 -
实现细节。
所有实验均使用PyTorch在NVIDIA A800 GPU上进行,使用Adam优化器。训练过程分为两个连续阶段。在第一阶段,我们将潜在空间设置为512维。我们从0.001的学习率开始,每100个周期减少0.2。模型训练了20k个周期,批量大小为128,大约在12小时内收敛。在第二阶段,我们将潜在空间设置为64维,批量大小设置为512。学习率设置为0.0002,训练了2k个周期,大约需要3小时。我们将Transformer块设置为4层,编码器和解码器中都有4个多头自注意力,最大序列长度设置为200。 -
实验结果。
表1 比较了我们的方法与三种最先进的方法在基于点云和基于图的指标下的表现。我们的模型在大多数任务中表现出竞争力,尤其是在基于图的结构保真度和拓扑一致性评估中。相比之下,VesselVAE在涉及众多连续点的复杂分支中表现不佳,导致性能始终次优。PointDiffusion在所有数据集上表现出最强的重建指标。然而,它在生成指标和样本质量方面表现不足,表明其无法充分捕捉血管的几何和拓扑结构。我们还观察到TreeDiffusion在ImageCAS数据集上报告了较高的JSD分数,但显示出较差的定性生成结果。由于JSD主要用于评估点云的空间分布,然而它未能考虑它们的拓扑结构的准确性。
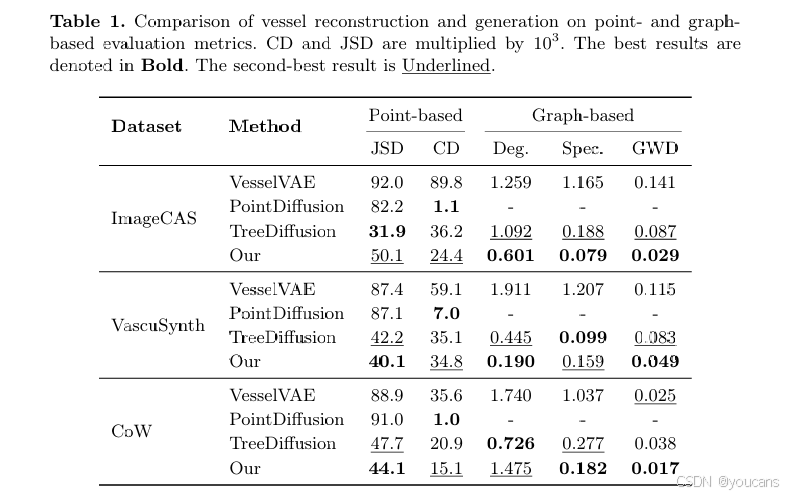
表 1 基于点云和基于图的评价指标下血管重建与生成性能对比(CD 和 JSD 均乘以 10³;最优结果以粗体标注,次优结果以下划线标注)
尽管PointDiffusion和TreeDiffusion在某些指标上优于我们的方法,但对它们的重建和生成样本进行视觉比较揭示了它们建模技术中的一些根本问题。一些重建和生成样本在图4和图5中进行了可视化。特别是,由于ImageCAS数据集中的复杂数据分布,所有比较模型都未能成功生成血管的基本形态。
如图4所示,我们的模型不仅以高精度复制了复杂的分支结构,还捕捉到了血管的细微形态变化。相比之下,基于点云的方法难以区分血管的内表面和外表面,导致重建网格中出现多个孔洞,对下游任务(如血管分析)产生负面影响。我们的基于骨架图的方法有效地处理了复杂的分支结构,并准确捕捉了形态变化,凸显了其在真实世界条件下的鲁棒性。通过直接利用骨架和半径信息,我们的方法自然避免了基于点云重建的陷阱,并为管状结构生成了更鲁棒的网格重建。
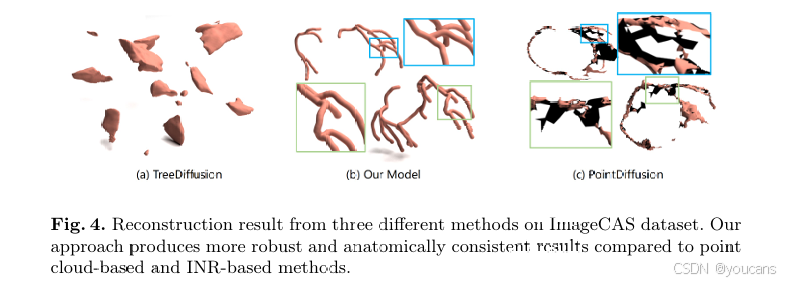
图 4 ImageCAS 数据集上三种方法的重建结果对比:与基于点云和基于隐式神经场(INR)的方法相比,所提方法能生成更稳健、解剖学一致性更强的结果
图 5 对比了所提方法与性能极具竞争力的 TreeDiffusion 的生成性能(采用 TreeDiffusion 性能最优的样本)。结果显示,在所有数据集上,TreeDiffusion 生成的样本常呈现不规则的块状结构和不连续组件,存在明显的结构异常;与之相反,所提模型能保持血管的连续性,生成更贴近真实、解剖学一致性更强的血管网络。
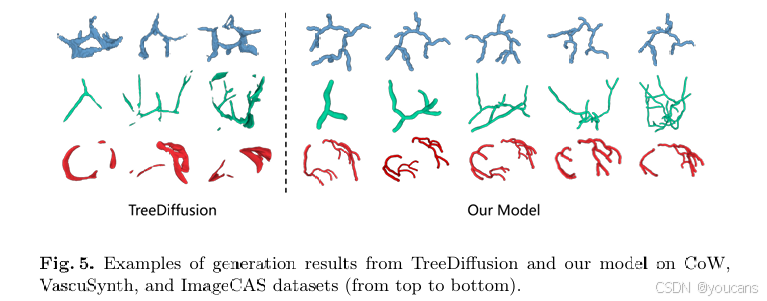
图 5 TreeDiffusion 与所提模型在 CoW、VascuSynth 和 ImageCAS 数据集上的生成结果示例(从上到下依次为三个数据集)
5. 结论
在本文中,我们提出了一个分层的基于部件的框架,用于3D血管生成,该框架将全局树状结构与局部几何分开。
我们的方法分为三个阶段:
- 首先,我们使用递归变分自编码器构建关键图以捕捉血管层次结构。
- 随后,我们引入基于Transformer的变分自编码器来合成详细的血管段。
- 最后,这些血管段被组装成完整的血管。
在三个公共数据集上的实验结果表明,我们的模型始终保留了血管的连续性和真实的局部曲线特征。
6. 代码下载与使用
6.1 项目下载
代码开源: 作者开源了代码,便于社区跟进研究和应用。
项目下载: github
数据集:
- ImageCAS: GitHub - ImageCAS
- Processed CoW: GitHub - vessel_diffuse
- Vascusynth: ]Vascusynth Data](https://vascusynth.cs.sfu.ca/Data.html)
6.2 项目应用
- 数据预处理:
/data_preprocess
graph2node.py
normalization.py
ske2keyseq.py
skeleton2key.py
skeleton2seq.py
utils_process.py
- 第一阶段:
python train_tree.py
程序代码如下。
from model.model_tree import RecursiveEncoder, RecursiveDecoder
from utils.torch_f import Fold, encode_structure_fold, decode_structure_fold
from utils.utils import setup_logging, graph_to_ply
from test_tree import decode_testing
from evaluation.structure_evaluator import get_stats_evalimport numpy as np
import time
import loggingdef train_one_epoch(encoder, decoder, dataloader, optimizer, arg):encoder.train()decoder.train()total_recon_loss = 0total_kl_loss = 0for _, (trees, num_nodes, graphs, file_names) in enumerate(dataloader):enc_fold = Fold(arg.device)enc_fold_nodes = [encode_structure_fold(enc_fold, tree) for tree in trees]enc_fold_nodes = enc_fold.apply(encoder, [enc_fold_nodes])enc_fold_nodes = torch.split(enc_fold_nodes[0], 1, 0)dec_fold = Fold(arg.device)dec_fold_nodes = []kld_fold_nodes = []for tree, fold_node in zip(trees, enc_fold_nodes):root_code, kl_div = torch.chunk(fold_node, 2, 1)dec_fold_nodes.append(decode_structure_fold(dec_fold, root_code, tree))kld_fold_nodes.append(kl_div)total_loss = dec_fold.apply(decoder, [dec_fold_nodes, kld_fold_nodes])num_nodes = torch.as_tensor(num_nodes, device=arg.device)recon_loss = torch.div(total_loss[0], num_nodes).sum() / len(trees)kl_loss = torch.stack(kld_fold_nodes).sum() / len(trees)loss = recon_loss + arg.kl_weight * kl_lossoptimizer.zero_grad()loss.backward()torch.nn.utils.clip_grad_norm_(list(encoder.parameters()) + list(decoder.parameters()), max_norm=1.0)optimizer.step()total_recon_loss += recon_loss.item()total_kl_loss += kl_loss.item()total_recon_loss /= len(dataloader)total_kl_loss /= len(dataloader)return total_recon_loss, total_kl_lossdef test(encoder, decoder, dataloader, arg, mode='Test'):encoder.eval()decoder.eval()all_results = []all_cd_results = []with torch.no_grad():for (trees, num_nodes, gt_graphs, file_names) in dataloader:test_enc_fold = Fold(arg.device)test_enc_fold_nodes = [encode_structure_fold(test_enc_fold, tree) for tree in trees]test_enc_fold_nodes = test_enc_fold.apply(encoder, [test_enc_fold_nodes])test_enc_fold_nodes = torch.split(test_enc_fold_nodes[0], 1, 0)recon_graphs = []for test_fold_node in test_enc_fold_nodes:test_root_code, _ = torch.chunk(test_fold_node, 2, 1)recon_tree = decode_testing(vector=test_root_code, max=50, decoder=decoder)recon_graph = recon_tree.to_graph(dec=True)recon_graphs.append(recon_graph)for i, (gt_graph, recon_graph) in enumerate(zip(gt_graphs, recon_graphs)):graph_to_ply(gt_graph, os.path.join(arg.log_dir, f'{file_names[i]}_gt.ply'))graph_to_ply(recon_graph, os.path.join(arg.log_dir, f'{file_names[i]}_pre.ply'))stats_eval_fn = get_stats_eval(arg)stats_results = stats_eval_fn(gt_graphs, recon_graphs)all_results.append(stats_results)eval_results = {key: np.mean([result[key] for result in all_results]) for key in all_results[0].keys()}logging.info(f"{mode} | " + " | ".join([f"{key}: {value:.4f}" for key, value in eval_results.items()]))return eval_resultsdef train_model(encoder, decoder, train_data, test_data, optimizer, arg):best_cd = 10writer, arg.log_dir = setup_logging(arg)scheduler = torch.optim.lr_scheduler.StepLR(optimizer, step_size=arg.lr_step_size, gamma=arg.lr_gamma)for epoch in range(arg.epochs):start_time = time.time()total_recon_loss, total_kl_loss = train_one_epoch(encoder, decoder, train_data, optimizer, arg)writer.add_scalar('Train/Recon_Loss', total_recon_loss, epoch)writer.add_scalar('Train/KL_Loss', total_kl_loss, epoch)if (epoch + 1) % arg.checkpoint == 0:eval_results_test = test(encoder, decoder, test_data, arg, mode='Test')eval_results_train = test(encoder, decoder, train_data, arg, mode='Train')for key, value in eval_results_test.items():writer.add_scalar(f'Test/{key}', value, epoch)for key, value in eval_results_train.items():writer.add_scalar(f'Train/{key}', value, epoch)epoch_cd = eval_results_test['chamfer_distance']if epoch_cd < best_cd:best_cd = epoch_cdtorch.save({'encoder': encoder.state_dict(), 'decoder': decoder.state_dict()},os.path.join(arg.log_dir, 'models', f'best_model.pth'))logging.info(f'New best model saved with Chamfer Distance: {best_cd:.4f}')torch.save({'encoder': encoder.state_dict(), 'decoder': decoder.state_dict()},os.path.join(arg.log_dir, 'models', f'{epoch}.pth'))scheduler.step()end_time = time.time()logging.info(f"Epoch [{epoch + 1}/{arg.epochs}] | Time: {end_time - start_time:.2f}s | "f"Recon Loss: {total_recon_loss:.4f} | KL Loss: {total_kl_loss:.4f}")return encoder, decoderif __name__ == '__main__':from config import argsfrom torch.utils.data import DataLoaderfrom utils.dataset import *from utils.utils import set_seedset_seed(args.seed)train_dataset = TreeDataset(args.dataset, args.data_path, is_train=True)test_dataset = TreeDataset(args.dataset, args.data_path, is_train=False)train_dataloader = DataLoader(train_dataset, batch_size=args.batch_size, num_workers=0, shuffle=True,collate_fn=coll_function)test_dataloader = DataLoader(test_dataset, batch_size=args.batch_size, num_workers=0, shuffle=True,collate_fn=coll_function)Encoder = RecursiveEncoder(input_size=args.input_size, feature_size=args.latent_size,hidden_size=args.hidden_size).to(args.device)Decoder = RecursiveDecoder(latent_size=args.latent_size, hidden_size=args.hidden_size,output_size=args.input_size, args=args).to(args.device)opt = torch.optim.Adam(list(Encoder.parameters()) + list(Decoder.parameters()), lr=args.lr)train_model(encoder=Encoder, decoder=Decoder, train_data=train_dataloader, test_data=test_dataloader,optimizer=opt, arg=args)```3. 第二阶段:`python train_seq.py````python
import logging
import os
import timeimport torch
from torch.utils.data import DataLoaderfrom utils.dataset import Skeleton
from evaluation.point_metric import evaluate_generation_condition, compute_l_d_c, evaluate_reconstruction
from model.model_seq import TransformerVAE
from test_seq import condition_gen
import utils.utils as utilsdef train(model, train_loader, test_loader, optimizer, args, writer):metrics = {'CD': 0, 'EMD': 0, 'MSE': 0,'MMD_lengths': 0, 'MMD_distances': 0, 'MMD_curvatures': 0,'JSD_lengths': 0, 'JSD_distances': 0, 'JSD_curvatures': 0}for epoch in range(1, args.epochs + 1):model.train()args.current_epoch = epochepoch_total_loss, epoch_recon_loss, epoch_kl_loss, epoch_length_loss, epoch_radius_loss = 0, 0, 0, 0, 0start_time = time.time()for i, (data, condition, filename) in enumerate(train_loader):pad_mask, tgt_mask = utils.generate_masks(data, args)data = data.to(args.device, dtype=torch.float32)condition = condition.to(args.device, dtype=torch.float32)pad_mask = pad_mask.to(args.device, dtype=torch.float32)tgt_mask = tgt_mask.to(args.device, dtype=torch.float32)true_radius = data[:, :, 3].to(args.device)recon, length_logit, radius, mu, log_var = model(data, condition, pad_mask, tgt_mask)recon_loss, kl_loss, length_loss, radius_loss = model.get_loss(recon, data, length_logit, mu, log_var,pad_mask, true_radius)total_loss = (args.recon_weight * recon_loss + args.kl_weight * kl_loss +length_loss * args.len_weight + radius_loss)optimizer.zero_grad()total_loss.backward()optimizer.step()epoch_total_loss += total_loss.item()epoch_recon_loss += recon_loss.item()epoch_length_loss += length_loss.item()epoch_kl_loss += kl_loss.item()epoch_radius_loss += radius_loss.item()num_batches = len(train_loader)epoch_total_loss /= num_batchesepoch_recon_loss /= num_batchesepoch_length_loss /= num_batchesepoch_kl_loss /= num_batchesepoch_radius_loss /= num_batchesepoch_time = time.time() - start_timelogging.info(f"Epoch [{epoch}/{args.epochs}], loss: {epoch_total_loss:.4f}, "f"recon_loss: {epoch_recon_loss:.4f}, len_loss: {epoch_length_loss:.4f}, "f"kl_loss: {epoch_kl_loss:.4f}, radius_loss: {epoch_radius_loss:.4f}, "f"time: {epoch_time:.2f}s")writer.add_scalar('Loss/total_loss', epoch_total_loss, epoch)writer.add_scalar('Loss/recon_loss', epoch_recon_loss, epoch)writer.add_scalar('Loss/length_loss', epoch_length_loss, epoch)writer.add_scalar('Loss/kl_loss', epoch_kl_loss, epoch)writer.add_scalar('Loss/radius_loss', epoch_radius_loss, epoch)if epoch % args.model_ckp == 0 or epoch == args.epochs:model_save_path = os.path.join(args.log_dir, f"model_epoch_{epoch}.pth")torch.save(model.state_dict(), model_save_path)if epoch % args.test_interval == 0:metrics, best_avg_metric = test(model, test_loader, args, writer, metrics, args.best_avg_metric)args.best_avg_metric = best_avg_metricdef test(model, test_loader, args, writer, metrics, best_avg_metric):model.eval()recon_keys = ['CD', 'EMD', 'MSE']evaluate_keys = ['MMD_lengths', 'MMD_distances', 'MMD_curvatures','JSD_lengths', 'JSD_distances', 'JSD_curvatures']with torch.no_grad():for batch, (test_data, test_condition, filename) in enumerate(test_loader):test_pad_mask, test_tgt_mask = utils.generate_masks(test_data, args)test_data = test_data.to(args.device, dtype=torch.float32)test_condition = test_condition.to(args.device, dtype=torch.float32)test_pad_mask = test_pad_mask.to(args.device, dtype=torch.float32)test_tgt_mask = test_tgt_mask.to(args.device, dtype=torch.float32)test_recon, _, _, _, _ = model(test_data, test_condition, test_pad_mask,test_tgt_mask)generation = condition_gen(model, test_condition, args, args.log_dir,num_samples=args.num_samples, save_files=False)recon_result = evaluate_reconstruction(test_data, test_recon)generated_condition = compute_l_d_c(generation)evaluate_results = evaluate_generation_condition(test_condition[:generated_condition.shape[0], :], generated_condition)if batch % 5 == 0:for i, file in enumerate(filename[:args.num_samples]):utils.numpy_to_ply(test_recon[i].cpu().detach().numpy(), args.log_dir + f'/{file}_recon.ply')utils.numpy_to_ply(test_data[i].cpu().detach().numpy(), args.log_dir + f'/{file}_refer.ply')utils.numpy_to_ply(generation[i], args.log_dir + f'/gen_{i}.ply')batch_results = {key: recon_result[key] for key in recon_keys}batch_results.update({key: evaluate_results[key] for key in evaluate_keys})metrics = utils.update_metrics(metrics, batch_results, len(test_loader))args.best_avg_metric = sum(metrics.values()) / len(metrics)if args.best_avg_metric < best_avg_metric:torch.save(model.state_dict(), os.path.join(args.log_dir, 'best_model.pth'))logging.info(f"Best model at epoch {args.current_epoch} with curvature {args.best_avg_metric:.4f}")for metric, avg_value in metrics.items():logging.info(f"Test Results - {metric}: {avg_value:.4f}")writer.add_scalar(f"eval/{metric}", avg_value, args.epochs)return metrics, args.best_avg_metricdef main(args):train_dataset = Skeleton(data_path=args.data_path, dataset_name=args.dataset, max_seq_len=args.max_seq_len,is_train=True)test_dataset = Skeleton(data_path=args.data_path, dataset_name=args.dataset, max_seq_len=args.max_seq_len,is_train=False)train_loader = DataLoader(train_dataset, batch_size=args.batch_size, shuffle=True, num_workers=0)test_loader = DataLoader(test_dataset, batch_size=args.batch_size, shuffle=True, num_workers=0)model = TransformerVAE(input_dim=args.input_dim, hidden_dim=args.hidden_dim, latent_dim=args.latent_dim,num_layers=args.num_layers, nhead=args.n_head, max_seq_len=args.max_seq_len,condition_dim=args.condition_dim).to(args.device)optimizer = torch.optim.Adam(model.parameters(), lr=args.lr)utils.set_seed(args.seed)writer, args.log_dir = utils.setup_logging(args)train(model, train_loader, test_loader, optimizer, args, writer)if __name__ == '__main__':from config import tree_argstree_args = tree_args()main(args=tree_args)print('Finished Training')
- 第三阶段:
python assembly/aseemble.py
import osfrom utils.utils import graph_to_ply, ply_to_graph
import utils_assemblydef assemble(tree_graphs, seq_args, assembly_args):model_seq = utils_assembly.load_model(tree_args=None, seq_args=seq_args, assembly_args=assembly_args)output_path = utils_assembly.create_result_folder(assembly_args, 'recon')for filename in os.listdir(input_folder):if filename.endswith('.ply'):input_path = os.path.join(input_folder, filename)tree_graph = ply_to_graph(input_path)skeleton = utils_assembly.traverse_and_paste_curves(tree_graph, model_seq, seq_args, assembly_args.dataset,target_distance=0.01)skeleton = utils_assembly.smooth_bifurcation_node(skeleton, iterations=10, smooth_factor=0.3)if skeleton.number_of_nodes() == 0:continuegraph_to_ply(skeleton, os.path.join(output_path, f'skeleton_{filename}'))return tree_graphsif __name__ == '__main__':from config import tree_args, seq_argsimport argparseparser = argparse.ArgumentParser(description="ASSEMBLY Arguments")parser.add_argument('--dataset', type=str, default='march', choices=['imagecas', 'march', 'cow'])parser.add_argument('--gen_num', type=int, default=50, help="Number of samples to generate")parser.add_argument('--mmd_distance', type=str, default='rbf', help="Type of MMD distance (e.g., 'rbf')")parser.add_argument('--max_subgraph', type=bool, default=True, help="Whether to use maximum subgraph")# File Pathsparser.add_argument('--result_path', type=str, default='./result/')parser.add_argument('--model_name', type=str, default=r'')parser.add_argument('--seq_path', type=str, default=r'')parser.add_argument('--max_seq_len', type=int, default=200, help="Maximum sequence length")assembly_arg = parser.parse_args()tree_arg = tree_args()seq_arg = seq_args()input_folder = r''assemble(input_folder, seq_arg, assembly_arg)
7. 参考文献
1. Achlioptas, P., Diamanti, O., Mitliagkas, I., Guibas, L.J.: Learning representations and generative models for 3d point clouds. In: Proceedings of the 35th International Conference on Machine Learning (ICML). pp. 40–49. PMLR (2018)2. Belli, D., Kipf, T.: Image-conditioned graph generation for road network extraction. arXiv preprint arXiv:1910.14388 (2019)3. Chang, A.X., Funkhouser, T., Guibas, L., Hanrahan, P., Huang, Q., Li, Z., Savarese, S., Savva, M., Song, S., Su, H., et al.: Shapenet: An information-rich 3d model repository. arXiv preprint arXiv:1512.03012 (2015)4. Chaudhuri, S., Ritchie, D., Wu, J., Xu, K., Zhang, H.: Learning generative models of 3d structures. In: Computer graphics forum. vol. 39, pp. 643–666. Wiley Online Library (2020)5. Dong, C., Xu, S., Dai, D., Zhang, Y., Zhang, C., Li, Z.: A novel multi-attention, multi-scale 3d deep network for coronary artery segmentation. Medical Image Analysis 85, 102745 (2023)6. Feldman, P., Fainstein, M., Siless, V., Delrieux, C., Iarussi, E.: Vesselvae: Recursive variational autoencoders for 3d blood vessel synthesis. In: International Conference on Medical Image Computing and Computer-Assisted Intervention. pp. 67–76. Springer (2023)7. Hamarneh, G., et al.: Vascusynth: Simulating vascular trees for generating volumetric image data with ground-truth segmentation and tree analysis. Computerized Medical Imaging and Graphics 34(8), 605–616 (2010)8. Hochmuth, A., Spetzger, U., Schumacher, M.: Comparison of three-dimensional rotational angiography with digital subtraction angiography in the assessment of ruptured cerebral aneurysms. American journal of neuroradiology 23(7), 11991205 (2002)9. Li, J., Niu, C., Xu, K.: Learning part generation and assembly for structure-aware shape synthesis. In: Proceedings of the AAAI conference on artificial intelligence. vol. 34, pp. 11362–11369 (2020)10. Li, J., Xu, K., Chaudhuri, S., Yumer, E., Zhang, H., Guibas, L.: Grass: Generative recursive autoencoders for shape structures. ACM Transactions on Graphics (TOG) 36(4), 1–14 (2017)11. Luo, S., Hu, W.: Diffusion probabilistic models for 3d point cloud generation. In: Proceedings of the IEEE/CVF conference on computer vision and pattern recognition. pp. 2837–2845 (2021)12. Lyu, X., Cheng, L., Zhang, S.: The reta benchmark for retinal vascular tree analysis. Scientific Data 9(1), 397 (2022)13. Mo, K., Guerrero, P., Yi, L., Su, H., Wonka, P., Mitra, N.J., Guibas, L.J.: Structurenet: hierarchical graph networks for 3d shape generation 38(6) (2019)14. Paetzold, J.C., McGinnis, J., Shit, S., Ezhov, I., Büschl, P., Prabhakar, C., Sekuboyina, A., Todorov, M., Kaissis, G., Ertürk, A., et al.: Whole brain vessel graphs: A dataset and benchmark for graph learning and neuroscience. In: Thirty-Fifth Conference on Neural Information Processing Systems Datasets and Benchmarks Track (2021)15. Prabhakar, C., Shit, S., Musio, F., Yang, K., Amiranashvili, T., Paetzold, J.C., Li, H.B., Menze, B.: 3d vessel graph generation using denoising diffusion. In: International Conference on Medical Image Computing and Computer-Assisted Intervention. pp. 3–13. Springer (2024)16. Schor, N., Katzir, O., Zhang, H., Cohen-Or, D.: Componet: Learning to generate the unseen by part synthesis and composition. In: Proceedings of the IEEE/CVF International Conference on Computer Vision. pp. 8759–8768 (2019)17. Sinha, A., Hamarneh, G.: TrIND: Representing Anatomical Trees by Denoising Diffusion of Implicit Neural Fields . In: proceedings of Medical Image Computing and Computer Assisted Intervention– MICCAI 2024. vol. LNCS 15012. Springer Nature Switzerland (October 2024)18. Socher, R., Huval, B., Bath, B., Manning, C.D., Ng, A.: Convolutional-recursive deep learning for 3d object classification. Advances in neural information processing systems 25 (2012)19. Socher, R., Lin, C.C., Manning, C., Ng, A.Y.: Parsing natural scenes and natural language with recursive neural networks. In: Proceedings of the 28th international conference on machine learning (ICML-11). pp. 129–136 (2011)20. Vahdat, A., Williams, F., Gojcic, Z., Litany, O., Fidler, S., Kreis, K., et al.: Lion: Latent point diffusion models for 3d shape generation. Advances in Neural Information Processing Systems 35, 10021–10039 (2022)21. Wang, Z., et al.: Cardiovascular medical image and analysis based on 3d vision: A comprehensive survey. Meta-Radiology 2(4), 100102 (2024)22. Wang, Z., Yi, R., Wen, X., Zhu, C., Xu, K.: Cardiovascular medical image and analysis based on 3d vision: A comprehensive survey. Meta-Radiology p. 100102 (2024)23. Wolterink, J.M., Leiner, T., Isgum, I.: Blood vessel geometry synthesis using generative adversarial networks. arXiv preprint arXiv:1804.04381 (2018)24. Xu, Q.C., Mu, T.J., Yang, Y.L.: A survey of deep learning-based 3d shape generation. Computational Visual Media 9(3), 407–442 (2023)25. Yang, G., Huang, X., Hao, Z., Liu, M.Y., Belongie, S., Hariharan, B.: Pointflow: 3d point cloud generation with continuous normalizing flows. In: Proceedings of the IEEE/CVF international conference on computer vision. pp. 4541–4550 (2019)26. Yang, K., Musio, F., Ma, Y., Juchler, N., Paetzold, J.C., Al-Maskari, R., Höher, L., Li, H.B., Hamamci, I.E., Sekuboyina, A., et al.: Benchmarking the cow with the topcow challenge: Topology-aware anatomical segmentation of the circle of willis for cta and mra. ArXiv pp. arXiv–2312 (2024)27. Yao, L., Shi, F., Wang, S., Zhang, X., Xue, Z., Cao, X., Zhan, Y., Chen, L., Chen, Y., Song, B., et al.: Tag-net: topology-aware graph network for centerline-based vessel labeling. IEEE transactions on medical imaging 42(11), 3155–3166 (2023)28. Zeng, A., Wu, C., Lin, G., Xie, W., Hong, J., Huang, M., Zhuang, J., Bi, S., Pan, D., Ullah, N., Khan, K.N., Wang, T., Shi, Y., Li, X., Xu, X.: Imagecas: A largescale dataset and benchmark for coronary artery segmentation based on computed tomography angiography images. Computerized Medical Imaging and Graphics 109, 102287 (2023)29. Zhang, G., Dong, C., Li, Y.: Topology-preserving hard pixel mining for tubular structure segmentation. In: 34th British Machine Vision Conference 2023, BMVC 2023, Aberdeen, UK, November 20-24, 2023. BMVA (2023)30. Zhang, G., Li, Y.: A geometric algorithm for blood vessel reconstruction from skeletal representation. In: International Symposium on Bioinformatics Research and Applications. pp. 114–126. Springer (2024)31. Zhao, J., Chen, X., Xiong, Z., Zha, Z.J., Wu, F.: Graph representation learning for large-scale neuronal morphological analysis. IEEE Transactions on Neural Networks and Learning Systems 35(4), 5461–5472 (2022)
版权说明:
本文由 youcans@xidian 对论文 一种用于生成逼真的3D血管的分层部件生成模型(Hierarchical Part-based Generative Model for Realistic 3D Blood Vessel) 进行摘编和翻译。该论文版权属于原文期刊和作者,本译文只供研究学习使用。
引用格式:
S. Chen, G. Zhang, J. Lai, et al. (2026). Hierarchical Part-Based Generative Model for Realistic 3D Blood Vessel. In: Gee, J.C., et al. Medical Image Computing and Computer Assisted Intervention – MICCAI 2025. MICCAI 2025. Lecture Notes in Computer Science, vol 15962. Springer, Cham. https://doi.org/10.1007/978-3-032-04947-6_25
youcans@xidian 作品,转载必须标注原文链接:
【医学影像 AI】一种用于生成逼真的3D血管的分层部件生成模型
(https://youcans.blog.csdn.net/article/details/153627635)
Crated:2025-10
