QIIME 2 2025.7:微生物数据分析再升级!
QIIME 2 2025.7
QIIME 2 2025.7 作为今年的功能版本,本次更新延续了团队一贯的高质量节奏,带来了多项插件增强、框架优化以及文档更新。
值得注意的是,QIIME 2 的版本发布节奏也更加清晰:每年 4 月和 10 月为“完整版本”,包含新功能、Bug 修复和依赖更新;而 1 月和 7 月则为“功能版本”,专注于新功能和修复,不涉及依赖变动。
在框架层面,未来两个完整版本将分别支持 Python 3.12(2025.10)和 Python 3.13(2026.4),为后续兼容性和性能打下基础。同时,缓存机制也得到了优化,旧版本遇到新缓存时将提示更明确的错误信息,提升使用体验。
插件方面,本次更新尤为丰富。比如 q2-alignment 插件新增了 --large 参数,优化了大文件比对时的内存使用; q2-annotate 支持 Kaiju 分类的拼接序列输入,并引入了 BUSCO 完整性与污染度指标; q2-assembly 则新增了模拟测序数据的功能,适合教学与方法开发。
以下是部分插件更新内容一览:
| 插件 | 更新亮点 |
|---|---|
q2-alignment | 支持 |
q2-annotate | 支持 Kaiju 拼接序列;新增 BUSCO 指标与 EC 编号提取 |
q2-assembly | 新增 Mason 模拟测序数据生成 |
q2-dada2 | denoise-paired 支持更多参数控制 |
q2-feature-classifier | 新增 |
q2-rescript | 可直接从 MIDORI 2 获取线粒体参考序列 |
q2-vizard | 图形支持自动缩放、交互式缩放、线宽调节等 |
此外,插件模板系统从 Cookiecutter 迁移至 Copier,未来将支持自动更新环境文件,方便插件开发者与主版本保持同步。文档方面也新增了通过 Galaxy + Docker 使用 QIIME 2 的说明,进一步降低入门门槛。
本次更新不仅提升了分析效率,也增强了可视化与数据处理的灵活性。对于从事微生物组、环境基因组或临床微生态研究的科研人员而言,QIIME 2 正在不断拓展其边界,成为不可或缺的分析平台。
关于QIIME 2

QIIME 2 是一个专为微生物组和多组学数据分析打造的开源平台,它不仅免费、可扩展,而且由全球科研社区共同开发与维护。这个平台的最大特点是“AI 就绪”,意味着它在每一步分析中都自动记录数据来源,包括使用的命令、参数和计算环境。这种机制确保了分析的可追溯性和可重复性,使得科研数据更具透明度和可信度。
在使用方式上,QIIME 2 提供了多种接口,满足不同背景研究者的需求。无论是偏好命令行的生信人员,还是习惯图形界面的初学者,都可以通过 CLI、Galaxy GUI 或 Python/R API 高效完成分析任务。平台集成了数十个插件和数百种方法,支持从扩增子到宏基因组的多种数据类型处理,并且可视化功能强大,交互性良好,适合科研展示与结果解读。
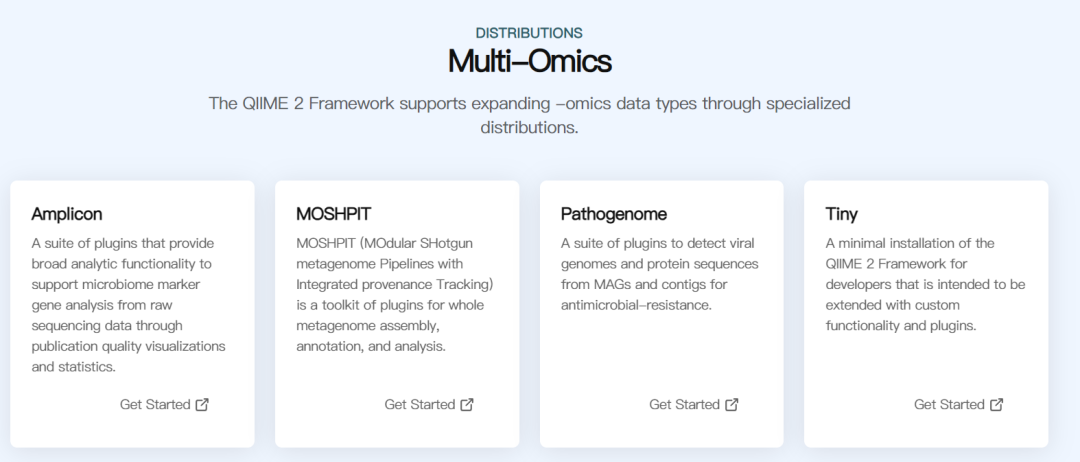
QIIME 2 的架构采用模块化设计,插件系统使得功能扩展变得非常灵活。研究者可以根据自己的需求开发新插件,或组合已有插件形成定制化工作流。目前平台已推出多个“分布版”,例如扩增子分析套件、MOSHPIT 宏基因组工具包、致病基因组检测模块,以及面向开发者的最小框架版,覆盖了从基础研究到临床应用的广泛场景。
值得一提的是,QIIME 2 的数据格式采用 .qza 和 .qzv 压缩文件,内部不仅包含分析结果,还嵌入了完整的分析过程信息。这种设计让数据本身就具备“自我说明”的能力,即使脱离平台,也能准确还原其生成路径。
整个项目由 NIH、CZI、NSF 等多个机构资助,社区活跃度高,论坛中聚集了大量开发者与用户,分享经验、发布插件、解决问题。对于希望深入参与的研究者,无论是否具备编程背景,都有明确的参与路径和贡献指南。
参考:
-
https://qiime2.org/
-
https://qiime2.org/news/qiime-2-2025-7-is-now-available-33447/
