蛋白质结构预测:从AlphaFold到未来的计算生物学革命

🌟 Hello,我是蒋星熠Jaxonic!
🌈 在浩瀚无垠的技术宇宙中,我是一名执着的星际旅人,用代码绘制探索的轨迹。
🚀 每一个算法都是我点燃的推进器,每一行代码都是我航行的星图。
🔭 每一次性能优化都是我的天文望远镜,每一次架构设计都是我的引力弹弓。
🎻 在数字世界的协奏曲中,我既是作曲家也是首席乐手。让我们携手,在二进制星河中谱写属于极客的壮丽诗篇!
摘要
我们从一条短短的氨基酸序列出发,试图重建其在三维空间中蜿蜒折叠的优雅姿态,这既是工程挑战,也是科学浪漫。传统基于物理的能量最小化方法如同在真空中计算行星轨道,需要精密的势函数与庞大的计算资源;同源建模则像借助星图导航,依赖已知结构的指引与比对精度;而以 AlphaFold2 为代表的深度学习方法则像引入了新的推进器,通过多序列比对(MSA)、注意力机制与几何约束,将进化信息与结构知识高效融合,实现从序列到结构的跃迁。站在工程视角,我更关心每一条数据管线的稳定性、每一个特征模块的可复用性,以及如何在资源受限的前提下保证实验的可重复。为此,本文不仅会展示从序列到结构的完整流程,还将强调模块化、约束化与可解释化的三大原则:模块化让复杂问题可被拆解,约束化让预测结果尊重几何与物理常识,可解释化则为每一个关键决策提供可视证据。在实践中,我们会权衡RMSD、TM-score与pLDDT等指标,建立面向科研与工业的“可度量即可管控”的方法论。无论你是第一次迈入结构预测的航道,还是准备将其嵌入药物研发的生产级系统,我都希望这篇文章能成为你可靠的导航星,帮助你在分子宇宙中稳健前行。
目录
- 背景与动机
- 方法综述与关键公式
- 物理驱动方法
- 同源建模策略
- 深度学习路线
- 工程化架构与数据流
- 特征工程与可解释性
- 序列层面
- 共进化信息
- 次级结构
- 三维几何
- 代码示例一:从序列到接触图(Contact Map)
- 代码示例二:轻量注意力嵌入与几何约束
- 评估指标与可视化
- 性能与资源权衡:表格对比
- 部署与应用场景
- 引用与箴言
- 总结(蒋星熠Jaxonic)
- 参考链接与关键词标签
背景与动机
蛋白质结构决定功能。预测结构不仅是理解生命机制的关键,也是药物设计、蛋白工程与合成生物学的重要抓手。过去,X 射线晶体学、核磁共振等实验方法成本高昂且周期长;计算预测成为加速创新的技术基座。随着深度学习与几何推理的结合,结构预测步入了高精度时代,但工程落地仍需要处理数据质量、计算资源、可解释性与部署稳定性。
方法综述与关键公式
物理驱动方法
- 基于势能函数 U(Φ) 与分子动力学(MD)或蒙特卡洛(MC)搜索最小能量构型。
- 优点:物理可解释性强,适合局部精炼与小分子相互作用评估。
- 缺点:计算耗时,参数敏感,初始构象与势函数选择影响大。
同源建模策略
- 通过序列比对(BLAST、HHblits)检索模板结构,构建保守域的三维模型。
- 优点:快速、稳健,适合保守蛋白与家族内预测。
- 缺点:模板依赖严重,对新颖拓扑与低同源序列不友好。
深度学习路线
- 代表:AlphaFold2、RoseTTAFold,融合 MSA、注意力、迭代几何约束。
- 优点:端到端高精度,适合复杂拓扑与长程依赖。
- 缺点:资源需求较高,模型推理与特征构建成本显著。
常用评估公式(RMSD):
RMSD = sqrt( (1/N) * Σ_i || x_i - y_i ||^2 )
其中 x_i、y_i 为对应原子坐标,N 为匹配原子数。TM-score 更适合跨长度比较,范围约在(0,1]。
工程化架构与数据流
图1:端到端结构预测流水线(flowchart,展示从序列到结构的流程)
图2:数据-模型交互时序(sequenceDiagram,展示请求、计算与返回)
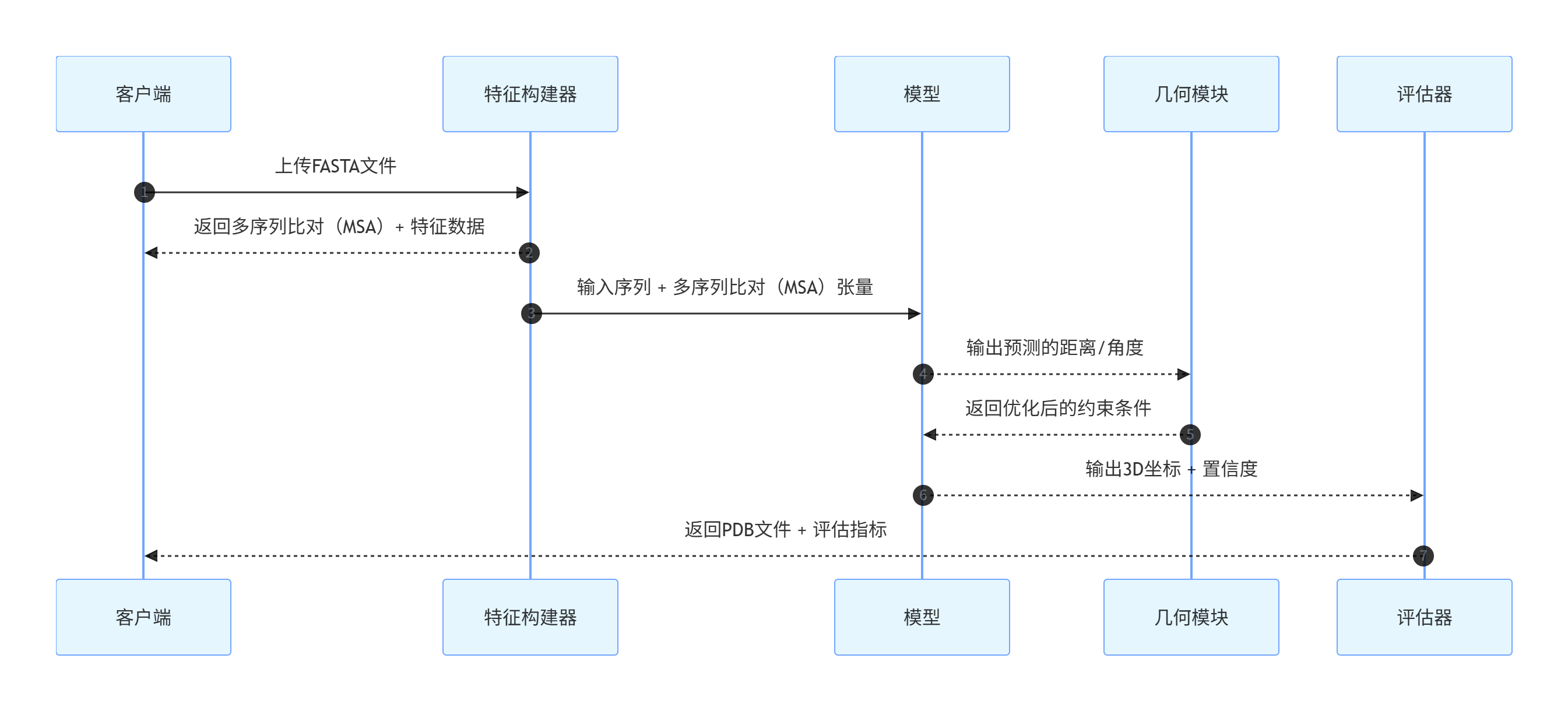
图3:系统组件架构(英文数据点)
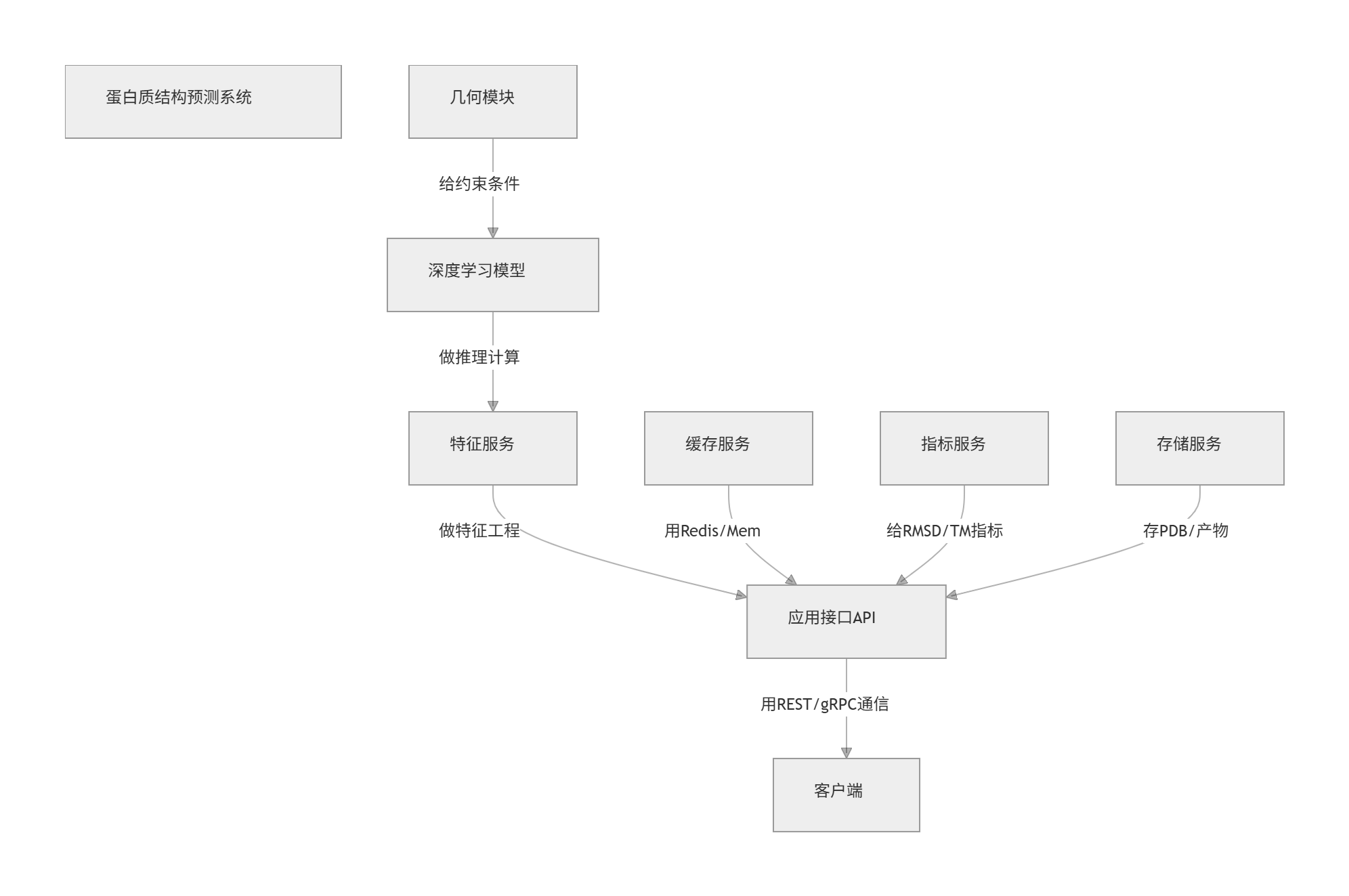
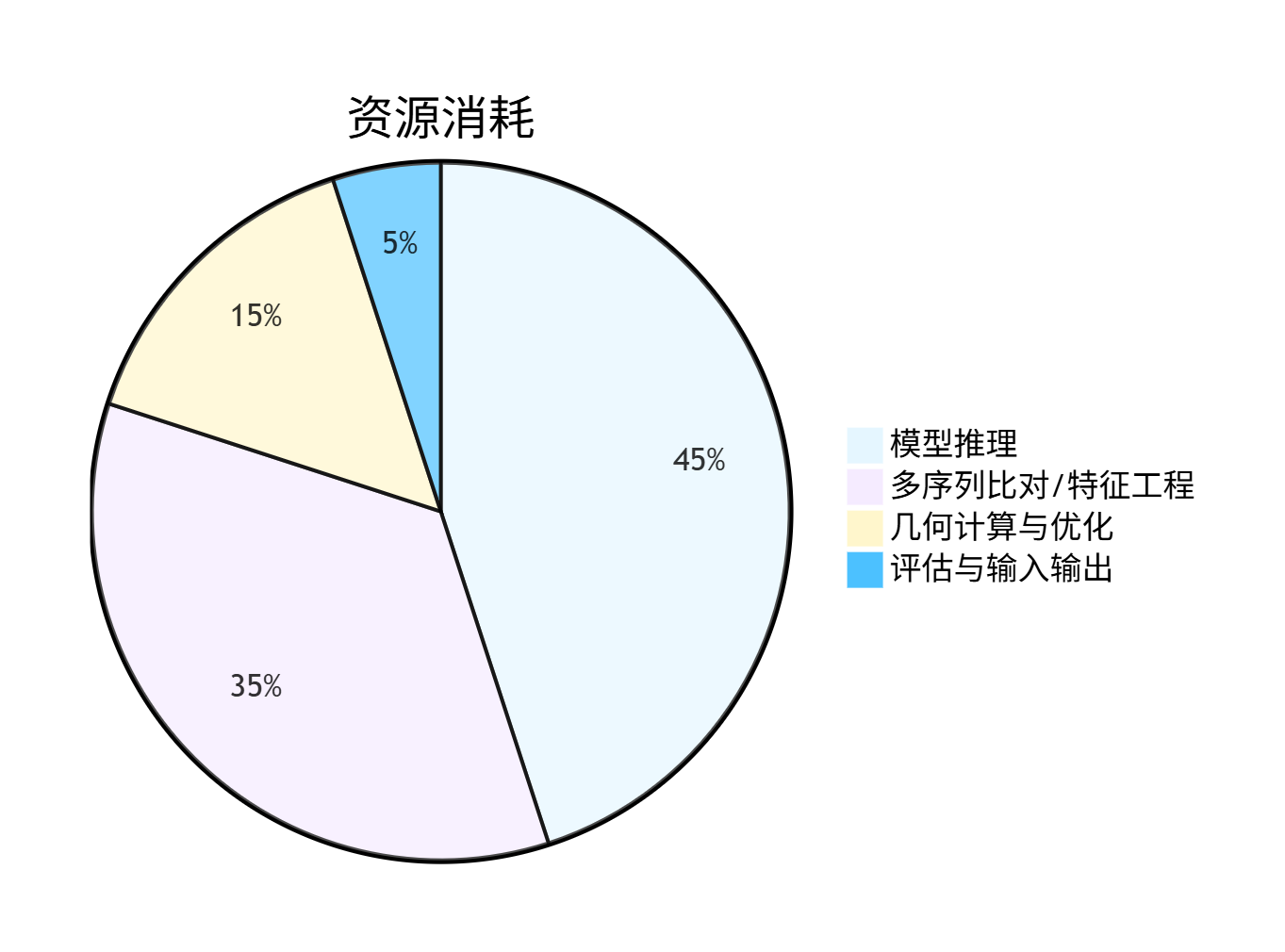
图4:资源占比(pie,展示训练/推理/特征占用)
特征工程与可解释性
序列层面
- One-hot 编码、氨基酸理化性质(疏水性、体积、带电)、位点权重与位置编码。
共进化信息
- 基于 MSA 的协变矩阵与互信息,揭示可能的长程接触对。
次级结构
- 轻量模型预测 Helix/Sheet/Coil,用作几何先验与约束。
三维几何
-
距离矩阵、二面角、刚体框架,保证物理合理性与拓扑一致。
-
可解释性:注意力热图与接触概率可视化,辅助定位关键位点。
代码示例一:从序列到接触图(Contact Map)
意图:给定 FASTA 序列与简化的氨基酸相互作用评分,生成一个粗略的接触概率矩阵,为后续几何约束与建模提供初步参考。示例采用 NumPy,配合注释强调工程要点。
# contact_map.py
# 目的:从FASTA序列和简化特征估计残基-残基接触概率
# 说明:示例为教学用途,未依赖MSA;实际工程建议加入协变信息import numpy as npAA = "ACDEFGHIKLMNPQRSTVWY"
# 简化氨基酸相容矩阵(示例值),现实可来源于PSSM或经验统计
compat = np.random.RandomState(42).rand(len(AA), len(AA))def read_fasta(fasta_str: str) -> str:lines = [ln.strip() for ln in fasta_str.splitlines() if ln.strip()]seq = "".join([ln for ln in lines if not ln.startswith(">")])return seqdef one_hot(seq: str) -> np.ndarray:idx = {a:i for i,a in enumerate(AA)}X = np.zeros((len(seq), len(AA)), dtype=np.float32)for i, ch in enumerate(seq):if ch in idx: X[i, idx[ch]] = 1.0return Xdef pair_score(X: np.ndarray) -> np.ndarray:# 计算两两残基的相容分数,归一化到[0,1]n = X.shape[0]S = np.zeros((n, n), dtype=np.float32)for i in range(n):for j in range(i+1, n):s = X[i] @ compat @ X[j].TS[i, j] = S[j, i] = s# 归一化S -= S.min()if S.max() > 0: S /= S.max()return Sdef distance_bias(n: int, lam: float = 0.02) -> np.ndarray:# 基于序列距离的先验:过近/过远的残基接触概率较低D = np.zeros((n, n), dtype=np.float32)for i in range(n):for j in range(n):if i == j: D[i, j] = 0.0else:gap = abs(i - j)D[i, j] = np.exp(-lam * gap)return Ddef contact_map(fasta_str: str) -> np.ndarray:seq = read_fasta(fasta_str)X = one_hot(seq)S = pair_score(X)D = distance_bias(len(seq))# 融合:相容分数 * 序列距离先验C = S * Dreturn Cif __name__ == "__main__":fasta = """>toy
MKTAYIAKQRQISFVKSHFSRQLEERLGLIEVQ"""C = contact_map(fasta)# 阈值化得到可能接触对contacts = np.argwhere(C > 0.6)print("contact candidates:", contacts.tolist())
关键行点评:
- compat 相容矩阵用于残基对的可相容性估计,可由 PSSM 或统计能量替换。
- distance_bias 提供跨序列距离的先验,避免短程伪接触。
- 融合策略(S * D)是可解释与可替换的模块接口,便于工程迭代。
代码示例二:轻量注意力嵌入与几何约束
意图:以 PyTorch 构建简化的注意力嵌入层,输出残基表征,并用三角不等式约束矫正距离矩阵。示例强调模块化与几何校正接口。
# lite_embed_geom.py
# 目的:用简化注意力生成残基嵌入,并进行距离矩阵几何校正
# 说明:教学代码,真实系统应引入MSA注意力与框架几何import torch
import torch.nn as nn
import torch.nn.functional as Fclass LiteEncoder(nn.Module):def __init__(self, dim=64, heads=4):super().__init__()self.emb = nn.Embedding(20, dim)self.attn = nn.MultiheadAttention(dim, heads, batch_first=True)self.ffn = nn.Sequential(nn.LayerNorm(dim),nn.Linear(dim, dim*4),nn.GELU(),nn.Linear(dim*4, dim))def forward(self, x_idx):x = self.emb(x_idx) # [B, L, D]y, _ = self.attn(x, x, x) # 自注意力z = self.ffn(y) + x # 残差return z # [B, L, D]def pair_dist(h: torch.Tensor) -> torch.Tensor:# 以嵌入间的欧氏距离作为proxy(真实应映射到物理坐标)B, L, D = h.shapeh = F.normalize(h, dim=-1)diff = h[:, :, None, :] - h[:, None, :, :]dist = torch.sqrt(torch.sum(diff*diff, dim=-1) + 1e-8)return dist # [B, L, L]def triangle_inequality(dist: torch.Tensor) -> torch.Tensor:# 简化三角不等式校正:d_ij <= d_ik + d_kjB, L, _ = dist.shapefor k in range(L):ik = dist[:, :, k]kj = dist[:, k, :]bound = ik[:, :, None] + kj[:, None, :]dist = torch.minimum(dist, bound)return distif __name__ == "__main__":# toy: 将序列映射到索引(A=0,...,Y=19)seq_idx = torch.randint(0, 20, (1, 32))enc = LiteEncoder(dim=64, heads=4)h = enc(seq_idx)d = pair_dist(h)d_corr = triangle_inequality(d)print("raw mean dist:", d.mean().item())print("corrected mean dist:", d_corr.mean().item())
关键行点评:
- LiteEncoder 用残差 + 多头注意力,模拟序列长程依赖的表征能力。
- pair_dist 使用嵌入间距离作为代理,真实工程需映射到坐标或用专用几何头。
- triangle_inequality 提供几何一致性约束接口,可扩展到角度与框架校正。
评估指标与可视化
- RMSD:整体结构偏差,受局部异常影响较大。
- TM-score:长度不敏感,更能反映拓扑一致性。
- pLDDT:模型对局部残基坐标的置信度(AlphaFold2 输出)。
可视化建议:
- 接触图热力图:揭示长程接触预测质量。
- 注意力权重:定位结构关键位点与支撑证据。
- 残基置信度分布:筛选可信构象与可疑区域。
性能与资源权衡:方案对比表
| 方案 | 准确性 | 可解释性 | 计算成本 | 适用场景 |
|---|---|---|---|---|
| 物理驱动仿真 | 高 | 中 | 很高 | 小规模高精度、局部精炼 |
| 同源建模 | 中-高 | 高 | 低-中 | 有模板、保守域 |
| 深度学习端到端 | 很高 | 中 | 中-高 | 大量序列、复杂拓扑 |
| 轻量混合(本文示例) | 中 | 高 | 低 | 快速原型、可解释性优先 |
部署与应用场景
- 批量预测服务:REST/gRPC 接入,Redis 缓存 MSA 与特征,MinIO 存储 PDB。
- 可视化平台:集成热力图、注意力、置信度面板,支持交互分析。
- 药物筛选:与结合位点预测耦合,快速评估突变影响。
- 教学与科研:轻量模块可复用,便于迭代与发表复现。
引用与箴言
“在复杂系统中追求简单、在不确定数据中保证稳健:这是工程与科学在蛋白质结构预测上的共同信条。”
数学补充:约束优化直觉
在几何一致性中,常用的约束优化目标可表示为:
minimize Σ_ij w_ij |d_ij - \hat{d}_ij| + λ·R(Θ)
其中 d_ij 为预测距离,\hat{d}_ij 为校正后或先验距离,R(Θ) 为正则项(例如平滑或框架一致性),λ 为权衡系数。这个目标兼顾精度与几何物理合理性。
总结
一条序列仿佛一条未标注的星际航线,我们要在进化的星图上定位参照点,在注意力的引力场里校准方向,用几何约束去抵达更稳健的坐标。在本文的实践里,我将复杂系统拆解为可复用的模块,将“黑盒”抽象为透明的接口,并以 RMSD、TM-score、pLDDT 织就一张可度量的观测网,使每次预测都能被比较、被解释、被持续改进。工程层面,我们强调数据质量与缓存策略,避免 MSAs 的重复开销;模型层面,我们强调轻量与可插拔,让不同精度需求在同一框架下自由切换;几何层面,我们引入三角不等式与框架一致性,逐步将表征空间映射回物理空间。未来,我希望进一步将注意力热图与接触概率联合优化,把置信度分布与功能位点标注联动,让结构预测与功能假设相互验证,构成闭环的科研工程体系。作为蒋星熠Jaxonic,我会继续在这条星际航线上迭代我的导航图,与同行者共创更优雅的模型、更透明的流程、更可靠的系统,让分子世界的宏大叙事在每一次推理中发光,在每一次部署中落地。
■ 我是蒋星熠Jaxonic!如果这篇文章在你的技术成长路上留下了印记
■ 👁 【关注】与我一起探索技术的无限可能,见证每一次突破
■ 👍 【点赞】为优质技术内容点亮明灯,传递知识的力量
■ 🔖 【收藏】将精华内容珍藏,随时回顾技术要点
■ 💬 【评论】分享你的独特见解,让思维碰撞出智慧火花
■ 🗳 【投票】用你的选择为技术社区贡献一份力量
■ 技术路漫漫,让我们携手前行,在代码的世界里摘取属于程序员的那片星辰大海!
参考链接
- https://www.nature.com/articles/s41586-021-03819-2
- https://github.com/deepmind/alphafold
- https://www.rcsb.org/
- https://www.ebi.ac.uk/services
- https://www.nature.com/articles/s41586-021-04043-4
