【Block总结】LAGA,大核分组注意力门,实现特征融合|即插即用
论文信息
- 标题:EMCAD: Efficient Multi-scale Convolutional Attention Decoding for Medical Image Segmentation(EMCAD:用于医学图像分割的高效多尺度卷积注意力解码器)
- 作者:Md Mostafijur Rahman, Mustafa Munir, Radu Marculescu
- 论文链接:https://arxiv.org/pdf/2405.06880
- 核心问题:现有的医学图像分割模型,尤其是结合了注意力机制的解码器,往往计算成本高昂(参数量大、FLOPs高),难以在计算资源受限的场景(如边缘设备、实时诊断)中部署。
- 核心贡献:提出了一种名为 EMCAD 的新型高效解码器,旨在同时优化分割性能和计算效率。该解码器可以与各种分层视觉编码器(如CNN或Transformer)配合使用。
创新点
论文提出了三个主要的创新模块,共同构成了高效的EMCAD解码器:
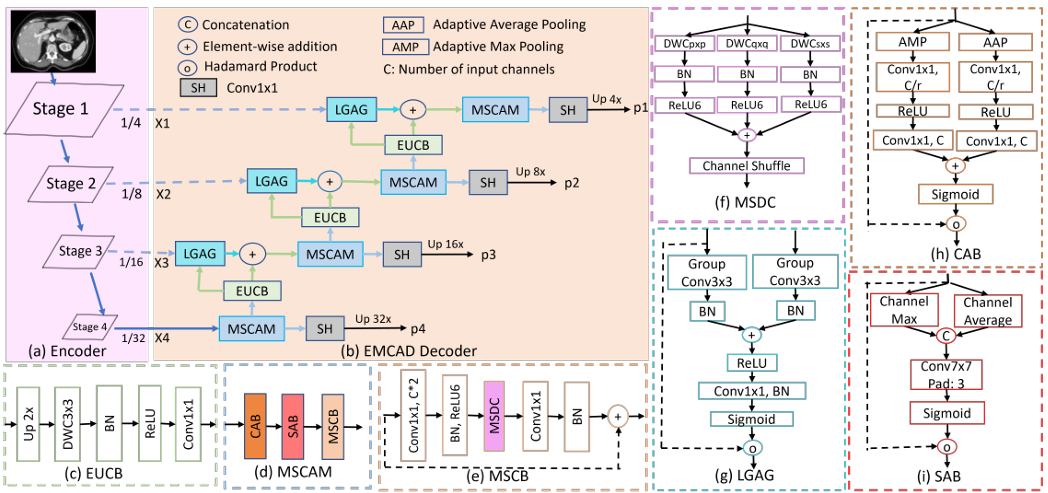
- 高效的多尺度卷积注意力模块 (MSCAM):该模块使用 多尺度深度可分离卷积 (Depth-wise Convolution) 来增强特征图,能够有效捕捉多尺度显著特征,同时通过通道注意力和空间注意力机制来聚焦关键区域。深度可分离卷积的使用是其高效的关键。
- 大核分组注意力门 (LGAG):这是一种新颖的特征融合机制。与传统的注意力门(如Attention U-Net中使用的1x1卷积)不同,LGAG在处理门控信号和输入特征时,采用了3x3的大核分组卷积。这使得模型能在更大的局部上下文中捕捉显著特征,且计算开销更低。
- 高效的上卷积块(EUCB):用于上采样特征图。它使用3x3深度可分离卷积替代了标准的3x3卷积,显著降低了上采样过程中的计算成本。
方法
EMCAD的整体架构是一个级联的全卷积注意力解码器,其工作流程如下:
- 输入:接收来自预训练分层视觉编码器(如PVTv2)的四个阶段(X1, X2, X3, X4)的多尺度特征图。
- 特征精炼:每个阶段的特征图首先通过一个 MSCAM 模块进行精炼,该模块依次应用通道注意力(CAB)、空间注意力(SAB)和多尺度卷积块(MSCB)。
- 中间输出:精炼后的每个阶段特征图都会通过一个**分割头 **(SH) 生成一个中间分割图(p1, p2, p3, p4)。
- 上采样与融合:
- 从最深层(X4)开始,精炼后的特征图通过 EUCB 进行上采样。
- 上采样后的特征图与来自编码器对应层的跳跃连接(Skip Connection)特征图一起送入 LGAG 模块。
- LGAG利用高层特征作为门控信号,对跳跃连接的特征进行加权,然后与上采样特征相加,实现高效的特征融合。
- 级联传递:融合后的特征图作为下一级的输入,重复步骤2-4,直至生成最终的高分辨率特征图。
- 最终输出与损失:论文将最后一个阶段(p4)的分割图作为最终输出。在训练时,采用多阶段损失策略,不仅计算最终输出的损失,还计算所有中间分割图(p1-p4)以及它们的加和的损失,以提供更丰富的监督信号。
LGAG(Large-kernel Grouped Attention Gate)模块详解
LGAG 是 EMCAD 解码器中用于高效融合上采样特征与跳跃连接(skip connection)特征的关键模块。其设计目标是在保留显著区域信息的同时,显著降低计算开销,特别适用于医学图像分割中对局部上下文敏感的场景。
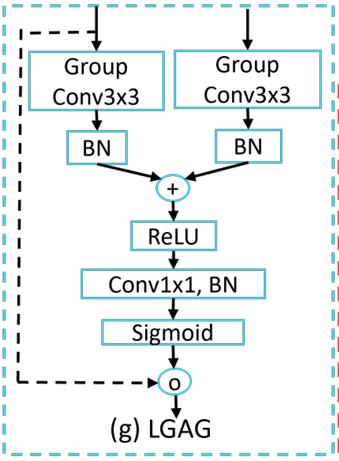
1. 设计动机
传统注意力门(如 Attention U-Net 中的 AG)通常使用 1×1 卷积来处理门控信号(gating signal)和输入特征,这种方式感受野有限,难以捕捉局部空间上下文信息。
LGAG 的创新在于:
- 使用 3×3 大核卷积
- 引入 分组卷积(Grouped Convolution)
这使得 LGAG 能在更大的局部感受野中建模空间依赖关系,同时通过分组策略大幅降低参数量和计算量。
2. 模块结构与计算流程
LGAG 接收两个输入:
- g:来自高层(上采样路径)的门控信号(gating signal)
- x:来自编码器对应层的跳跃连接特征
其计算分为以下几步(对应论文公式 1 和 2):
步骤 1:分别对 g 和 x 进行 3×3 分组卷积
- 对门控信号 g:应用分组卷积 GCg(g)GC_g(g)GCg(g)
- 对跳跃特征 x:应用分组卷积 GCx(x)GC_x(x)GCx(x)
步骤 2:批归一化(BN)与融合
- 对 GCx(x)GC_x(x)GCx(x) 先做 BN
- 将 GCg(g)GC_g(g)GCg(g) 与 BN(GCx(x))BN(GC_x(x))BN(GCx(x)) 逐元素相加
- 对相加结果再做 BN,并通过 ReLU 激活
该过程定义为:
qatt(g,x)=ReLU(BN(GCg(g)+BN(GCx(x))))
q_{att}(g, x) = \text{ReLU}\left( \text{BN}\left( GC_g(g) + \text{BN}(GC_x(x)) \right) \right)
qatt(g,x)=ReLU(BN(GCg(g)+BN(GCx(x))))
步骤 3:生成注意力权重
- 对 qatt(g,x)q_{att}(g, x)qatt(g,x) 应用 1×1 卷积 + BN,将其压缩为单通道特征图
- 通过 Sigmoid 激活函数生成注意力权重 α∈[0,1]\alpha \in [0,1]α∈[0,1]
步骤 4:加权输出
- 将注意力权重 α\alphaα 与原始输入特征 x 进行逐元素相乘(Hadamard product)
- 得到最终的门控输出:
LGAG(g,x)=x⊛σ(BN(C(qatt(g,x)))) \text{LGAG}(g, x) = x \circledast \sigma\left( \text{BN}(C(q_{att}(g, x))) \right) LGAG(g,x)=x⊛σ(BN(C(qatt(g,x))))
其中:
- C(⋅)C(\cdot)C(⋅) 表示 1×1 卷积
- σ(⋅)\sigma(\cdot)σ(⋅) 为 Sigmoid 函数
- ⊛\circledast⊛ 表示逐元素相乘
3. 关键优势
| 特性 | 说明 |
|---|---|
| 大感受野 | 3×3 卷积相比 1×1 能捕获更大局部上下文,有助于精确定位医学结构边界 |
| 计算高效 | 分组卷积显著减少参数量和 FLOPs(见补充材料 Table 8:相比传统 AG,LGAG 参数减少 82.6%~91.2%) |
| 可扩展性强 | 在通道数增加时(如 B2 模型),LGAG 的效率优势更加明显 |
| 性能提升 | 在 Synapse 数据集上,使用 LGAG 比传统 AG 提升 DICE 分数(B0: 81.97 vs 81.74;B2: 83.63 vs 83.51) |
代码
import torch
import torch.nn as nn
from timm.models.helpers import named_apply
from functools import partial
from timm.models.layers import trunc_normal_tf_# Other types of layers can go here (e.g., nn.Linear, etc.)
def _init_weights(module, name, scheme=''):if isinstance(module, nn.Conv2d) or isinstance(module, nn.Conv3d):if scheme == 'normal':nn.init.normal_(module.weight, std=.02)if module.bias is not None:nn.init.zeros_(module.bias)elif scheme == 'trunc_normal':trunc_normal_tf_(module.weight, std=.02)if module.bias is not None:nn.init.zeros_(module.bias)elif scheme == 'xavier_normal':nn.init.xavier_normal_(module.weight)if module.bias is not None:nn.init.zeros_(module.bias)elif scheme == 'kaiming_normal':nn.init.kaiming_normal_(module.weight, mode='fan_out', nonlinearity='relu')if module.bias is not None:nn.init.zeros_(module.bias)else:# efficientnet likefan_out = module.kernel_size[0] * module.kernel_size[1] * module.out_channelsfan_out //= module.groupsnn.init.normal_(module.weight, 0, math.sqrt(2.0 / fan_out))if module.bias is not None:nn.init.zeros_(module.bias)elif isinstance(module, nn.BatchNorm2d) or isinstance(module, nn.BatchNorm3d):nn.init.constant_(module.weight, 1)nn.init.constant_(module.bias, 0)elif isinstance(module, nn.LayerNorm):nn.init.constant_(module.weight, 1)nn.init.constant_(module.bias, 0)def act_layer(act, inplace=False, neg_slope=0.2, n_prelu=1):# activation layeract = act.lower()if act == 'relu':layer = nn.ReLU(inplace)elif act == 'relu6':layer = nn.ReLU6(inplace)elif act == 'leakyrelu':layer = nn.LeakyReLU(neg_slope, inplace)elif act == 'prelu':layer = nn.PReLU(num_parameters=n_prelu, init=neg_slope)elif act == 'gelu':layer = nn.GELU()elif act == 'hswish':layer = nn.Hardswish(inplace)else:raise NotImplementedError('activation layer [%s] is not found' % act)return layerdef channel_shuffle(x, groups):batchsize, num_channels, height, width = x.data.size()channels_per_group = num_channels // groups# reshapex = x.view(batchsize, groups,channels_per_group, height, width)x = torch.transpose(x, 1, 2).contiguous()# flattenx = x.view(batchsize, -1, height, width)return x# Efficient up-convolution block (EUCB)
class EUCB(nn.Module):def __init__(self, in_channels, out_channels, kernel_size=3, stride=1, activation='relu'):super(EUCB, self).__init__()self.in_channels = in_channelsself.out_channels = out_channelsself.up_dwc = nn.Sequential(nn.Upsample(scale_factor=2),nn.Conv2d(in_channels, in_channels, kernel_size=kernel_size, stride=stride,padding=kernel_size // 2, groups=in_channels, bias=False),nn.BatchNorm2d(in_channels),act_layer(activation, inplace=True))self.pwc = nn.Sequential(nn.Conv2d(in_channels, out_channels, kernel_size=1, stride=1, padding=0, bias=True))self.init_weights('normal')def init_weights(self, scheme=''):named_apply(partial(_init_weights, scheme=scheme), self)def forward(self, x):x = self.up_dwc(x)x = channel_shuffle(x, self.in_channels)x = self.pwc(x)return x# Large-kernel grouped attention gate (LGAG)
class LGAG(nn.Module):def __init__(self, F_g, F_l, F_int, kernel_size=3, groups=1, activation='relu'):super(LGAG, self).__init__()if kernel_size == 1:groups = 1self.W_g = nn.Sequential(nn.Conv2d(F_g, F_int, kernel_size=kernel_size, stride=1, padding=kernel_size // 2, groups=groups,bias=True),nn.BatchNorm2d(F_int))self.W_x = nn.Sequential(nn.Conv2d(F_l, F_int, kernel_size=kernel_size, stride=1, padding=kernel_size // 2, groups=groups,bias=True),nn.BatchNorm2d(F_int))self.psi = nn.Sequential(nn.Conv2d(F_int, 1, kernel_size=1, stride=1, padding=0, bias=True),nn.BatchNorm2d(1),nn.Sigmoid())self.activation = act_layer(activation, inplace=True)self.init_weights('normal')def init_weights(self, scheme=''):named_apply(partial(_init_weights, scheme=scheme), self)def forward(self, g, x):g1 = self.W_g(g)x1 = self.W_x(x)psi = self.activation(g1 + x1)psi = self.psi(psi)return x * psiif __name__ == "__main__":# 定义输入张量大小(Batch、Channel、Height、Wight)B, C, H, W = 16, 64, 40, 40input_tensor = torch.randn(B, C, H, W) # 随机生成输入张量input_tensor1 = torch.randn(B, C, H, W) # 随机生成输入张量dim = C# 创建 ARConv 实例block = LGAG(dim,dim,dim)device = torch.device("cuda" if torch.cuda.is_available() else "cpu")sablock = block.to(device)print(sablock)input_tensor = input_tensor.to(device)input_tensor1 = input_tensor1.to(device)# 执行前向传播output = sablock(input_tensor,input_tensor1)# 打印输入和输出的形状print(f"Input: {input_tensor.shape}")print(f"Output: {output.shape}")
效果
论文在12个涵盖6种不同医学图像分割任务(息肉、皮肤病变、细胞、乳腺癌、腹部器官、心脏器官)的公共数据集上进行了广泛的实验,结果证明了EMCAD的优越性:
- 性能领先:提出的 PVT-EMCAD-B2 模型在多个数据集上达到了新的SOTA(State-of-the-Art)性能。例如:
- 在10个二值分割数据集上的平均DICE得分为 91.10%。
- 在Synapse腹部多器官分割数据集上的平均DICE得分为 83.63%。
- 在ACDC心脏分割数据集上的平均DICE得分为 92.12%。
- 效率极高:EMCAD在取得优异性能的同时,计算成本极低。
- 与强大的基线模型 PVT-CASCADE 相比,EMCAD在Synapse数据集上以 79.4%更少的参数(1.91M vs 9.27M)和 80.3%更少的FLOPs(0.381G vs 1.93G)实现了更高的DICE分数(83.63% vs 82.78%)。
- 其小型版本 PVT-EMCAD-B0 仅需 3.92M参数和 0.84G FLOPs,性能就远超许多现有方法。
- 通用性强:EMCAD可以无缝集成到不同的分层编码器(如PVTv2-B0, PVTv2-B2)中,并在各种分割任务上均表现出色,证明了其良好的通用性和适应性。
总结
EMCAD论文成功地解决了一个医学图像分割领域的关键痛点:如何在有限的计算资源下实现高性能分割。其核心思想是通过深度可分离卷积和分组卷积对解码器进行系统性的高效设计。
通过提出的三个创新模块——MSCAM(用于高效多尺度特征增强)、LGAG(用于高效大核上下文特征融合)和EUCB(用于高效上采样),EMCAD在12个基准数据集上实现了SOTA性能,同时将参数量和计算量(FLOPs)分别大幅降低了79.4%和80.3%。
这项工作不仅为资源受限的医疗场景(如移动端、实时诊断)提供了强大的工具,也为未来高效视觉模型的设计提供了有价值的思路。其开源的代码也促进了该领域的进一步研究和发展。
