CV 医学影像分类、分割、目标检测,之【肺结节目标检测】项目拆解
CV 医学影像分类、分割、目标检测,之【肺结节目标检测】项目拆解
- 项目流程梳理 - 数据源阶段
- 数据探索阶段,了解数据结构
- 第一部分:文件遍历收集
- 第二部分:XML标注文件收集
- 第三部分:DICOM文件读取
- 第四部分:UID提取与匹配
- 第五部分:XML与DICOM配对
- 第六部分:XML解析函数
- 第七部分:可视化
- 核心概念关联图
- 数据准备阶段,为模型训练做准备
- 新增部分1:分布式训练准备
- 新增部分2:文件复制操作
- 新增部分3:DICOM文件保存
- 新增部分4:张量转换处理
- 新增部分5:不同的图像转换方式
- 新增部分6:图像保存操作
- 代码问题分析
- 核心差异总结:从看数据 到 用数据
- 模型训练阶段:训练一个SSD目标检测模型来自动识别CT图像中的肺结节
- 一、整体架构理解
- 二、核心组件:Dataset类
- **自定义数据集类**
- **数据获取逻辑**
- **标签处理**
- 三、数据加载器配置
- 四、模型选择与初始化
- 五、训练循环深度解析
- **前向传播与损失计算**
- **评估指标:IoU**
- 六、训练策略分析
- **优化器配置**
- **训练细节**
- 七、模型保存策略
- 八、代码问题诊断
- **严重问题**
- **逻辑错误**
- **内存泄漏风险**
- 九、完整训练流程图
项目流程梳理 - 数据源阶段
医生的经验(看片子识别肺结节)↓标注数据(框出位置)↓数据预处理(数据源阶段工作)↓模型学习(训练模型阶段工作)↓
AI的能力(自动识别肺结节)
原始数据├── CT影像文件(DICOM格式)│ └── 包含:像素数据 + 病人信息 + 扫描参数│└── 标注文件(XML格式)└── 包含:肺结节位置坐标 + 类型标签↓数据预处理↓┌──────────────────────────┐│ 1. 文件收集与整理 ││ 2. 影像与标注配对 ││ 3. 数据验证与可视化 ││ 4. 格式转换与标准化 │└──────────────────────────┘↓训练数据集↓深度学习模型训练↓目标检测模型
问4:为什么有两种不同的文件(DICOM和XML)?
答4:DICOM是CT机器生成的原始影像,XML是医生后期添加的标注。
问5:这两种文件是怎么对应的?
答5:通过唯一标识符(UID)——每张CT图像都有独一无二的ID。
问6:为什么不把标注直接写在DICOM里?
答6:DICOM格式固定,不便修改;XML灵活,便于多人协作标注。
数据探索阶段,了解数据结构
第一部分:文件遍历收集
dcm_file=[]
for root,dirs,files in os.walk(r'E:\肺结节目标检测\manifest-1626051497651\Lung-PET-CT-Dx'):for file in files:file_path=os.path.join(root,file)if 'dcm' in file_path:dcm_file.append(file_path)
问1:这段代码的目的是什么?
答1:收集所有DICOM格式的医学影像文件路径。
问2:什么是DICOM文件?
答2:医学数字成像和通信标准文件,存储CT、MRI等医学影像数据。
问3:os.walk做什么?
答3:递归遍历目录树,返回(当前目录路径,子目录列表,文件列表)三元组。
问4:为什么要递归遍历?
答4:因为医学影像通常按病人/检查日期/序列分层存储在多级目录中。
问5:os.path.join的作用是什么?
答5:将路径组件智能地拼接成完整路径,自动处理斜杠问题。
第二部分:XML标注文件收集
xml_file=[]
for root,dirs,files in os.walk(r'E:\肺结节目标检测\Lung-PET-CT-Dx-Annotations-XML-Files-rev12222020'):for file in files:file_path=os.path.join(root,file) if 'xml' in file_path:xml_file.append(file_path)
问6:为什么需要XML文件?
答6:XML文件包含了医生标注的肺结节位置信息(边界框坐标)。
问7:什么是标注(Annotation)?
答7:人工标记的真实答案,用于训练深度学习模型识别病灶。
第三部分:DICOM文件读取
import pydicom
im=pydicom.read_file('E:/肺结节目标检测/manifest-1626051497651/Lung-PET-CT-Dx/Lung_Dx-A0001/04-04-2007-Chest-07990/2.000000-5mm-40805/1-01.dcm')
问8:pydicom是什么?
答8:专门读取DICOM医学影像格式的Python库。
问9:为什么不用普通图像库如PIL?
答9:DICOM包含元数据(病人信息、扫描参数等),需要专门解析。
第四部分:UID提取与匹配
im_name=[]
for dcm in dcm_file:im=pydicom.read_file(dcm)dcm_uid=im.SOPInstanceUIDim_name.append(dcm_uid)
问10:SOPInstanceUID是什么?
答10:Service-Object Pair Instance Unique Identifier,每个DICOM图像的唯一标识符。
问11:为什么要提取UID?
答11:用于将影像文件与对应的标注文件精确匹配。
第五部分:XML与DICOM配对
xml_dcm=[]
for xml in xml_file:xml_file_name=xml[77:-4] # 提取文件名(去掉路径和扩展名)if xml_file_name in im_name:xml_dcm.append(xml_file_name)
问12:为什么用[77:-4]切片?
答12:77是路径长度,-4去掉".xml"扩展名,得到纯UID。
问13:这种硬编码有什么问题?
答13:路径改变就会出错,应该用os.path.basename等方法。
第六部分:XML解析函数
def get_labelFromXml(xml_file): tree=ET.parse(an_file)root = tree.getroot()for object in root.findall('object'): cancer_type=object.find('name').text.upper()xmin=object.find('bndbox').find('xmin').text# ... 获取坐标bbox=[int(xmin),int(ymin),int(xmax),int(ymax)]
问14:ET.parse做什么?
答14:将XML文件解析成树形结构,便于提取标注信息。
问15:bndbox是什么?
答15:bounding box(边界框),用矩形框住病灶的最小矩形。
问16:为什么要检查xmin0或xminxmax?
答16:过滤无效标注(零值或退化成点/线的框)。
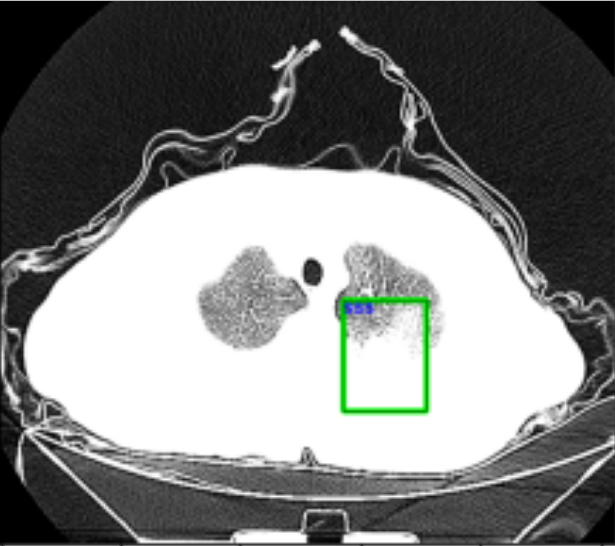
第七部分:可视化
im_array=np.array(im.pixel_array,dtype=np.float32)
image=cv2.cvtColor(im_array,cv2.COLOR_GRAY2RGB)
src_img=cv2.convertScaleAbs(image,alpha=(0.2))
cv2.rectangle(src_img,(x1,y1),(x2,y2),(0,255,0),2)
问17:pixel_array是什么?
答17:DICOM图像的像素数据矩阵,通常是灰度值。
问18:为什么COLOR_GRAY2RGB?
答18:CT图像是灰度的,但绘制彩色框需要RGB格式。
问19:convertScaleAbs的alpha=0.2做什么?
答19:调整亮度,CT原始值范围大,需要缩放到0-255显示。
问20:(0,255,0)代表什么?
答20:BGR格式的纯绿色,用于绘制检测框。
核心概念关联图
医学影像处理├── 数据格式│ ├── DICOM(影像+元数据)│ └── XML(标注信息)├── 数据组织│ ├── 文件遍历(os.walk)│ └── UID匹配(唯一标识)└── 目标检测├── 边界框(坐标定位)├── 标签(病灶类型)└── 可视化(验证标注)
这段代码本质是数据预处理管道:将散乱的医学影像和标注文件整理配对,为深度学习训练准备数据。每个步骤都是这个管道的必要环节。
数据准备阶段,为模型训练做准备
这份代码与之前的代码主体框架相似,但有几个关键差异。让我用连续提问法帮你理解这些不同之处:
新增部分1:分布式训练准备
import torchvision
import torch.distributed as dist
问1:为什么导入torch.distributed?
答1:准备进行分布式训练,可以在多GPU或多机器上并行训练。
问2:分布式训练的目的是什么?
答2:加速训练大规模医学影像模型,单GPU训练太慢。
新增部分2:文件复制操作
shutil.copy(file_path,'E:/肺结节目标检测/data/new_xml/R{}'.format(file))
问3:这行与之前注释掉的代码有何不同?
答3:这里直接执行了复制,将XML文件集中到新目录管理。
问4:为什么要复制而不是直接使用原路径?
答4:创建工作副本,避免修改原始标注文件。
新增部分3:DICOM文件保存
im.save_as('E:\肺结节目标检测\data\image\R{}.dcm'.format(dcm_uid))
问5:save_as与之前只读取有何区别?
答5:这里将DICOM文件重新保存,可能进行了格式标准化。
问6:为什么要重新保存DICOM文件?
答6:统一文件命名规则,用UID作为文件名便于配对。
新增部分4:张量转换处理
im_tensor=torch.from_numpy(im_array/255.)
im_tensor=torch.unsqueeze(im_tensor,0)
问7:为什么除以255?
答7:归一化到[0,1]范围,这是深度学习的标准预处理。
问8:unsqueeze(0)做什么?
答8:增加batch维度,从[H,W]变成[1,H,W],符合PyTorch输入格式。
问9:为什么需要batch维度?
答9:神经网络期望批量输入,即使只有一张图也要保持维度一致。
新增部分5:不同的图像转换方式
src_img=(im_array*255.).astype(np.uint8) # 新代码
# vs
src_img=cv2.convertScaleAbs(image,alpha=(0.2)) # 旧代码
问10:这两种方式有何区别?
答10:新代码直接乘255转uint8,旧代码用alpha=0.2降低亮度。
问11:为什么新代码不降低亮度?
答11:可能原始数据已经在合适范围,不需要额外调整。
新增部分6:图像保存操作
outputImg = Image.fromarray(outputImg*255.0)
outputImg = outputImg.convert('L')
outputImg.save('/Users/86187/Desktop/change_detection/YR-A-result.bmp')
问12:Image.fromarray做什么?
答12:将numpy数组转换为PIL Image对象。
问13:convert(‘L’)的作用?
答13:L表示Luminance(亮度),转换为8位灰度图。
问14:为什么保存为.bmp格式?
答14:BMP是无损格式,保留所有像素信息,适合医学影像。
代码问题分析
cv2.rectangle(src_img,(x1,y1),(x2,y2),random_color(),thickness=1)
问15:random_color()函数在哪里定义?
答15:代码中没有定义,这会导致运行错误。
问16:如何修复?
答16:需要定义:def random_color(): return (random.randint(0,255), random.randint(0,255), random.randint(0,255))
核心差异总结:从看数据 到 用数据
数据探索阶段流程:
读取 → 匹配 → 可视化数据准备阶段流程:
读取 → 复制整理 → 保存重组 → 张量转换 → 可视化 → 导出结果↓ ↓ ↓ ↓ ↓(备份) (标准化) (深度学习) (验证) (持久化)
模型训练阶段:训练一个SSD目标检测模型来自动识别CT图像中的肺结节
一、整体架构理解
问1:这段代码在做什么?
答1:训练一个SSD目标检测模型来自动识别CT图像中的肺结节。
问2:为什么之前是数据准备,现在是模型训练?
答2:机器学习流程:数据准备→模型构建→训练→评估,这是第2-3步。
二、核心组件:Dataset类
自定义数据集类
class CellDetection(Dataset):def __init__(self,img,label,transform=None):self.img = imgself.label=labelself.transform = transform
问3:为什么要继承Dataset类?
答3:PyTorch要求的标准接口,让数据能被DataLoader自动批处理。
问4:Dataset必须实现哪些方法?
答4:__getitem__(获取单个样本)和__len__(数据集大小)。
数据获取逻辑
def __getitem__(self, index):img_open=pydicom.read_file(img)img_array=img_open.pixel_arrayimg_array = np.array(img_array, dtype=np.float32)img_pic=Image.fromarray(img_array)img_tensor=self.transform(img_pic)
问5:为什么要经历这么多格式转换?
答5:
- DICOM → numpy:提取像素数据
- numpy → PIL.Image:为了使用transforms
- PIL → Tensor:模型需要的格式
问6:dtype=np.float32的作用?
答6:统一数据类型,避免整数运算丢失精度,GPU计算需要浮点数。
标签处理
class_to_id={'A':1}
cell=object.find('name').text.upper()
cell_id=class_to_id[cell]
问7:为什么要把’A’转成1?
答7:神经网络只认识数字,不认识字符串,这叫标签编码。
问8:为什么是{‘A’:1}而不是{‘A’:0}?
答8:0通常预留给背景类,1开始才是真实目标类别。
三、数据加载器配置
def detection_collate(x):return list(tuple(zip(*x)))dl_train=DataLoader(train_data,batch_size=4,shuffle=True,collate_fn=detection_collate)
问9:collate_fn是什么?
答9:自定义批处理函数,因为目标检测每张图的框数量不同,不能简单堆叠。
*问10:zip(x)在做什么魔法?
答10:转置操作——把[(img1,label1),(img2,label2)]变成([img1,img2],[label1,label2])。
问11:为什么batch_size=4这么小?
答11:医学图像分辨率高(512×512),显存有限,大batch会爆显存。
四、模型选择与初始化
model=torchvision.models.detection.ssd300_vgg16(pretrained=False, progress=True, num_classes=2)
问12:什么是SSD?
答12:Single Shot MultiBox Detector,单次前向传播就能检测多个目标的模型。
问13:为什么选SSD而不是YOLO或Faster R-CNN?
答13:SSD速度快,精度适中,适合医学图像这种目标相对规则的场景。
问14:num_classes=2代表什么?
答14:背景(0) + 肺结节(1) = 2个类别。
问15:pretrained=False的影响?
答15:从随机权重开始训练,没有利用ImageNet预训练,可能需要更多数据。
五、训练循环深度解析
前向传播与损失计算
model.train()
loss_dict=model(img,label)
losses = sum(loss for loss in loss_dict.values())
问16:model.train()做了什么?
答16:启用BatchNorm和Dropout层,让模型处于训练模式。
问17:loss_dict包含什么?
答17:SSD的多个损失——分类损失(classification_loss)和定位损失(bbox_regression_loss)。
问18:为什么要sum所有损失?
答18:多任务学习,同时优化分类和定位,需要联合优化。
评估指标:IoU
iou_tensor=torchvision.ops.box_iou(pic_boxes,label_boxes)
iou_total=np.mean(torch.max(iou_tensor,dim=1)[0].detach().numpy())
问19:IoU是什么?
答19:Intersection over Union,交并比,衡量预测框和真实框的重叠程度。
问20:IoU怎么计算?
答20:交集面积÷并集面积,完全重合=1,不重合=0。
问21:torch.max(iou_tensor,dim=1)[0]在做什么?
答21:每个预测框找最匹配的真实框,取最大IoU值。
六、训练策略分析
优化器配置
optimizer=torch.optim.Adam(model.parameters(),lr=0.0001)
# 后期:
optimizer=torch.optim.Adam(model.parameters(),lr=0.00001)
问22:为什么用Adam而不是SGD?
答22:Adam自适应学习率,对医学图像这种数据量少的任务更稳定。
问23:为什么学习率从0.0001降到0.00001?
答23:学习率衰减策略——前期快速下降,后期精细调整。
训练细节
try:pred=model(images)# IoU计算
except:continue
问24:为什么要try-except?
答24:某些批次可能没有目标,会导致IoU计算失败,需要跳过。
问25:这种处理方式好吗?
答25:不好,应该明确捕获特定异常,否则会掩盖真正的bug。
七、模型保存策略
torch.save(static_dict,'./data/checkpoint/{}_train_mIou_{}_test_mIou_{}.pth'.format(epoch,round(mIou, 3),round(test_mIou,3)))
问26:为什么文件名包含mIoU?
答26:方便直接从文件名看出模型性能,快速找到最佳模型。
问27:为什么每个epoch都保存?
答27:防止过拟合——后期模型可能变差,需要回溯到最佳点。
八、代码问题诊断
严重问题
# 问题1:训练集测试集划分
train_img,train_xml=new_img[:2000],new_xml[:2000]
test_img,test_xml=new_img[2000:],new_xml[2000:]
问28:这种划分有什么问题?
答28:没有打乱,可能导致数据分布不均(如前2000张都是某一个病人的)。
逻辑错误
# 问题2:测试时不应该计算梯度
for images, targets in tqdm(dl_test):model.train() # ← 错误!测试时应该model.eval()
问29:测试时用model.train()会怎样?
答29:BatchNorm使用批次统计而非全局统计,导致测试结果不稳定。
内存泄漏风险
loss_epoch.append(losses.cpu().numpy()) # losses仍保留计算图
问30:为什么需要.detach()?
答30:不detach会保留整个计算图在内存,导致内存泄漏。
九、完整训练流程图
数据准备↓
Dataset封装 → DataLoader批处理↓
模型初始化(SSD)↓
训练循环 ┌─────────────┐├─→ │ 前向传播 │├─→ │ 计算损失 │├─→ │ 反向传播 │├─→ │ 更新权重 │├─→ │ 评估IoU │└─→ │ 保存checkpoint│└─────────────┘↓
学习率调整 → 继续训练
