【AI论文】细胞锻造(CellForge):虚拟细胞模型的智能体化设计
摘要:虚拟细胞建模代表了人工智能与生物学交叉领域的一个新兴前沿,旨在定量预测细胞对各种干扰因素的反应等数量特征。然而,由于生物系统的复杂性、数据模式的异质性以及跨多个学科领域专业知识的需求,自主构建虚拟细胞的计算模型颇具挑战性。在此,我们介绍了细胞锻造(CellForge)系统,这是一个智能体化系统,利用多智能体框架,将所提供的生物数据集和研究目标直接转化为针对虚拟细胞的优化计算模型。更具体地说,CellForge仅需以原始单细胞多组学数据和任务描述作为输入,即可输出针对虚拟细胞模型训练和推理的优化模型架构和可执行代码。该框架集成了三个核心模块:用于数据集特征描述和相关文献检索的任务分析模块、由专业智能体协同开发优化建模策略的方法设计模块,以及用于代码自动化生成的实验执行模块。设计模块中的智能体分为不同视角的专家和一个中央协调者,它们必须协同交换解决方案,直至达成合理共识。我们利用六个涵盖基因敲除、药物治疗和细胞因子刺激等多种模式的不同数据集,展示了CellForge在单细胞干扰预测方面的能力。CellForge始终优于针对特定任务的最新方法。总体而言,CellForge展示了具有不同视角的大语言模型(LLM)智能体之间的迭代交互如何能提供比直接解决建模挑战更好的解决方案。我们的代码可在Github获取。Huggingface链接:Paper page,论文链接:2508.02276
研究背景和目的
研究背景:
随着人工智能(AI)技术的快速发展,其在科学发现中的应用日益广泛,尤其是在生物学领域。虚拟细胞建模作为AI与生物学交叉的一个新兴前沿,旨在通过定量预测细胞对各种干扰因素(如基因编辑、化学处理、环境变化等)的反应,推动计算生物学的发展。然而,构建虚拟细胞的计算模型面临诸多挑战,包括生物系统的复杂性、数据模式的异质性以及跨学科领域专业知识的需求。传统的建模方法往往依赖于大量手工设计和编码,且缺乏跨领域知识的整合,导致模型在面对新情境时的泛化能力有限。
研究目的:
本研究旨在开发一个名为CellForge的智能体化系统,通过多智能体框架自动将生物数据集和研究目标转化为优化的虚拟细胞计算模型。具体目标包括:
- 自动化建模流程:减少对人工干预的依赖,提高建模效率。
- 优化模型架构:通过智能体间的协作,设计出针对特定任务的优化模型架构。
- 生成可执行代码:自动生成用于训练和推理虚拟细胞模型的可执行代码。
- 跨领域知识整合:集成生物学、统计学和机器学习等多学科知识,提升模型的预测准确性。
- 验证模型性能:在多个单细胞干扰预测任务上验证CellForge的性能,并比较其与现有方法的优劣。
研究方法
数据集:
研究使用了六个公开可用的单细胞干扰数据集,涵盖基因敲除、药物治疗和细胞因子刺激等多种干扰类型和细胞类型。这些数据集提供了丰富的生物信息,用于训练和测试CellForge模型。
多智能体框架:
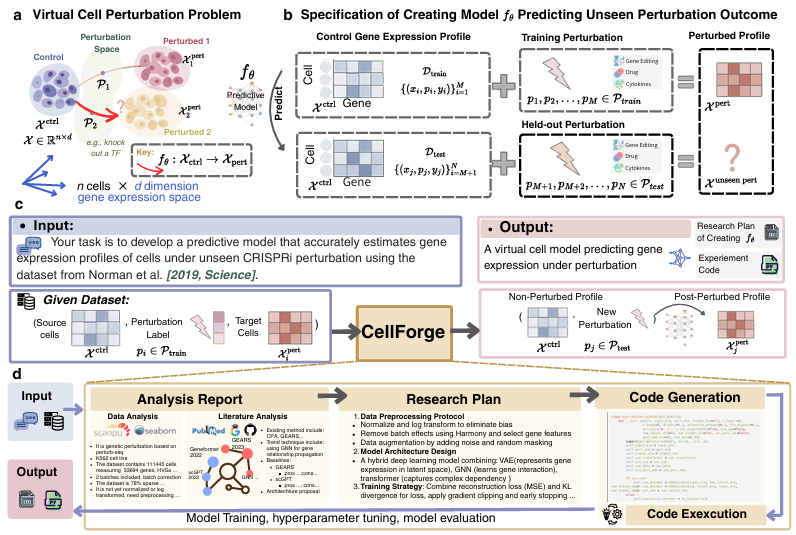
CellForge系统采用了一个多智能体框架,包含三个核心模块:
- 任务分析模块(Task Analysis Module):
- 数据解析:从单细胞数据集中提取关键元数据,包括干扰类型、基因特征和细胞群体。
- 文献检索:结合静态文献库和动态检索功能,通过PubMed和GitHub API检索相关文献,提取设计原则和候选架构。
- 分析报告:生成包含数据集特征、相关文献和任务特定约束的分析报告,为后续模块提供基础。
- 方法设计模块(Method Design Module):
- 专家智能体:包括数据预处理专家、模型设计专家和生物学解释专家等,每个专家从不同角度提出建模策略。
- 图结构辩论:专家智能体通过图结构辩论形式,迭代地提出、批评和融合候选架构,直至达成共识。
- 研究计划:输出包含数据预处理策略、模型架构设计和具体实现细节的研究计划。
- 实验执行模块(Experiment Execution Module):
- 代码生成:将研究计划转化为可执行的Python代码,包括数据预处理、模型训练和推理等步骤。
- 自动调试和再训练:在代码执行过程中自动调试错误并重新训练模型,直至满足验证目标。
模型架构:
CellForge设计的模型架构结合了变分自编码器(VAE)、图神经网络(GNN)和Transformer等多种组件,以捕捉基因之间的复杂关系和干扰效应。具体架构根据任务类型和数据特征动态调整,确保最佳预测性能。
研究结果
预测性能:
在六个单细胞干扰预测任务上,CellForge设计的模型显著优于现有方法。例如,在药物干扰任务中,CellForge将Pearson相关系数提高了20%;在scATAC-seq数据集上,实现了约16倍的Pearson相关系数增益。此外,CellForge在所有任务上均保持了较高的预测准确性,尤其是在处理未见过的干扰类型和细胞类型时。
模型解释性:
通过分析模型架构和注意力权重,CellForge提供了对基因干扰效应的生物学解释。例如,Transformer的自注意力层能够突出重要的基因相互作用,为理解基因调控网络提供了有价值的见解。
多智能体协作效果:
多智能体框架通过迭代交互和协作,显著提升了模型设计的质量和效率。与单一智能体或直接优化固定架构的方法相比,CellForge生成的模型在预测准确性和泛化能力上均有显著提升。
研究局限
计算和经济成本:
CellForge系统需要大量的计算资源和LLM调用,导致较高的经济成本。根据实验数据,每个实验的平均成本在0.38美元至18.90美元之间,具体取决于所选的LLM后端。此外,完整的模型训练和推理过程可能需要数小时至数十小时的GPU时间。
真实世界部署:
尽管CellForge在计算实验中表现优异,但其预测结果尚未在真实实验室环境中进行验证。未来的研究需要收集更多来自合作生物学家的实际查询,并测试CellForge预测在湿实验中的有效性。
失败案例分析:
在实验过程中,CellForge遇到了多种失败案例,包括计算执行错误、无效类型或操作以及模型配置错误等。这些失败案例反映了系统在处理复杂生物数据和建模任务时的局限性,需要进一步优化和改进。
未来研究方向
降低成本和提高效率:
未来的研究将探索更高效的计算架构和LLM调用策略,以降低CellForge的经济成本。例如,通过模型压缩和量化技术减少模型大小和计算量,或者利用更便宜的LLM API进行初步筛选和优化。
真实世界验证:
计划与计算生物学家合作,收集更多实际查询并测试CellForge在真实实验室环境中的预测效果。通过湿实验验证预测结果的准确性和生物学意义,进一步提升CellForge的实用性和可靠性。
扩展应用场景:
除了单细胞干扰预测外,CellForge还可以扩展到其他虚拟细胞建模挑战中,如预测细胞对环境变化的响应、模拟发育轨迹等。通过调整模型架构和训练策略,CellForge有望成为下一代虚拟细胞研究的基础工具。
增强模型解释性:
未来的研究将致力于提高模型的解释性,通过更精细的注意力机制和可视化技术揭示基因调控网络的复杂关系。这将有助于生物学家更好地理解模型预测结果,并推动计算生物学的发展。
持续优化和迭代:
CellForge系统将持续优化和迭代,以适应不断变化的生物数据和建模需求。通过引入新的算法和技术,不断提升模型的预测准确性和泛化能力,为科学发现提供更强大的支持。
