读取第三方的单细胞rds文件进行单细胞分析教程
读取第三方的单细胞rds文件进行分析
查看该rds文件中的meta.data,数据结构等信息内容
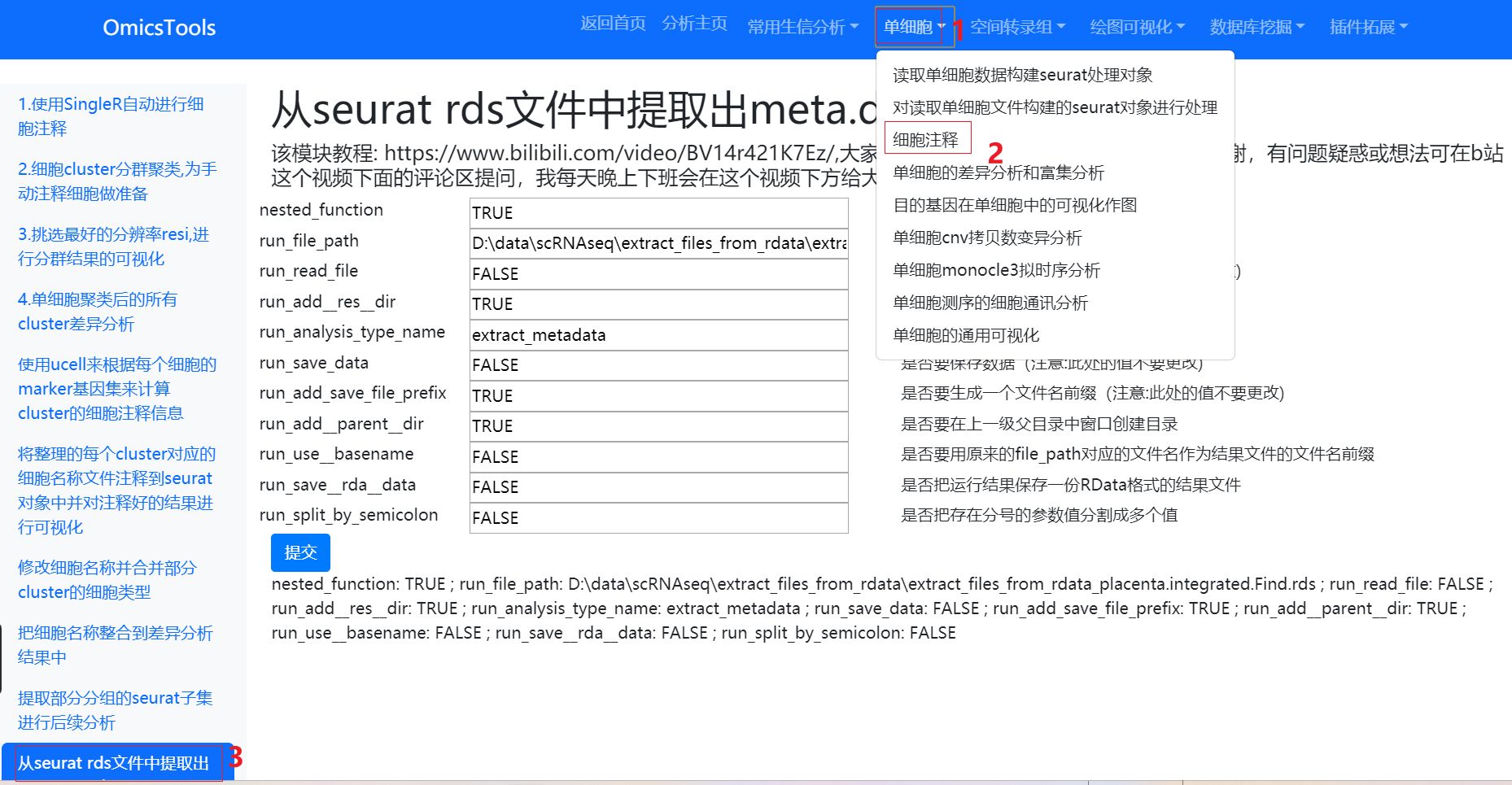
从seurat rds文件中提取出meta.data文件
软件界面
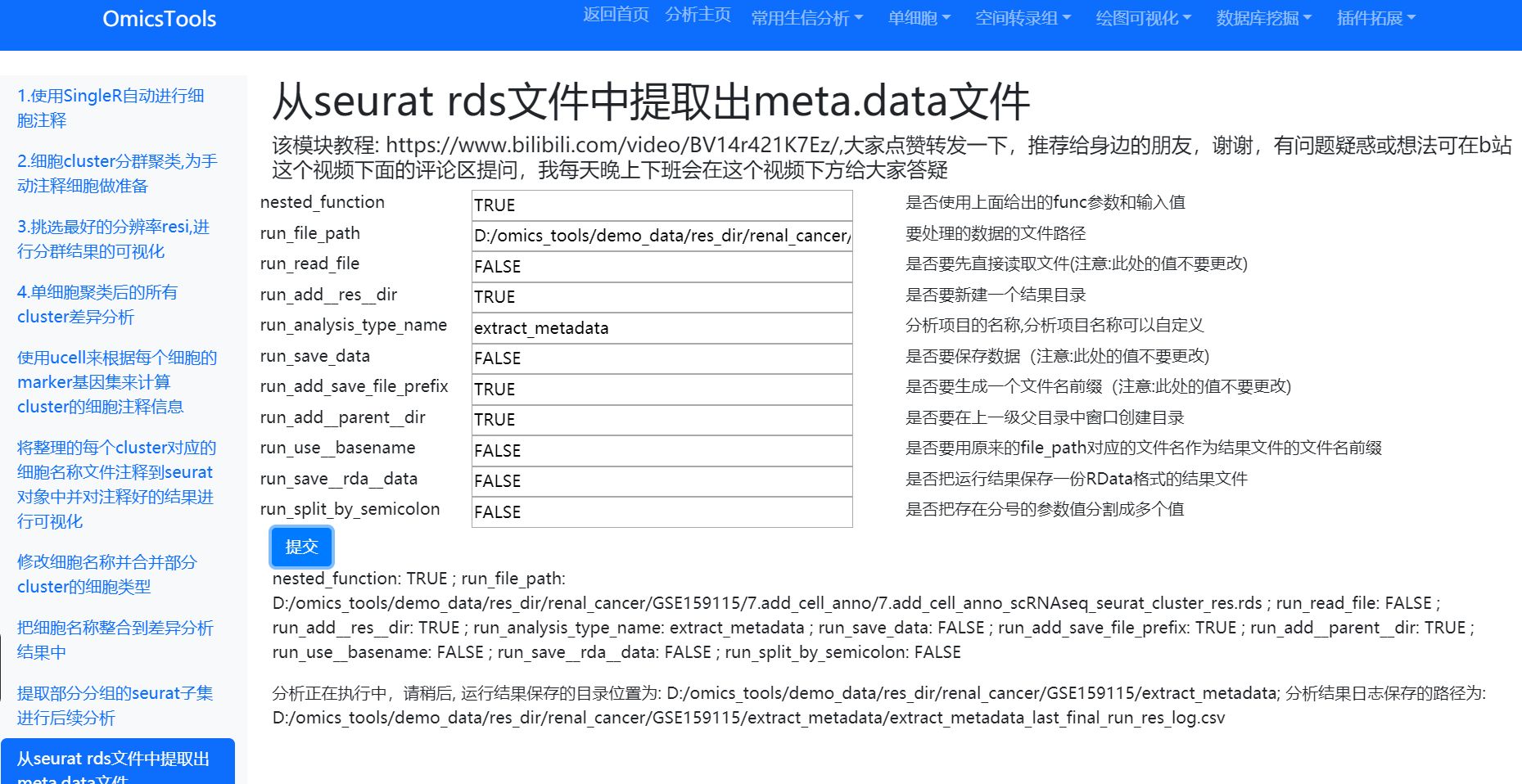
运行结果
结果文件列表
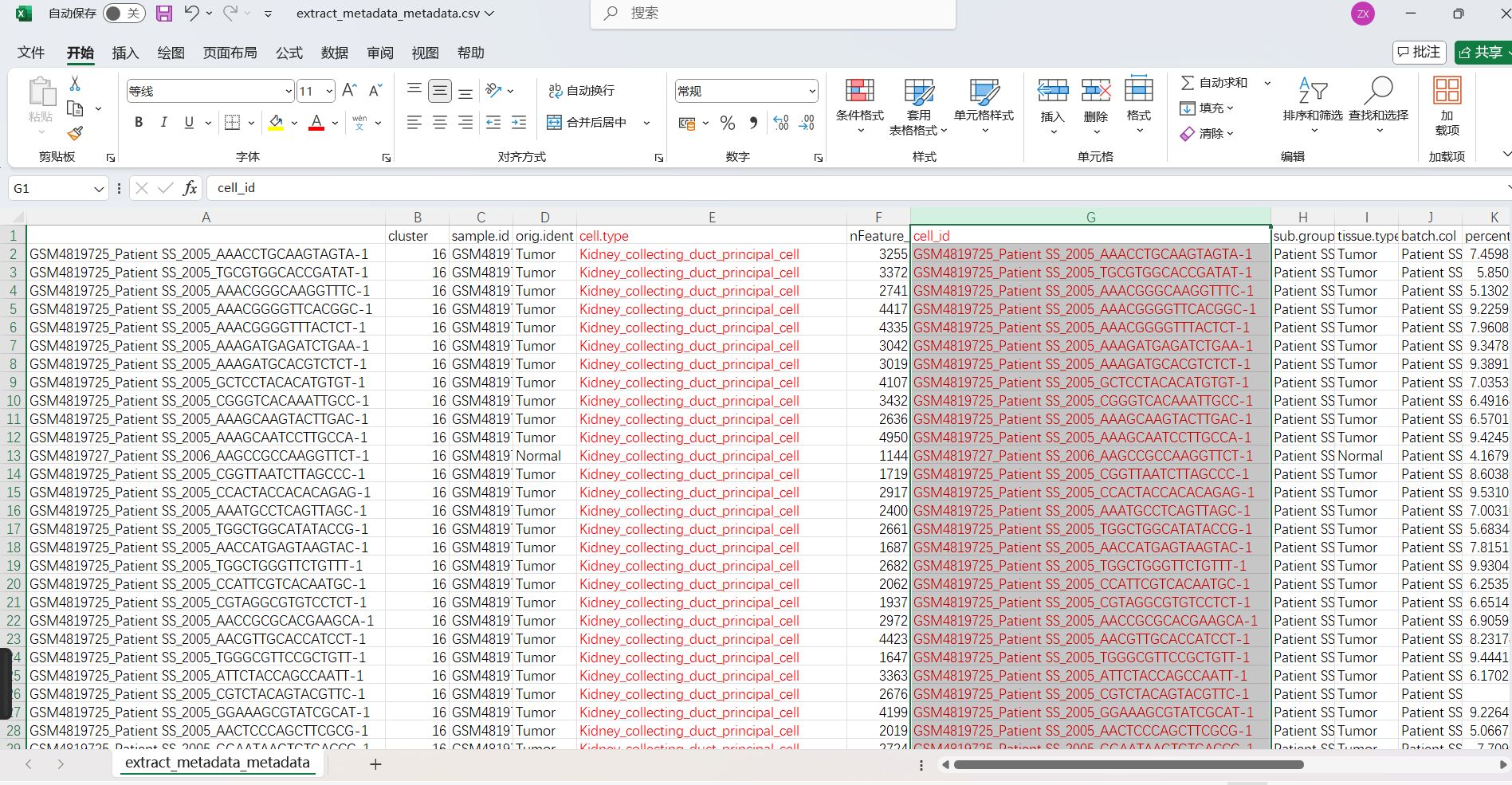
meta.data文件中的内容
基于该rds对象进行后续的单细胞下游分析
2.0 先完整运行一遍GSE159115这个演示数据集来理解整个分析流程
先完整运行一遍GSE159115这个演示数据集,然后把当前的数据集跟演示数据集每一步的运行结果进行比较,分析前面提取出的meta.data等内容才能来判断是否可以进行后续的下游分析或者应该从哪一步开始分析
2.1 分析前面提取出的meta.data等内容来判断是否可以进行后续的下游分析或者应该从哪一步开始分析
meta.data文件中的内容
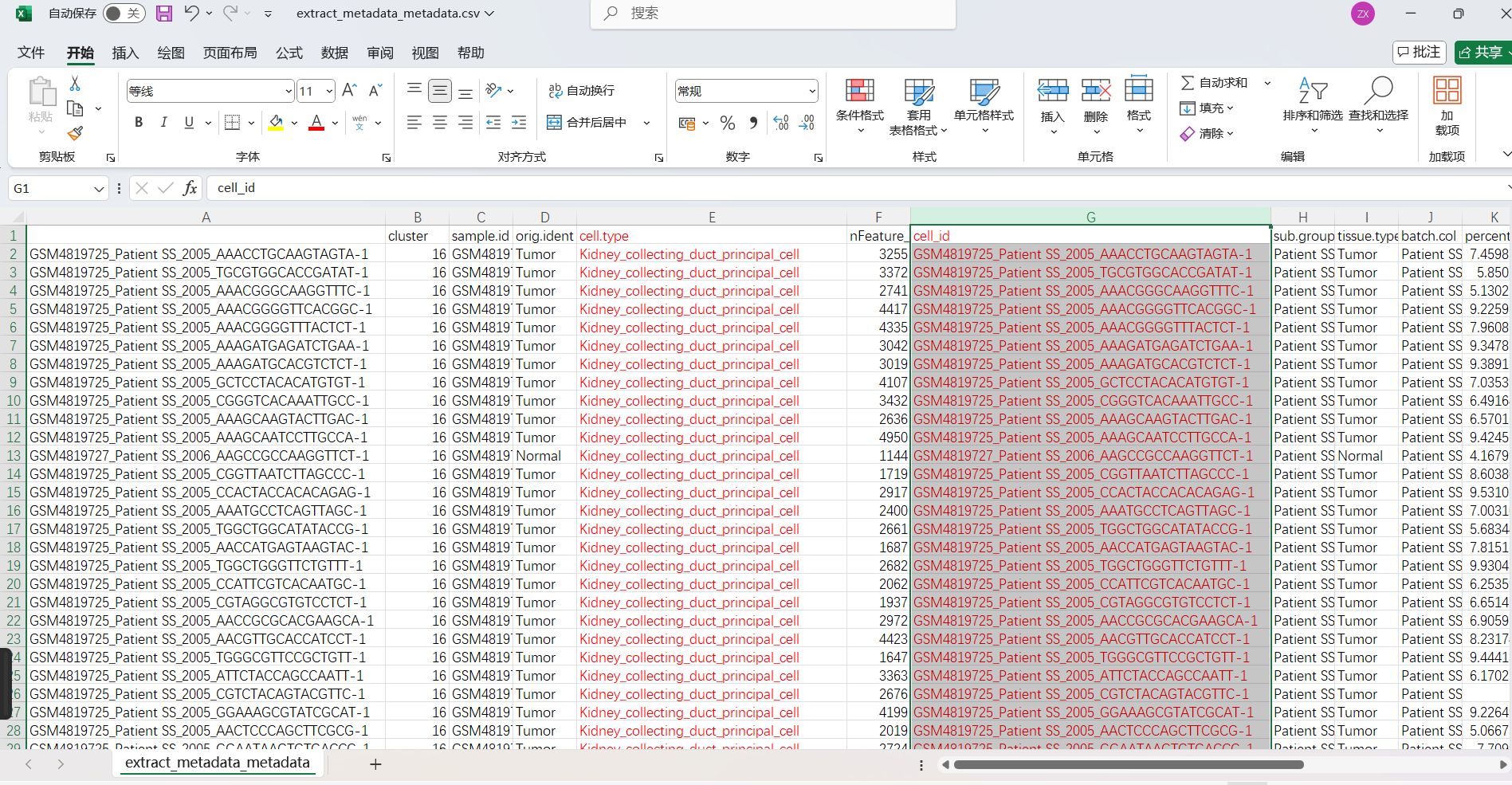
我们需要从meta.data文件中查看里面的每列信息,一般一定要有细胞标签id列,如cell_id列,可能会有细胞名称列,如cell.type列,如果有细胞名称列,说明已经注释好细胞名称,不用再重新注释,如果没有,则后续需要进行细胞注释。
查看extract_metadata_seurat_detail.txt中的seurat数据结构
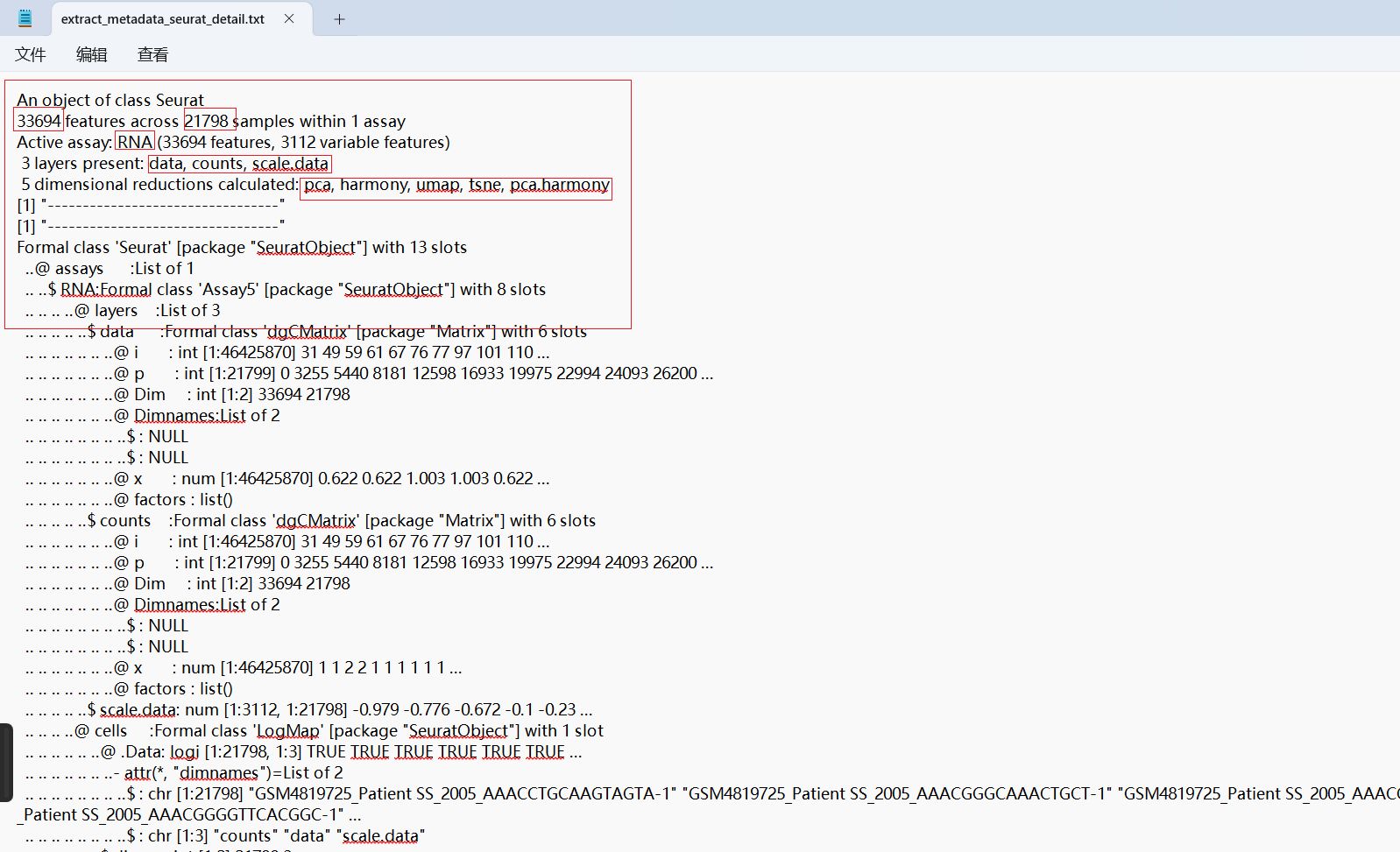
可以看到这个seurat单细胞对象重点信息如下:
- 33694个基因,21798个细胞
- 激活的assay是'RNA'
- 有3个数据层,分别是'counts','data'和'scale.data', 一般有'scale.data'层说明该单细胞数据之前做过了质控
- 有5种降维处理,分别是pca, harmony, umap, tsne, pca.harmony,说明该数据做过harmony多样本整合,pca降维,umap和tsne降维
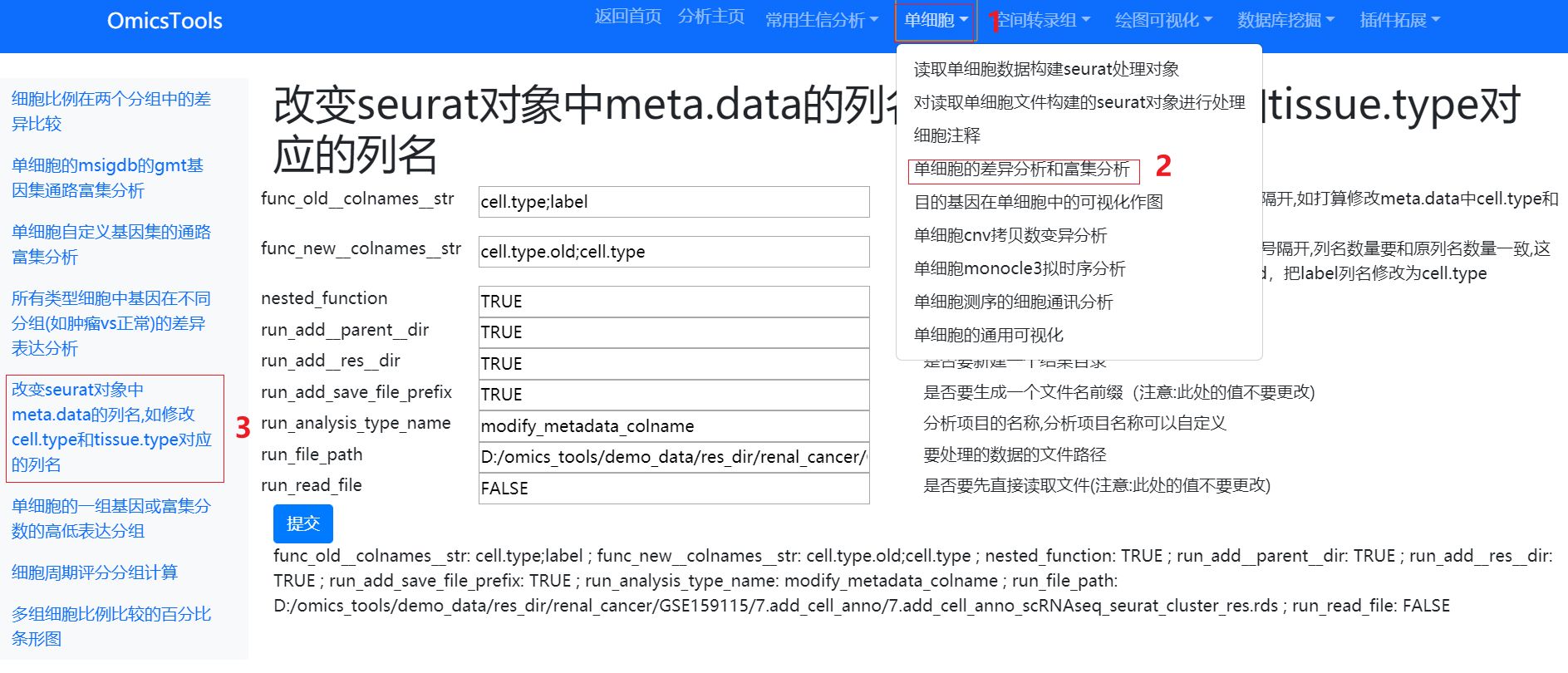
2.2 更改seurat对象中对应的meta.data中的细胞名称,样本名称,分组名称等重要列名保持这些重要列名跟演示数据一致
软件界面
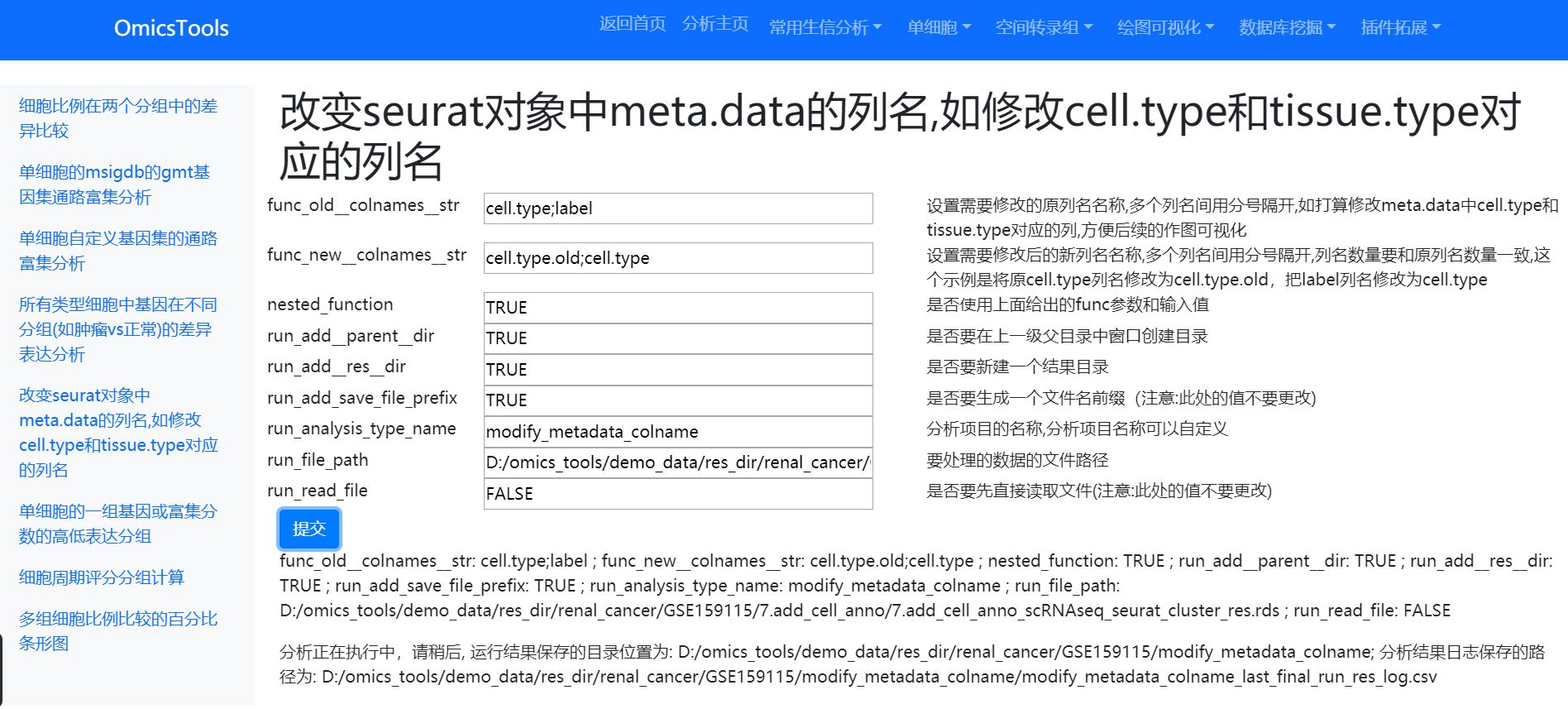
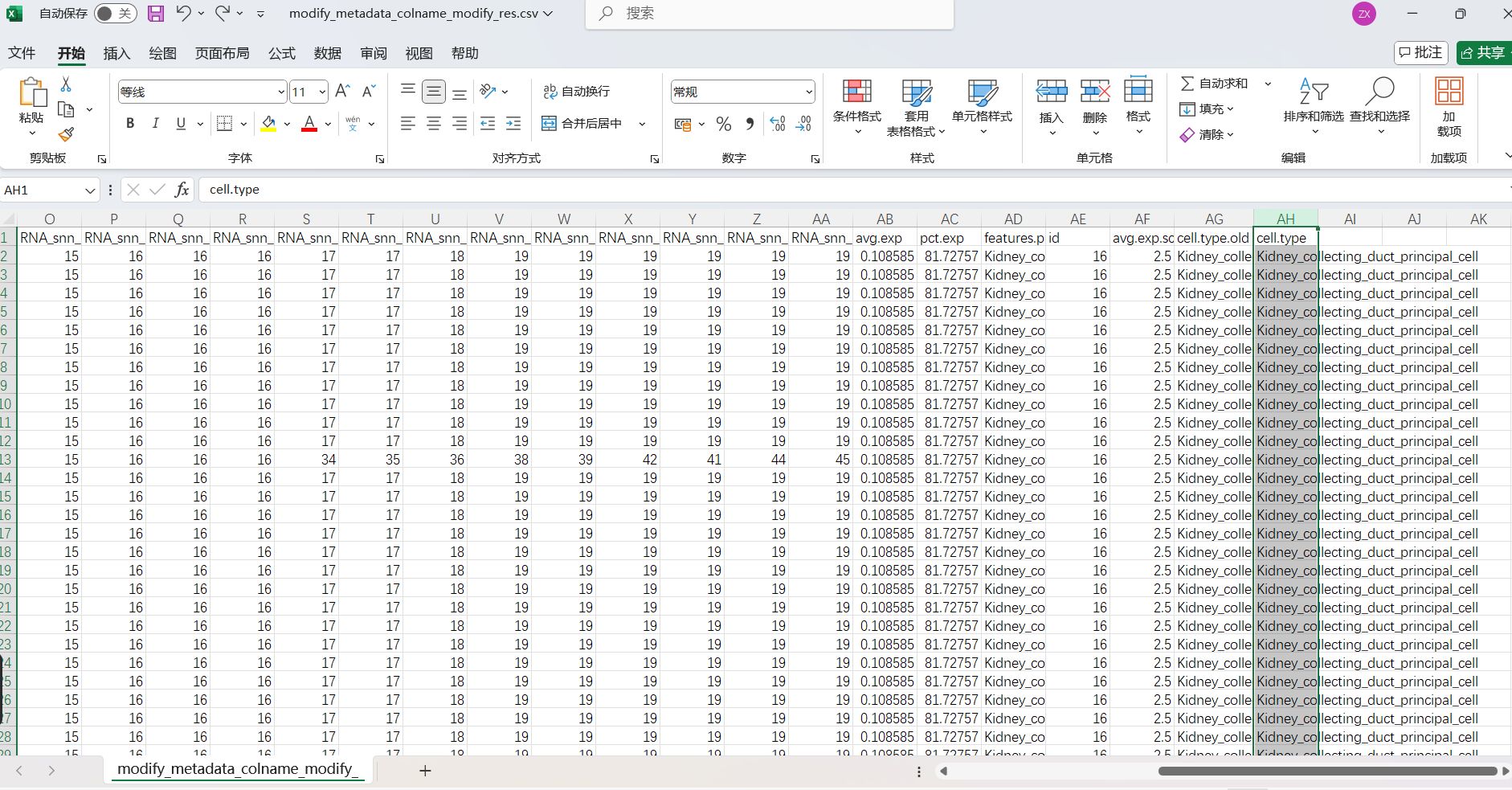
运行结果
重新构建Seurat分析对象从头开始分析(首推这种方法)
3.1将seurat rds文件提取出单细胞基因表达矩阵的表格文件
软件界面
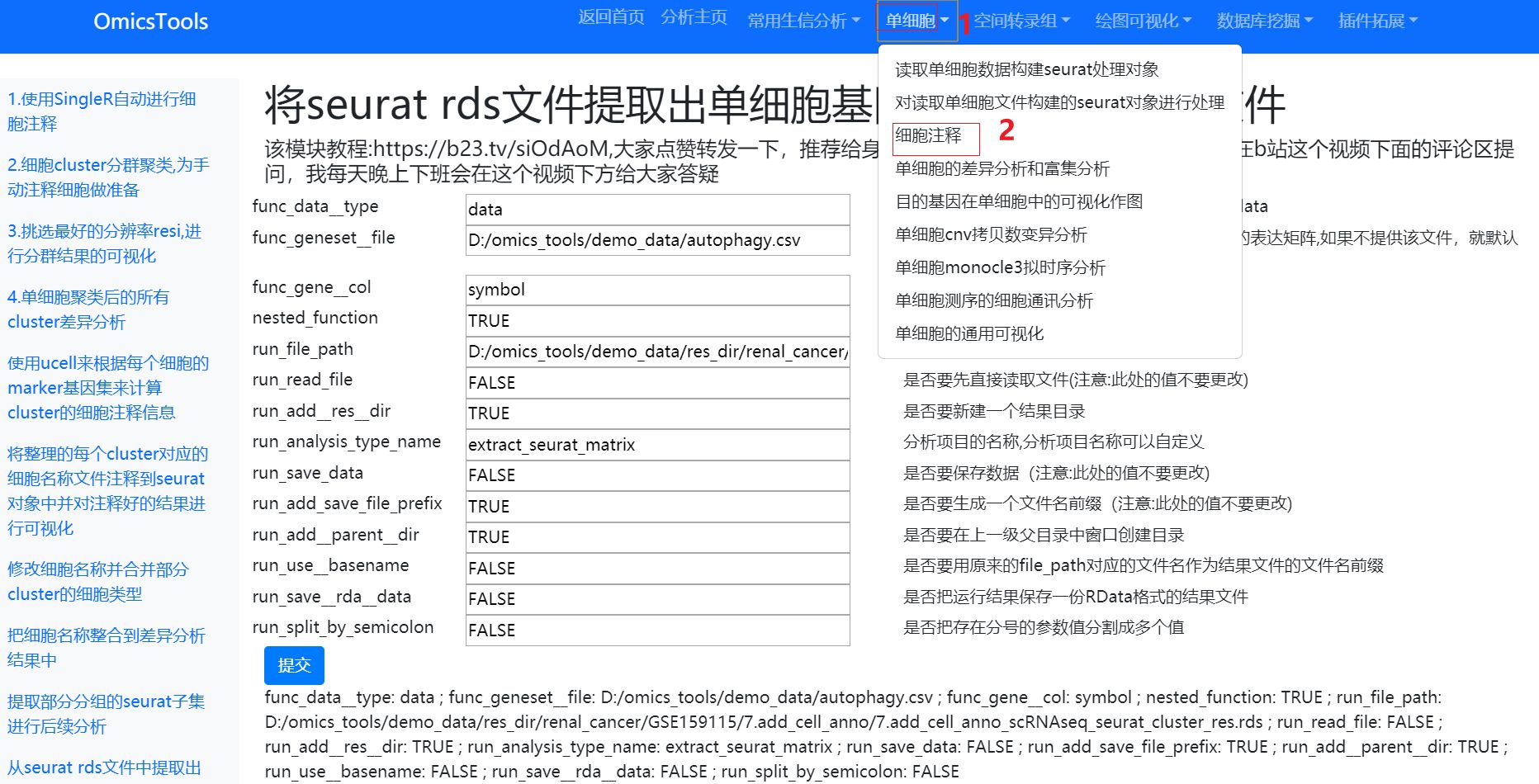
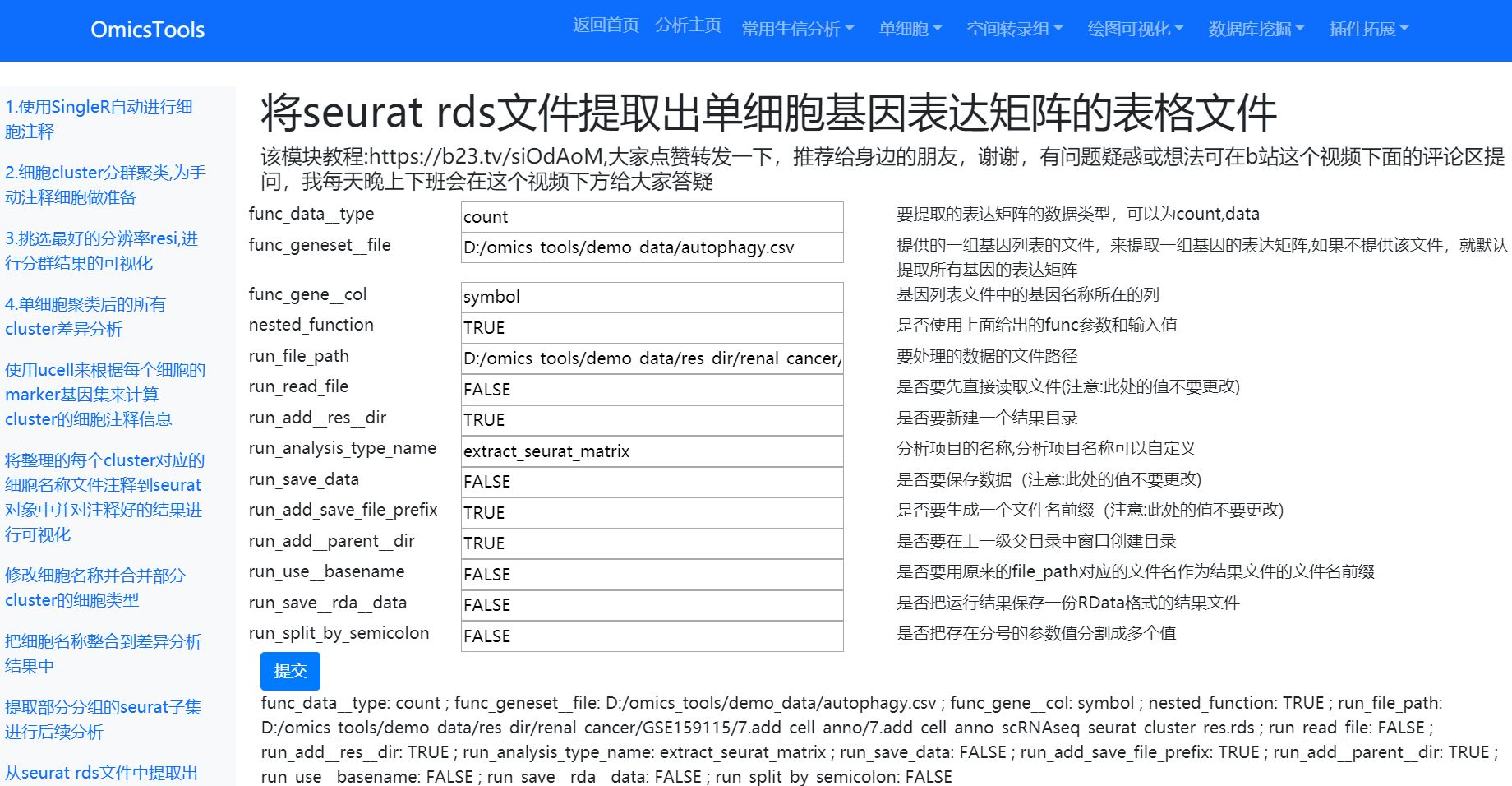
运行结果
结果文件列表
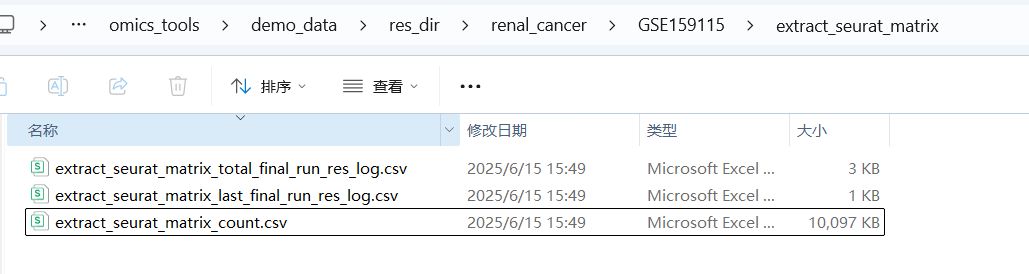
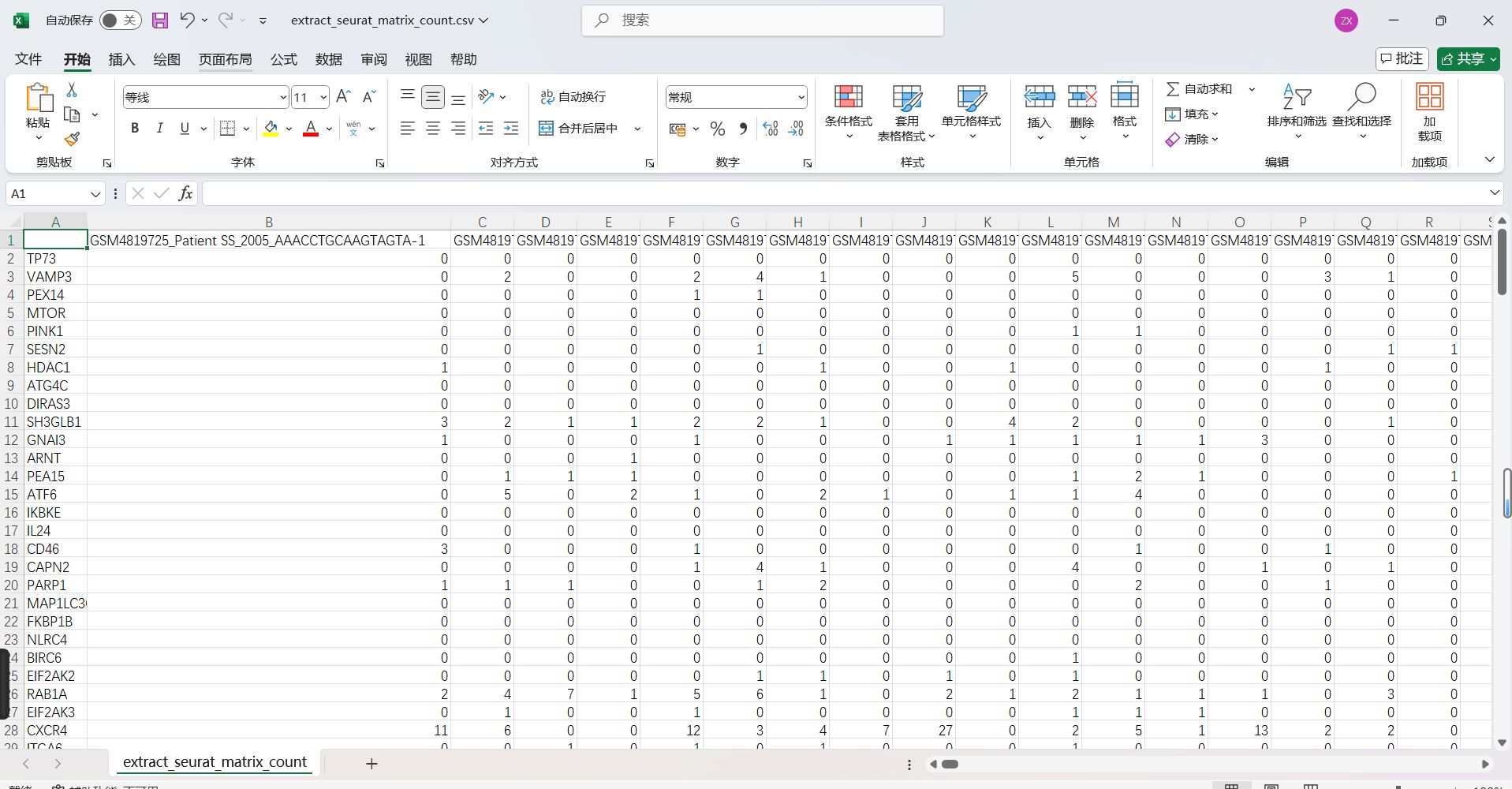
3.2读取单个单细胞表达矩阵文件构建seurat分析对象
软件界面
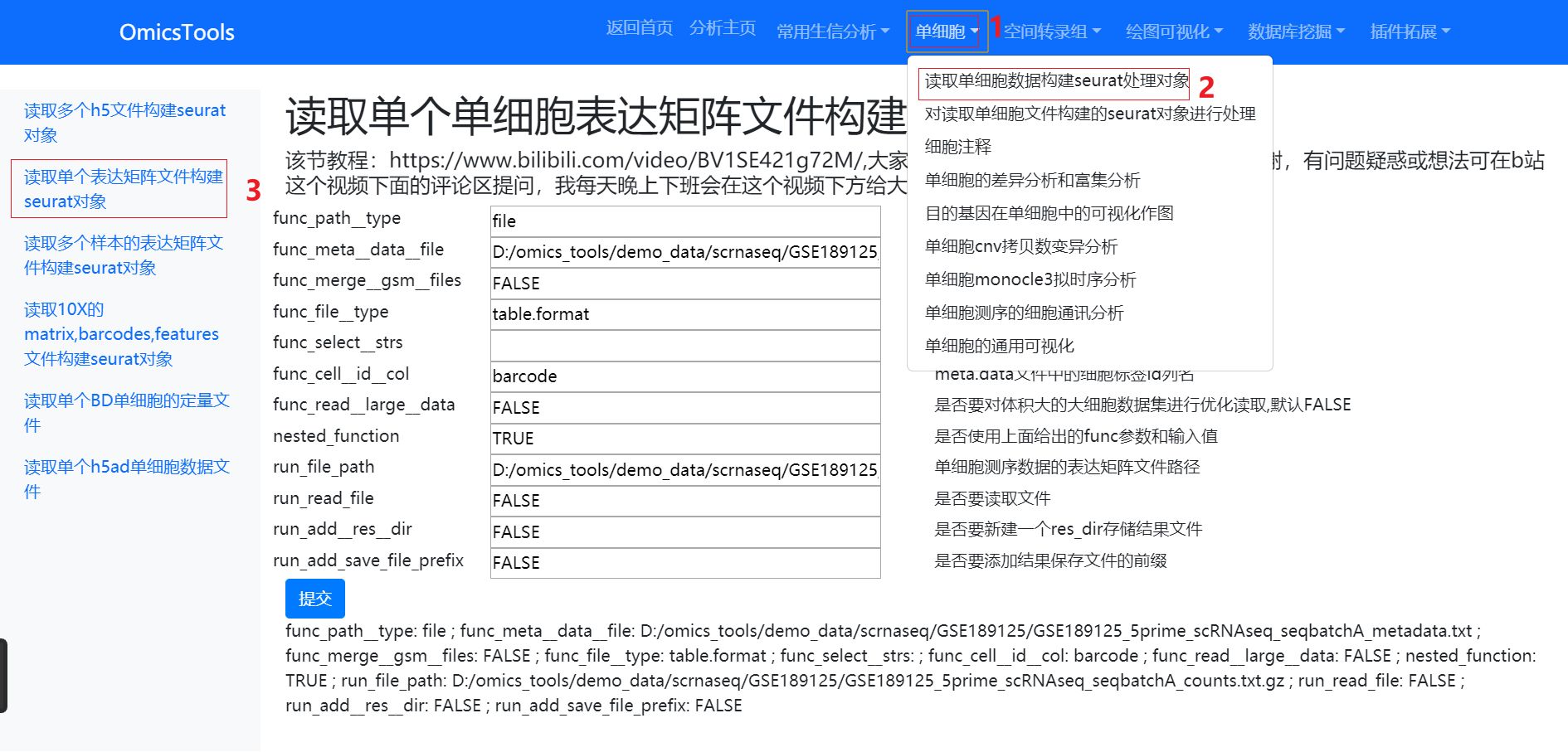
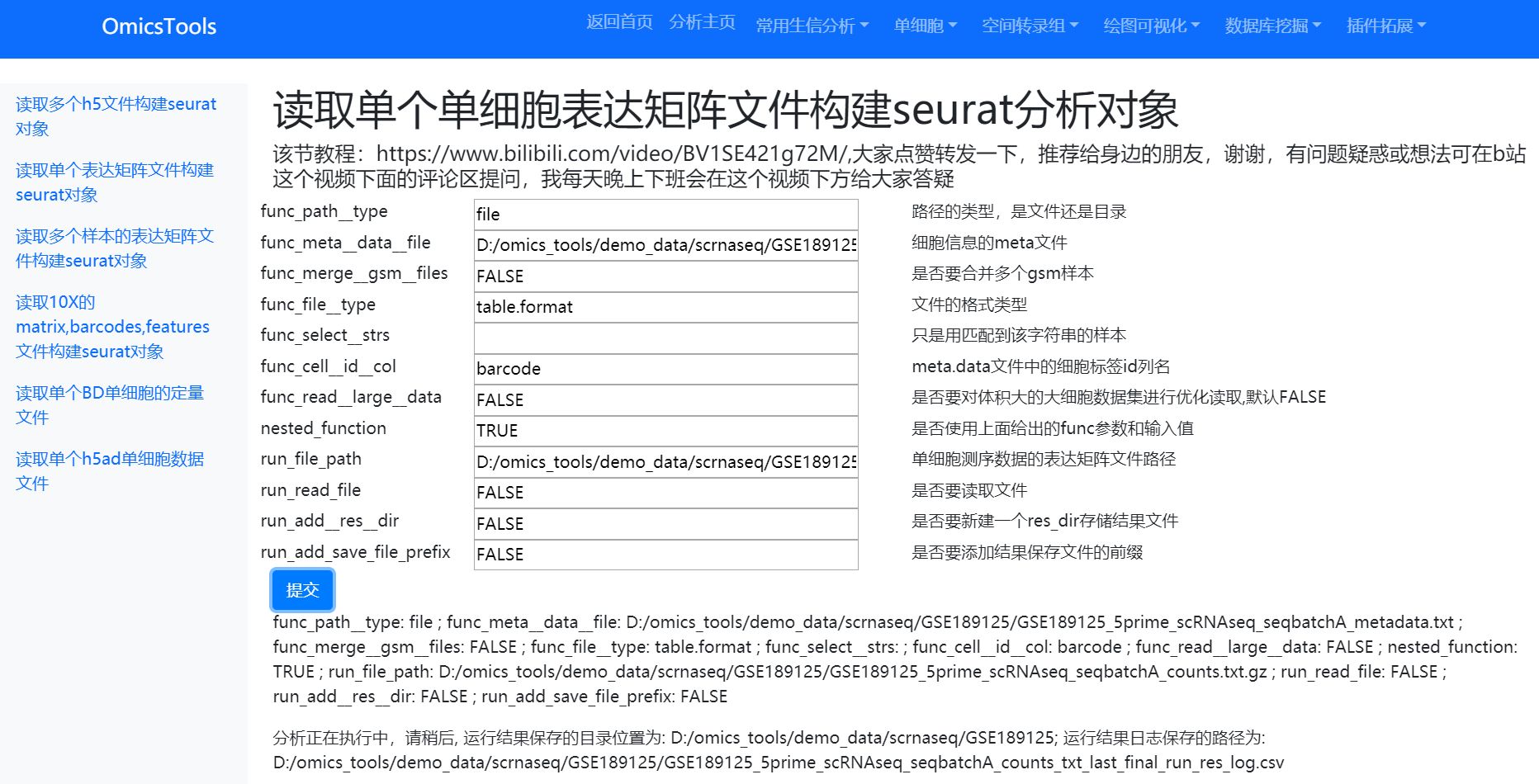
运行结果
OmicsTools软件和分析教程介绍
前言和简介
OmicsTools全能医学生物生信分析电脑软件简介
我开发了一款本地电脑无限使用的零代码生信数据分析作图神器一站式全流程电脑软件OmicsTools,旨在成为可以做各种医学生物生信领域科研数据分析作图的的全能科研软件,欢迎大家使用OmicsTools进行生物医学科研数据分析和作图,该软件件能让大家在不需要任何编程和代码编写的基础上,分析次数没有限制,可以无限使用,让您在自己电脑上快速进行大量的生信分析和加速大家的科研。
OmicsTools生信分析电脑软件可以做医学生物生信各个领域的科研数据分析和作图,并致力于成为医学生物生信领域的综合全能分析软件,一个软件帮助大家做医学生物生信领域的各种研究,快速出成果。

软件下载获取
我开发的本地电脑无限使用无限分析作图的生信零代码一键分析电脑软件神器OmicsTools 软件在github上的zihaoxingstudy1/OmicsTools(https://github.com/zihaoxingstudy1/OmicsTools)仓库中,也可以到我的生信交流q群群文件中下载,q群中的软件版本会更新一些,大家可以下载安装OmicsTools进行各种生信分析和可视化作图。
现在1群满员,会提示加2群,2群也可以下载到软件。
持续整理的各领域生信分析文档和答疑文档
所有人可编辑提问我对各种问题跟答疑答疑的腾讯文档
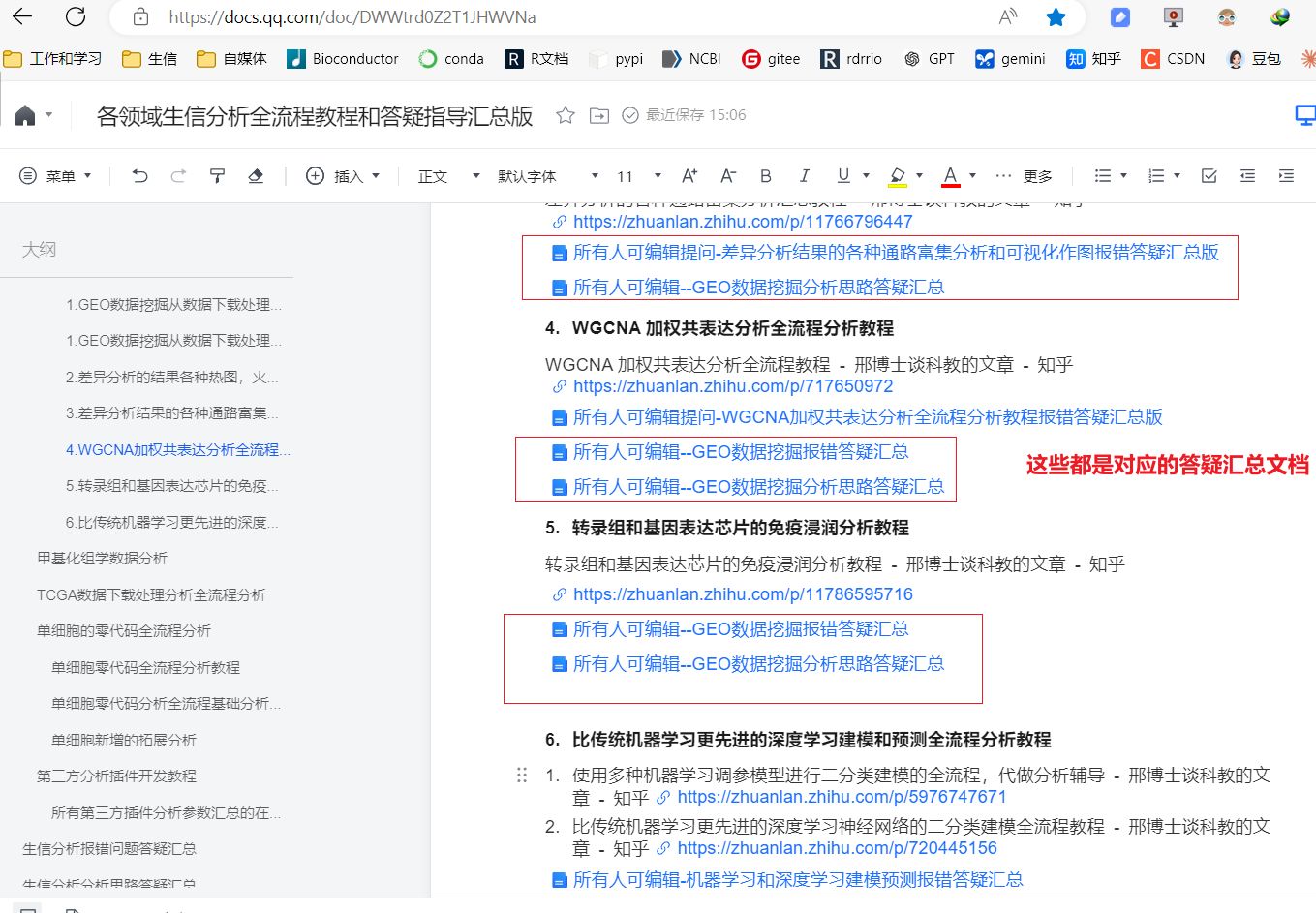
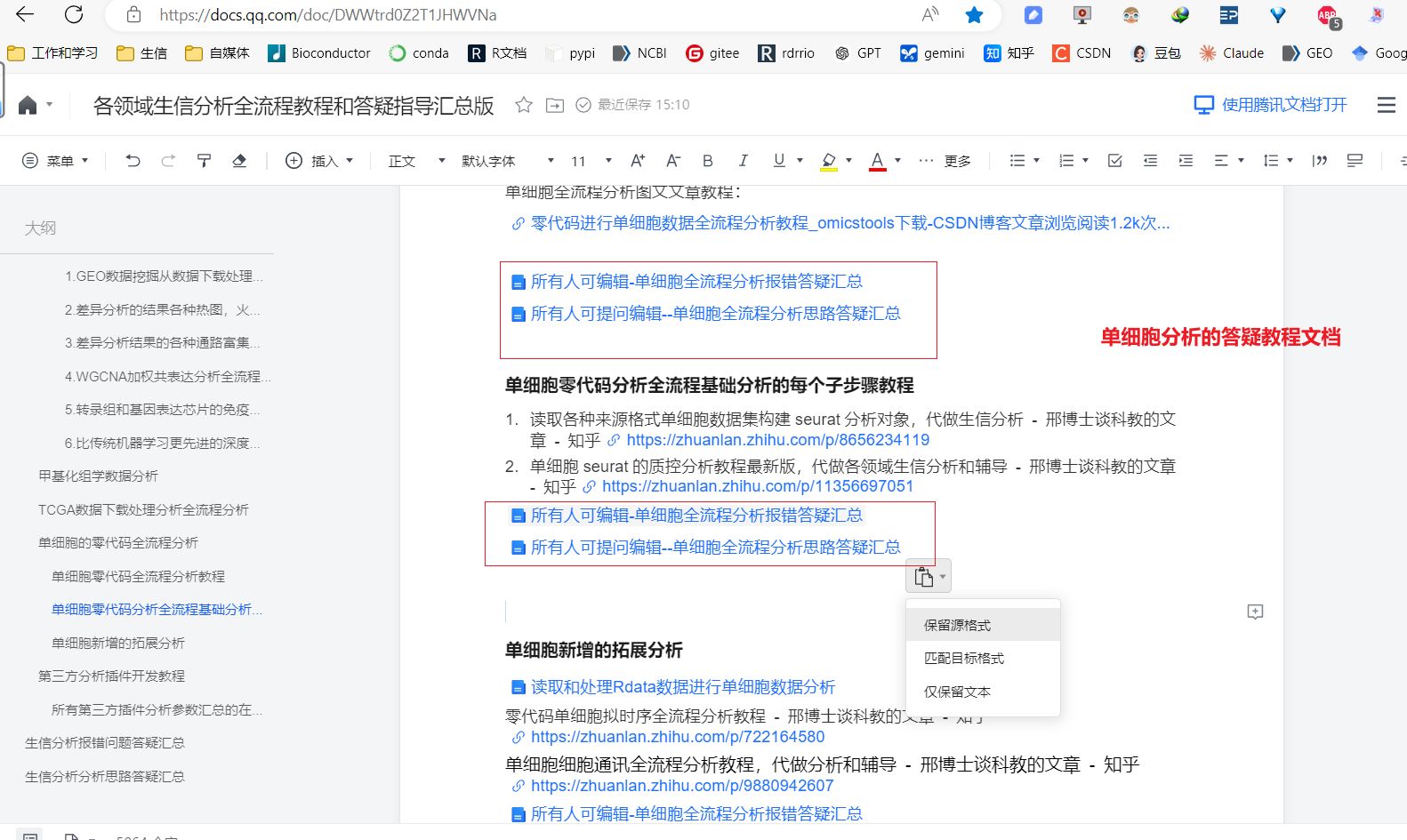
【腾讯文档】各领域生信分析全流程教程和答疑指导汇总版
https://docs.qq.com/doc/DWWtrd0Z2T1JHWVNa
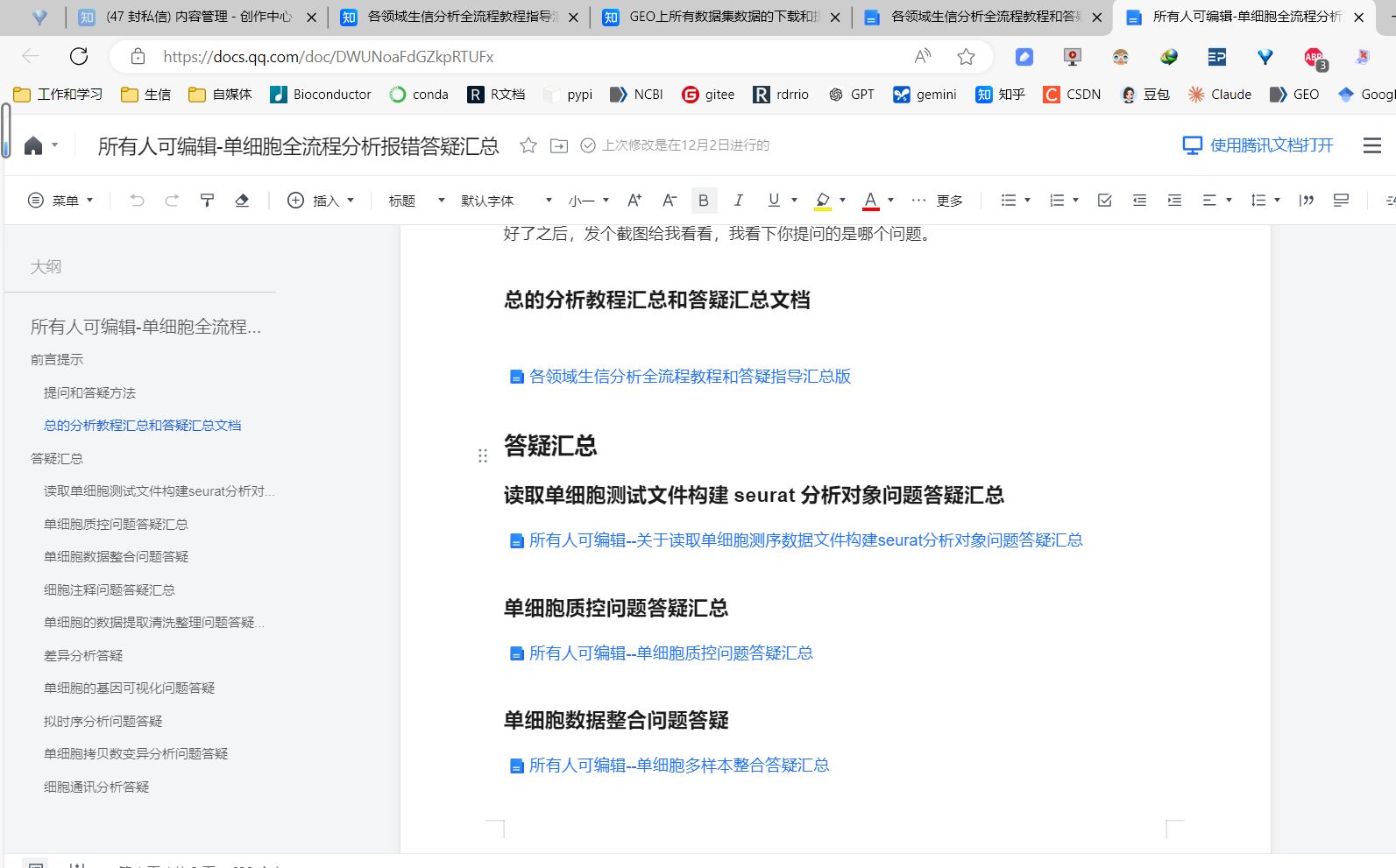
所有大家遇到的各种生信分析问题都在我的这篇腾讯文档对应的答疑文档中进行,腾讯文档的答疑文档支持所有人编辑和提问。
这篇总的腾讯文档是各领域生信分析答疑指导汇总文档的入口,以后所有的生信分析教程资料都在这个在线word文档中就能检索到,答疑汇总也能在这个word文档中检索到,都在这个在线word文档对应的提问答疑文档文件中提问,提问的问题在文档中用红色字体显示,我答疑在文档中用黑色字体显示,提问答个疑的文档和教程的文档所有人都可编辑。大家在腾讯文档里提问好了之后,现在答疑文档也是比较多的,发个截图和答疑文档的链接网址给我看看,这样我能快速定位到你提问的是哪个问题。
可以提供的科研服务清单