InSTAnT·细胞内空间转录组学分析包

InSTAnT 是一个专为 单分子分辨率空间转录组数据(如 MERFISH、seqFISH、Xenium) 设计的分析框架,目标是在亚细胞尺度上定量识别基因间的空间共定位模式(colocalization patterns),并推断其在细胞层面的功能关系。
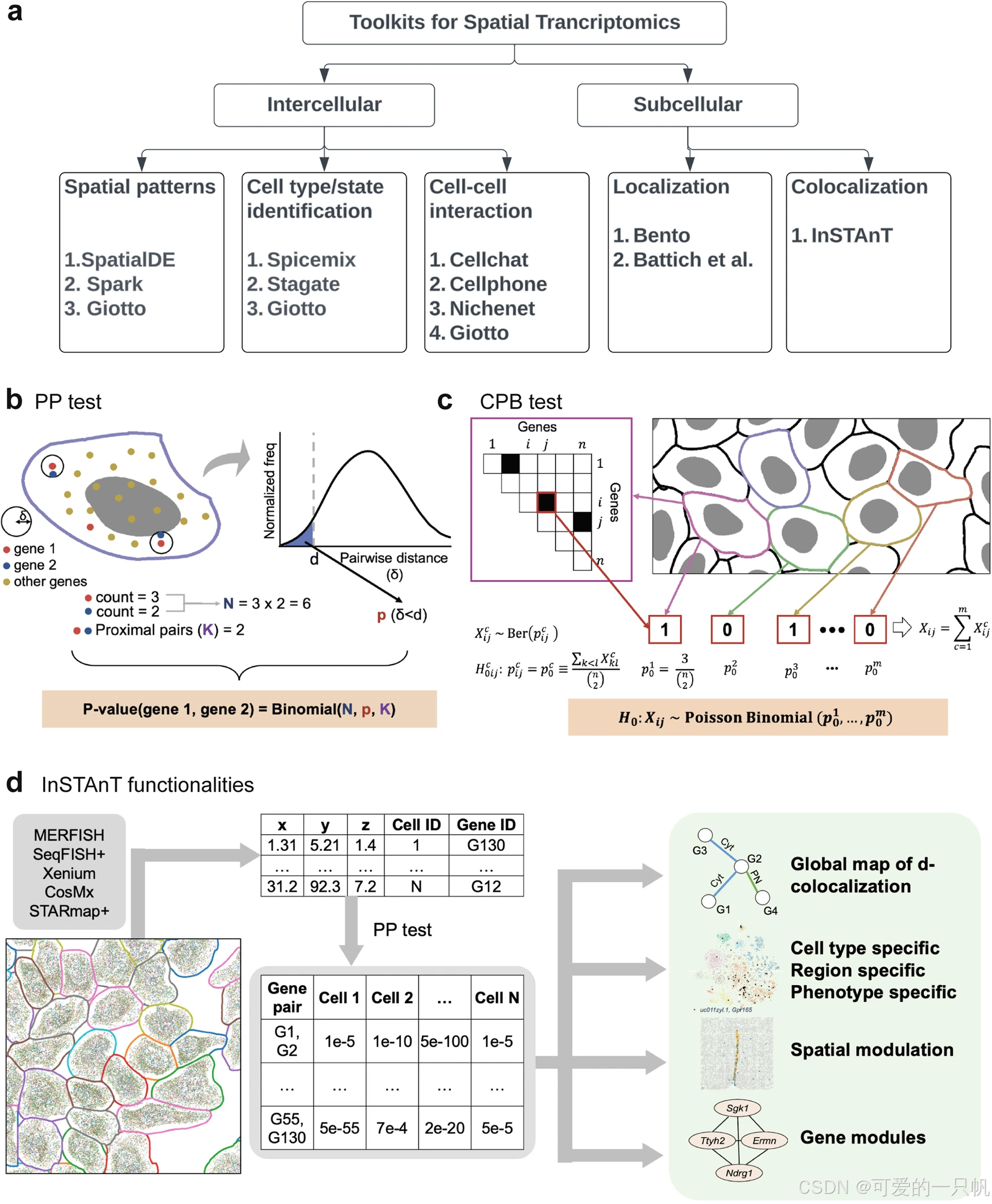
算法流程:
Step 1:输入数据
输入为单分子分辨率空间转录组数据表,包含:
-
每个 RNA 分子的空间坐标 (x, y, z);
-
所在细胞的标识信息。
Step 2:计算基因对的空间接近度
对每对基因 (A, B),在每个细胞内:
-
计算 A 分子与最近的 B 分子之间的距离;
-
若距离小于阈值 d(通常 1–4 µm),记为一次“接近事件”;
-
统计该事件在所有细胞中的出现频率。
Step 3:建立空间随机模型(Permutation Test)
为了判断“接近”是否超出随机期望:
-
在保持每个细胞 RNA 数量与形状不变的前提下,随机打乱基因标签;
-
重复多次生成空模型分布;
-
计算观测值的 p 值,严格控制假阳性。
Step 4:定义 Proximal Pair (PP)
若基因对 (A, B) 在多细胞中反复显著“靠近”(FDR < 0.05),
则定义为 Proximal Pair(PP) ——
代表该基因对的转录本在细胞内具有空间共定位倾向。
Step 5:定义 Conditional Proximal Bias (CPB)
在特定条件下(如细胞类型、亚细胞区域或表型)进一步检验:
-
A 与 B 的靠近是否显著增强或减弱;
-
若显著增强,则定义为 Conditional Proximal Bias (CPB)。
参考:Nature Communications volume 15, Article number: 7794 (2024)
