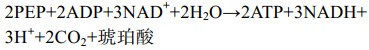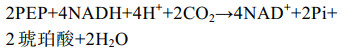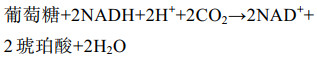调控大肠杆菌胞内ATP和NADH水平促进琥珀酸生产--文献精读172
Regulation of intracellular level of ATP and NADH in Escherichia coli to promote succinic acid production
调控大肠杆菌胞内ATP和NADH水平促进琥珀酸生产

摘要
琥珀酸作为一种重要的C4平台化合物,广泛应用于食品、化学、医药等领域。利用大肠杆菌(Escherichia coli)发酵生产琥珀酸受胞内辅因子不平衡的影响,存在产率低、生产强度低、副产物多等问题。为此,对不同氧气条件下琥珀酸产量和化学计量学分析发现,微厌氧条件下E. coli FMME-N-26高效积累琥珀酸需要借助三羧酸循环(tricarboxylic acid cycle, TCA)为还原性三羧酸途径(reductive tricarboxylic acid pathway, r-TCA)提供足够的ATP和NADH。通过减少ATP消耗、强化ATP合成、阻断NADH竞争途径和构建NADH回补路径等代谢工程策略,组合调控胞内ATP与NADH含量,获得工程菌株E. coli FW-17。通过发酵条件优化,菌株E. coli FW-17在5 L发酵罐能积累139.52 g/L琥珀酸,比出发菌株提高了17.81%,乙酸浓度为1.40 g/L,降低了67.59%。进一步在1 000 L发酵罐中进行放大实验,琥珀酸产量和乙酸浓度分别为140.2 g/L和1.38 g/L。
作为一种重要的C4平台化合物,琥珀酸广泛应用于食品、化学、医药等工业领域[1],被美国能源部列为12种最有潜力的大宗生物基化学品首位[2]。利用可再生资源为原料,通过微生物发酵生产琥珀酸,成为最受关注的方法。能发酵生产琥珀酸的菌种包括产琥珀酸放线杆菌(Actinobacillus succinogenes)[3]、产琥珀酸曼氏杆菌(Mannheimia succiniciproducens)[4]、酿酒酵母(Saccharomyces cerevisiae)[5]和大肠杆菌(Escherichia coli)等。其中大肠杆菌因为遗传背景清晰、代谢网络明确、易操作等优点[6],成为生产琥珀酸的重要菌种。大肠杆菌琥珀酸的合成路径包括还原性三羧酸途径(reductive tricarboxylic acid pathway, r-TCA)、乙醛酸途径和三羧酸途径(tricarboxylic acid pathway, TCA)。其中r-TCA因受到胞内辅因子含量的限制,琥珀酸对葡萄糖的产率为1.00 mol/mol葡萄糖[7];乙醛酸循环中琥珀酸产率为1.25 mol/mol葡萄糖[8],但在厌氧条件下,乙醛酸途径与r-TCA途径结合,可将理论产率提高到1.71 mol/mol葡萄糖[9];有氧条件TCA途径的理论产率为1.00 mol/mol葡萄糖[10]。
影响琥珀酸合成的关键因素是胞内辅因子平衡,目前主要策略包括:(1) 增加NADH供给。在大肠杆菌中过表达磷酸核糖转移酶(phosphoribosyltransferase, NAPRTase)和来自乳酸菌的丙酮酸羧化酶(pyruvate carboxylase, PYC),将NAD(H)含量比出发菌提高了9.8倍,琥珀酸产率达到了1.60 mol/mol葡萄糖[11];(2) 提高ATP供给。异源表达琥珀酸放线菌磷酸烯醇丙酮酸羧激酶,将磷酸烯醇式丙酮酸羧(phosphoenolpyruvate carboxylic, PEP)转化为草酰乙酸(oxaloacetic acid, OAA),促进ATP生成,使琥珀酸产量和菌体浓度分别提高了5.5倍和0.76倍[12];(3) 平衡NADH/NAD+比值和ATP水平。通过表达酿酒酵母可溶性富马酸还原酶、NADH还原酶,并引入ATP无效循环,使NADH/NAD+比值和ATP水平分别达到0.3和1.0 mmol/g DCW,琥珀酸产量比出发菌株提高了39% [13]。然而,上述策略研究工作仅通过单独提高辅因子(NADH、ATP)供应或调节辅因子平衡的问题,没有将两种策略进行组合考虑,导致琥珀酸发酵过程中仍存在乙酸、乳酸和丙酮酸等副产物,显著降低了琥珀酸产率,增加了琥珀酸的生产与下游提取成本[14-15]。在之前的研究中,在复合诱变所筛选的菌株中通过组合敲除pflB-focA、ldhA、pta、tdcD和tdcE后,为了增强ATP和NADH的供给,在基因组上组合表达产琥珀酸放线杆菌的PEP羧激酶(Actinobacillus succinogenes PEP carboxylkinase, AsPCK)和博伊丁假丝酵母(Candida boydin)的甲酸脱氢酶(Candida boydin formate dehydrogenase, CbFDH),最终构建了菌株E. coli FMME-N-26,琥珀酸产量提高了42.7%[16],但与ATP和NADH供给相关的基因涉及较少,供给能力不强,在发酵后期生产强度变弱。为了进一步提高ATP和NADH供给,促进琥珀酸生产,本研究进行琥珀酸生产化学计量学分析,在菌株E. coli FMME-N-26基础上,借助代谢工程策略组合调控ATP与NADH含量,构建了菌株E. coli FW-17,最终,将厌氧阶段残糖浓度进行优化并控制在1 g/L时,在1 000 L发酵罐中琥珀酸产量和生产强度分别达到140.2 g/L和1.95 g/(L·h),为工业化生产奠定了基础。
1 材料与方法
1.1 材料
1.1.1 菌株、质粒和引物
本研究使用的大肠杆菌E. coli JM109和E. coli FMME-N-26分别用于表达载体的构建和生产菌株。其中本研究所使用的基因工程菌、重组质粒和引物分别如表 1–3所示。
Table 1 Strains used in this study
| Strains | Description | Source |
| E. coli JM109 | General cloning host | TaKaRa Bio. |
| FMME-N | Wild type screened from rumen of camel (preservation number, CCTCCM2018568) | Lab stock |
| FMME-N-1 | After ARTP mutagenesis breeding from FMME-N | Lab stock |
| FMME-N-2 | After 60Co-γ irradiation mutagenesis breeding from FMME-N-1 | Lab stock |
| FMME-N-26 | FMME-N-2 ΔpflB-focAΔldhAΔptaΔtdcDΔtdcE | Lab stock |
| FW-1 | FMME-N-26-PJ01-Zmglf | This study |
| FW-2 | FMME-N-26-PJ01-pgm | This study |
| FW-3 | FMME-N-26-PJ01-Aspck | This study |
| FW-4 | FMME-N-26-ΔpoxB-Aspck | This study |
| FW-5 | FMME-N-26-ΔpoxB-Aspck-PJ01-H-pgm-H-Zmglf | This study |
| FW-6 | FMME-N-26-ΔpoxB-Aspck-PJ01-H-pgm-M-Zmglf | This study |
| FW-7 | FMME-N-26-ΔpoxB-Aspck-PJ01-H-pgm-L-Zmglf | This study |
| FW-8 | FMME-N-26-ΔpoxB-Aspck-PJ01-M-pgm-H-Zmglf | This study |
| FW-9 | FMME-N-26-ΔpoxB-Aspck-PJ01-M-pgm-M-Zmglf | This study |
| FW-10 | FMME-N-26-ΔpoxB-Aspck-PJ01-M-pgm-L-Zmglf | This study |
| FW-11 | FMME-N-26-ΔpoxB-Aspck-PJ01-L-pgm-H-Zmglf | This study |
| FW-12 | FMME-N-26-ΔpoxB-Aspck-PJ01-L-pgm-M-Zmglf | This study |
| FW-13 | FMME-N-26-ΔpoxB-Aspck-PJ01-L-pgm-L-Zmglf | This study |
| FW-14 | FMME-N-26-ΔpoxB-Aspck-PJ01-H-pgm-M-Zmglf-ΔadhE | This study |
| FW-15 | FMME-N-26-ΔpoxB-Aspck-PJ01-H-pgm-M -Zmglf-pCDR-gapA | This study |
| FW-16 | FMME-N-26-ΔpoxB-Aspck-PJ01-H-pgm-M -Zmglf-pCDR-pncB | This study |
| FW-17 | FMME-N-26-ΔpoxB-Aspck-PJ01-H-pgm-M-Zmglf-ΔadhE-pCDR-H-gapA-H-pncB | This study |
| FW-18 | FMME-N-26-ΔpoxB-Aspck-PJ01-H-pgm-M-Zmglf-ΔadhE-pCDR-H-gapA-M-pncB | This study |
| FW-19 | FMME-N-26-ΔpoxB-Aspck-PJ01-H-pgm-M-Zmglf-ΔadhE-pCDR- H-gapA-L-pncB | This study |
| FW-20 | FMME-N-26-ΔpoxB-Aspck-PJ01-H-pgm-M-Zmglf-ΔadhE-pCDR-M-gapA-H-pncB | This study |
| FW-21 | FMME-N-26-ΔpoxB-Aspck-PJ01-H-pgm-M-Zmglf-ΔadhE-pCDR-M-gapA-M-pncB | This study |
| FW-22 | FMME-N-26-ΔpoxB-Aspck-PJ01-H-pgm-M-Zmglf-ΔadhE-pCDR-M-gapA-M-pncB | This study |
| FW-23 | FMME-N-26-ΔpoxB-Aspck-PJ01-H-pgm-M-Zmglf-ΔadhE-pCDR-L-gapA-H-pncB | This study |
| FW-24 | FMME-N-26-ΔpoxB-Aspck-PJ01-H-pgm-M-Zmglf-ΔadhE-pCDR-L-gapA-M-pncB | This study |
| FW-25 | FMME-N-26-ΔpoxB-Aspck-PJ01-H-pgm-M-Zmglf-ΔadhE-pCDR-L-gapA-L-pncB | This study |
Table 2 Plasmids used in this study
| Plasmids | Characteristics | References |
| pCas | pMB1 ori, Kan, Pcas-cas9, ParaB-Red, Ptrc-sgRNA | Lab stock |
| pTargetF | pMB1 ori, SpeR, PJ23119 promoter | Lab stock |
| pJ01 | pMB1 ori, AmpR, PJ23119 promoter | [17] |
| pQE-Aspck | pMB1 ori, KanR, T5 promoter | [18] |
| pJ01-119-Glf-k-AroG-tktA | pMB1 ori, AmpR, PJ23119 promoter | Lab stock |
| pCDR-A | pBR322 ori, AmpR, Ptrc promoter | Lab stock |
| pJ01-Zmglf | pMB1 ori, AmpR, PJ23119, Zmglf | This study |
| pJ01-pgm | pMB1 ori, AmpR, PJ23119, pgm | This study |
| pJ01-Aspck | pMB1 ori, AmpR, PJ23119, Aspck | This study |
| pJ01-RBS0034(H)-pgm-RBS0034(H)-Zmglf | pMB1 ori, AmpR, PJ23119, pgm, Zmglf, RBS0034, RBS0034 | This study |
| pJ01-RBS0034(H)-pgm-RBS0031(M)-Zmglf | pMB1 ori, AmpR, PJ23119, pgm, Zmglf, RBS0034, RBS0031 | This study |
| pJ01-RBS0034(H)-pgm-RBS0011(L)-Zmglf | pMB1 ori, AmpR, PJ23119, pgm, Zmglf, RBS0034, RBS0011 | This study |
| pJ01-M-pgm-H-Zmglf | pMB1 ori, AmpR, PJ23119, pgm, Zmglf, RBS0031, RBS0034 | This study |
| pJ01-M-pgm-M-Zmglf | pMB1 ori, AmpR, PJ23119, pgm, Zmglf, RBS0031, RBS0031 | This study |
| pJ01-M-pgm-L-Zmglf | pMB1 ori, AmpR, PJ23119, pgm, Zmglf, RBS0031, RBS0011 | This study |
| pJ01-L-pgm-H-Zmglf | pMB1 ori, AmpR, PJ23119, pgm, Zmglf, RBS0011, RBS0034 | This study |
| pJ01-L-pgm-M-Zmglf | pMB1 ori, AmpR, PJ23119, pgm, Zmglf, RBS0011, RBS0031 | This study |
| pJ01-L-pgm-L-Zmglf | pMB1 ori, AmpR, PJ23119, pgm, Zmglf, RBS0011, RBS0011 | This study |
| pCDR-A-gapA | CloDF13 ori, SpeR, PJ23119, gapA | This study |
| pCDR-A-pncB | CloDF13 ori, SpeR, PJ23119, pncB | This study |
| pCDR-A-H-gapA-H-pncB | CloDF13 ori, AmpR, PJ23119, gapA, pncB, RBS0034, RBS0034 | This study |
| pCDR-A-H-gapA-M-pncB | CloDF13 ori, AmpR, PJ23119, gapA, pncB, RBS0034, RBS0031 | This study |
| pCDR-A-H-gapA-L-pncB | CloDF13 ori, AmpR, PJ23119, gapA, pncB, RBS0034, RBS0011 | This study |
| pCDR-A-M-gapA-H-pncB | CloDF13 ori, AmpR, PJ23119, gapA, pncB, RBS0031, RBS0034 | This study |
| pCDR-A-M-gapA-M-pncB | CloDF13 ori, AmpR, PJ23119, gapA, pncB, RBS0031, RBS0031 | This study |
| pCDR-A-M-gapA-L-pncB | CloDF13 ori, AmpR, PJ23119, gapA, pncB, RBS0031, RBS0011 | This study |
| pCDR-A-L-gapA-H-pncB | CloDF13 ori, AmpR, PJ23119, gapA, pncB, RBS0011, RBS0034 | This study |
| pCDR-A-L-gapA-M-pncB | CloDF13 ori, AmpR, PJ23119, gapA, pncB, RBS0011, RBS0031 | This study |
| pCDR-A-L-gapA-L-pncB | CloDF13 ori, AmpR, PJ23119, gapA, pncB, RBS0011, RBS0011 | This study |
| pTargetF-poxB | pMB1 ori, SpeR, PJ23119, poxB-N20 | This study |
Table 3 Primer used in this study
| Primer name | Primer sequence (5′→3′) | Size (bp) |
| poxB-500-up-F | GCGGCCCGGCTCCGTATATG | 20 |
| poxB-500-up-R | CCTCTTTACTAGTATTATACCTAGGACTGAGCTAGCTGTCAAGGTTCTCCATCTCCTGAATGTG | 64 |
| Aspck-Mid-F | CTCAGTCCTAGGTATAATACTAGTAAAGAGGAGAAAAAGCTTATGACTGACTTAAACAAACTCG | 64 |
| Aspck-Mid-R | GACGGGAAATGCCACCCTTTTTATGCTTTTGGACCGGCGC | 40 |
| poxB-500-down-F | GCGCCGGTCCAAAAGCATAAAAAGGGTGGCATTTCCCGTC | 40 |
| poxB-500-down-R | AATTCCCATGCTTCTTTCAGGTATT | 25 |
| poxB-JP-check-F | ACAATATTGCGTGATCTCTTTCAGT | 25 |
| poxB-JP-check-R | GTGGCAGAAAATAACGTTACCGAAG | 25 |
| poxB-N20-F | TGTCGTGTTACCAGGCGACGGTTTTAGAGCTAGAAATAGCAAGTT | 45 |
| poxB-N20-R | CGTCGCCTGGTAACACGACAACTAGTATTATACCTAGGACTGAGC | 45 |
| YZ-S-Ptarget | ACGCGGCCTTTTTACGGTTCCTGGCCTTTT | 30 |
| adhE-500-up-F | TGCAGGCCGTGCCAGTCATCCTTCA | 25 |
| adhE-500-up-R | AGTTTAACATTATCAGGAGAGCATTTCAGTAGCGCTGTCTGGCAACATAA | 50 |
| adhE-500-down-F | TTATGTTGCCAGACAGCGCTACTGAAATGCTCTCCTGATAATGTTAAACT | 50 |
| adhE-500-down-R | AAAATCAAAAAAGGTCTGAATCACG | 25 |
| adhE-JP-check-F | GGCGTTCTGCCGCTTAGTGG | 20 |
| adhE-JP-check-R | CTGCCGCTGTCTGATAACTG | 20 |
| adhE-N20-F | GGACGCCGCGAAGATCATGTGTTTTAGAGCTAGAAATAGCAAGTT | 45 |
| adhE-N20-R | ACATGATCTTCGCGGCGTCCACTAGTATTATACCTAGGACTGAGC | 45 |
| AspckA-dao-F | GTAAAGAGGAGAAAAAGCTTATGACTGACTTAAACAAACTCGTTAAAGAA | 50 |
| AspckA-dao-R | AATGATGATGATGATGATGGTCGACTTATGCTTTTGGACCGGCGC | 45 |
| pgm-dao-F | GTAAAGAGGAGAAAAAGCTTATGGCAATCCACAATCGTGC | 40 |
| pgm-dao-R | AATGATGATGATGATGATGGTCGACTTACGCGTTTTTCAGAACTTCGCTA | 50 |
| Zmglf-dao-F | GTAAAGAGGAGAAAAAGCTTATGAGTTCTGAAAGTAGTCAGGGTC | 45 |
| Zmglf-dao-R | AATGATGATGATGATGATGGTCGACCTACTTCTGGGAGCGCCACA | 45 |
| YZ-PJ01-F | AAAATAGGCGTATCACGAGGCA | 22 |
| YZ-PJ01-R | AGGCCCACCCGAAGGT | 16 |
| gapA-dao-F | GTAAAGAGGAGAAAAAGCTTATGACTATCAAAGTAGGTATCAACGGTTTT | 50 |
| gapA-dao-R | TGATGATGATGATGGTCGACTTATTTGGAGATGTGAGCGATCAGG | 45 |
| pncB-dao-F | GTAAAGAGGAGAAAAAGCTTATGACACAATTCGCTTCTCCTGTTC | 45 |
| pncB-dao-R | TGATGATGATGATGGTCGACTTAACTGGCTTTTTTAATATGCGGAAGGTC | 50 |
| YZ-pCDR-F | TCATGAGCCCGAAGTGGCGA | 20 |
| YZ-pCDR-R | CCAAGGTAGTCGGCAAATAA | 20 |
| pgm-dao-F-0031 | GTTCACACAGGAAACCAAGCTTATGGCAATCCACAATCGTGC | 42 |
| pgm-dao-F-0011 | AGTAGGGACAGGATAAGCTTATGGCAATCCACAATCGTGC | 40 |
| pgm-glf-fp-R | TTTTTCTCCTCTTTACTAGTTTACGCGTTTTTCAGAACTTCGCTA | 40 |
| pgm-glf-fp-F | CGCTCCCAGAAGTAGGTCGACCATCATCATCATCATCATT | 40 |
| pgm-glf-dao-F-0034 | AACTAGTAAAGAGGAGAAAAAGCTTATGAGTTCTGAAAGTAGTCAGGGTC | 50 |
| pgm-glf-dao-F-0031 | CTAGTTCACACAGGAAACCAAGCTTATGAGTTCTGAAAGTAGTCAGGGTC | 50 |
| pgm-glf-dao-F-0011 | AAACTAGTAGGGACAGGATAAGCTTATGAGTTCTGAAAGTAGTCAGGGTC | 50 |
| pgm-glf-dao-R-0034 | TGATGATGATGATGGTCGACCTACTTCTGGGAGCGCCACA | 40 |
| pgm-glf-dao-R-0031 | GGTTTCCTGTGTGAACTAGTTTACGCGTTTTTCAGAACTTCGCTA | 45 |
| pgm-glf-dao-R-0011 | CTTATCCTGTCCCTACTAGTTTACGCGTTTTTCAGAACTTCGCTA | 45 |
| pCDR-pncB-F-0034 | CCACGCGATCGCTGACGTCGGTACCAAAGAGGAGAAACACGCGATGACAC | 50 |
| pCDR-pncB-F-0031 | CCACGCGATCGCTGACGTCGGTACCTCACACAGGAAACCCACGCGATGAC | 50 |
| pCDR-pncB-F-0011 | CCACGCGATCGCTGACGTCGGTACCAGGGACAGGATCACGCGATGACACA | 50 |
| pCDR-pncB-xhoI-R | CGCAGCAGCGGTTTCTTTACCAGACTCGAGTTAACTGGCTTTTTTAATATGCGGAA | 56 |
| pCDR-gapA-F-0034 | CCAATTAAGCTTAAAGAGGAGAAAGAATTCATGACTATCAAAGTAGGTATCAACGGTT | 58 |
| pCDR-gapA-F-0031 | CCAATTAAGCTTTCACACAGGAAACCGAATTCATGACTATCAAAGTAGGT | 50 |
| pCDR-gapA-F-0011 | CCAATTAAGCTTAGGGACAGGATGAATTCATGACTATCAAAGTAGGTATC | 50 |
| gapA-bglii-R | GCGTGGCCGGCCGATATCCAATTGAGATCTTTATTTGGAGATGTGAGCGATCAGG | 55 |
1.1.2 主要仪器和试剂
PCR扩增仪、全自动凝胶成像系统、电转仪、核酸电泳仪,Bio-Rad公司产品;恒温培养箱,上海跃进医疗器械厂产品;紫外可见分光光度计,岛津公司产品;SBA生物传感器,山东科学院生物研究所产品;精密pH计,METTLER公司产品;UltiMate 3000液相色谱仪,赛默飞世尔科技有限公司产品;高速离心机,Eppendorf公司产品;5 L全自动搅拌式发酵罐,上海保兴生物设备工程有限公司;1 000 L迪必尔全自动搅拌式发酵罐,迪必尔生物工程(上海)有限公司。
限制性内切酶、Prime Star高保真酶、Taq DNA聚合酶、Hind Ⅲ、Sal Ⅰ、EcoR I、Bgl Ⅱ、Kpn I、Xho I、DNA marker等,购自TaKaRa (大连)有限公司;一步同源重组酶,购自南京巨匠生物科技有限公司;质粒提取试剂盒、胶回收试剂盒、产物纯化试剂盒;氨苄青霉素、硫酸卡那霉素、大观霉素、阿拉伯糖、异丙基-β-d-硫代半乳糖苷(isopropyl-β-d-thiogalactoside, IPTG),购自生工生物工程(上海)股份有限公司;细菌基因组提取试剂盒,购自天根生化科技(北京)有限公司;琥珀酸、乳酸、乙酸、甲酸,Sigma公司;PCR引物由亦欣生物科技(上海)有限公司合成;其他试剂购自国药集团化学试剂有限公司。
1.1.3 培养基
LB培养基:10 g/L NaCl,5 g/L酵母提取物,10 g/L蛋白胨。
发酵培养基:40 g/L葡萄糖,5 g/L玉米浆,3.3 g/L (NH4)2SO4,0.6 g/L KH2PO4,1.4 g/L K2HPO4·3H2O,0.4 g/L MgSO4·7H2O。
1.2 方法
1.2.1 基于CRISPR-Cas9系统的基因编辑
基因组整合基因采用CRISPR-Cas9系统[19]。
1.2.2 功能质粒的构建
单独或组合表达pgm基因、来自运动假单胞菌(Zymomonas mobilis)的Zmglf基因,来自A. succinogenes的Aspck基因的表达载体PJ01、单独或组合表达pncB、gapA基因的表达载体pCDR-A、含有Zmglf基因的表达载体pJ01-119-Glf-k-AroG-tktA、含有Aspck基因的表达载体pQE-Aspck为本实验室保存,且表达载体PJ01、pCDR-A为组成型表达载体。
通过设计的上下游引物利用聚合酶链式反应(polymerase chain reaction, PCR)从pJ01-119- Glf-k-AroG-tktA表达载体上扩增Zmglf基因片段,从pQE-Aspck表达载体上扩增Aspck基因片段,从大肠杆菌(Escherichia coli) MG1655基因组上扩增pgm基因片段,将其同源重组至载体PJ01的Hind Ⅲ和Sal Ⅰ位点之间,分别构建表达载体pJ01-Zmglf、pJ01-Aspck、pJ01-pgm。同样,通过上游引物将RBS0034、RBS0031、RBS0011引入pgm基因上游并将其同源重组至载体PJ01的Hind Ⅲ和Sal Ⅰ位点之间,随后对构建好的载体进行反向PCR得到相应的线性载体,通过上游引物将RBS0034、RBS0031、RBS0011引入Zmglf基因上游,并分别同源重组于3种线性载体上构建出不同RBS强度的pgm、Zmglf基因组合表达载体。
通过设计的上下游引物利用PCR从大肠杆菌MG1655基因组上扩增pncB、gapA基因片段,将其同源重组至载体pCDR-A的Kpn I和Xho I位点之间,构建表达载体pCDR-A-gapA、pCDR-A-pncB。同样,通过上游引物将RBS0034、RBS0031、RBS0011引入pncB基因上游并将其同源重组至载体pCDR-A的Kpn I和Xho I位点之间,随后通过上游引物将RBS0034、RBS0031、RBS0011引入gapA基因上游,并分别同源重组于3种载体的EcoR I、Bgl Ⅱ之间构建出不同RBS强度的gapA、pncB基因组合表达载体。
1.2.3 培养条件
平板培养:取保存于–80 ℃装有菌液的甘油管,三区划线于LB固体培养基中,在培养箱中37 ℃倒置培养16 h,得到大小均匀的单菌落。
种子培养方法:一级种子培养,挑取平板上大小均匀的单菌落,接种于装有25 mL液体LB培养基的100 mL锥形瓶中,37 ℃、220 r/min培养9 h;二级种子培养,将一级种子液转接100 µL至装有50 mL液体LB培养基的250 mL锥形瓶中,37 ℃、220 r/min培养8 h。
两阶段摇瓶发酵:按照1% (体积分数)的接种量将二级种子培养液接种于装有80 mL发酵培养基的500 mL锥形瓶中,发酵条件为38 ℃、220 r/min,培养12 h,迅速倒入100 mL无菌厌氧瓶中,同时加入4 g MgCO3用于控制pH,发酵条件为38 ℃、200 r/min,培养60 h。在厌氧阶段发酵过程中,每隔12 h添加800 g/L的葡萄糖维持发酵液的葡萄糖浓度在5 g/L,同时添加碳酸镁维持发酵液的pH在6.2以上。
有氧发酵罐发酵:采用5 L发酵罐进行有氧分批发酵。发酵罐初始装液量为3 L。将二级种子液按总体积10% (体积分数)的接种量接种至发酵培养基中,开始有氧发酵,发酵温度为38 ℃,初始转速为500 r/min,通入100%无菌过滤空气进行有氧发酵,通气量为0.7 vvm (vvm表示每分钟通气量与罐体实际料液体积的比值),先通过调整转速将溶氧(dissolved oxygen, DO)维持在20%以上,待转速升至800 r/min后通过调整通气量维持DO。通过流加纯氨水将pH维持为7.0,培养8 h左右,溶氧DO上升,40 g/L的初始葡萄糖消耗完全,随后通过流加800 g/L葡萄糖,通过自动控制不同时间段的补糖速率维持葡萄糖浓度为1 g/L,通过加入MgCO3控制pH,发酵至72 h结束。
厌氧发酵罐发酵:采用5 L发酵罐进行厌氧分批发酵。发酵罐初始装液量为3 L。将二级种子液按总体积10% (体积分数)的接种量接种至发酵培养基中,发酵温度为38 ℃,转速为200 r/min,通入100%无菌过滤CO2进行厌氧发酵,通气量为0.1 vvm,通过流加纯氨水将pH维持为7.0,培养8 h左右,pH上升,40 g/L的初始葡萄糖消耗完全,随后通过流加800 g/L葡萄糖,通过自动控制不同时间段的补糖速率维持葡萄糖浓度为1 g/L,通过加入MgCO3控制pH,发酵至72 h结束。
两阶段发酵罐发酵:采用5 L发酵罐进行两阶段补料分批发酵。发酵罐初始装液量为3 L。将二级种子液按总体积10% (体积分数)的接种量接种至发酵培养基中,开始发酵。有氧阶段:发酵温度38 ℃,初始转速为500 r/min,通入100%无菌过滤空气,通气量为0.7 vvm,发酵过程中通过流加纯氨水将pH维持为7.0;培养8 h左右,溶氧DO上升,40 g/L的初始葡萄糖消耗完全,随后通过补加800 g/L葡萄糖维持葡萄糖浓度为1 g/L,约8.5 h,停止通气,降低转速至200 r/min,发酵温度38 ℃,转速200 r/min,随后流加800 g/L葡萄糖,通过自动控制不同时间段的补糖速率维持葡萄糖浓度为5 g/L,通过加入MgCO3用于控制pH,发酵至72 h结束。
1.2.4 分析检测方法
细胞浓度测定:取发酵液进行适当稀释,使用紫外分光光度计在波长600 nm的条件下测定OD600。大肠杆菌细胞干重(dry cell weight, DCW)=0.41×吸光值×n (n为样品的稀释倍数),最后计算细胞的生物量。
葡萄糖浓度测定:取发酵液12 000 r/min离心8 min,收集上清液进行适当稀释,使用M-100生物传感器测定葡萄糖浓度(准确测量范围0–2 g/L)。
辅因子(ATP、NAD+和NADH总量)测定:取不同时间的发酵液,用pH为7.4的0.01 mol/L PBS溶液洗涤3次,并稀释至OD600=1.0,使用ATP检测试剂盒S0027以及NADH检测试剂盒(WST-8法)进行测定(上海碧云天生物科技有限公司)。
有机酸浓度测定:取发酵液12 000 r/min离心8min,收集上清液,高效液相色谱(high performance liquid chromatography, HPLC)测定琥珀酸、乳酸、甲酸和乙酸的含量。HPLC检测条件为,色谱分离柱为Aminex HPX-87H (300 mm×7.8 mm),流动相为5 mmol/L稀硫酸,柱温52 ℃,紫外检测波长为210 nm,进样量为10 µL,流速0.6 mL/min。
2 结果与分析
2.1 大肠杆菌发酵生产琥珀酸的化学计量学分析
在5 L发酵上研究了不同氧气条件下E. coli FMME-N-26生产琥珀酸的情况,结果如图 1所示。有氧发酵罐发酵条件下,胞内ATP与NADH含量分别为4.52 μmol/(L·g DCW)和27.53 μmol/(L·g DCW),乙酸浓度为1.19 g/L,琥珀酸几乎不积累。厌氧发酵罐发酵条件下,ATP与NADH含量分别为0.24 μmol/(L·g DCW)和0.58 μmol/(L·g DCW),NADH/NAD+比值为0.22,琥珀酸产量为59.25 g/L,副产物乙酸浓度为16.62 g/L,比有氧条件提高了129.66%,可能与胞内ATP含量不足有关[20]。在两阶段发酵罐发酵条件下,胞内ATP与NADH含量分别为1.21 μmol/(L·g DCW)和17.08 μmol/(L·g DCW),NADH/NAD+比值为0.48,分别比厌氧条件提高了404.17%、2 844.83%、118.18%,此时琥珀酸产量为101.70 g/L,而乙酸浓度降为5.74 g/L。上述结果表明,菌株E. coli FMME-N-26高效生产琥珀酸的条件是微厌氧,且在生产过程中需要借助TCA循环为r-TCA途径提供一定量的NADH和ATP。为了研究胞内NADH和ATP含量如何影响菌株E. coli FMME-N-26生产琥珀酸,由式(1)计算得到大肠杆菌中葡萄糖经过糖酵解途径生成磷酸烯醇式丙酮酸(PEP)的过程可生成2 mol NADH。结合公式(1)+(2)得公式(3),发现有氧条件下1 mol葡萄糖可生成1 mol琥珀酸,产生5 mol NADH和2 mol ATP;结合公式(1)+(4)得公式(5),发现厌氧条件下,大肠杆菌中PEP可直接经r-TCA循环生成琥珀酸,也即1 mol葡萄糖可生成2 mol琥珀酸,但需要补充2 mol NADH,且无ATP生成。通过调节大肠杆菌的PP途径与糖酵解途径的碳通量比和激活乙醛酸循环使得71.4%的碳流向r-TCA途径,28.6%碳流向TCA循环,从而获得最大产率1.714 mol/mol [9, 21-22] (图 1E)。通过(3)×0.286+(5)×0.714得公式(6),从而得到葡萄糖转化为琥珀酸不需要额外的NADH,并有额外ATP生成来维持细胞代谢,避免生产琥珀酸过程中NADH与ATP不足的问题。表明部分碳流向TCA循环以生成能量与辅因子,从而缓解因能量与辅因子不足而导致r-TCA合成琥珀酸能力下降的瓶颈。
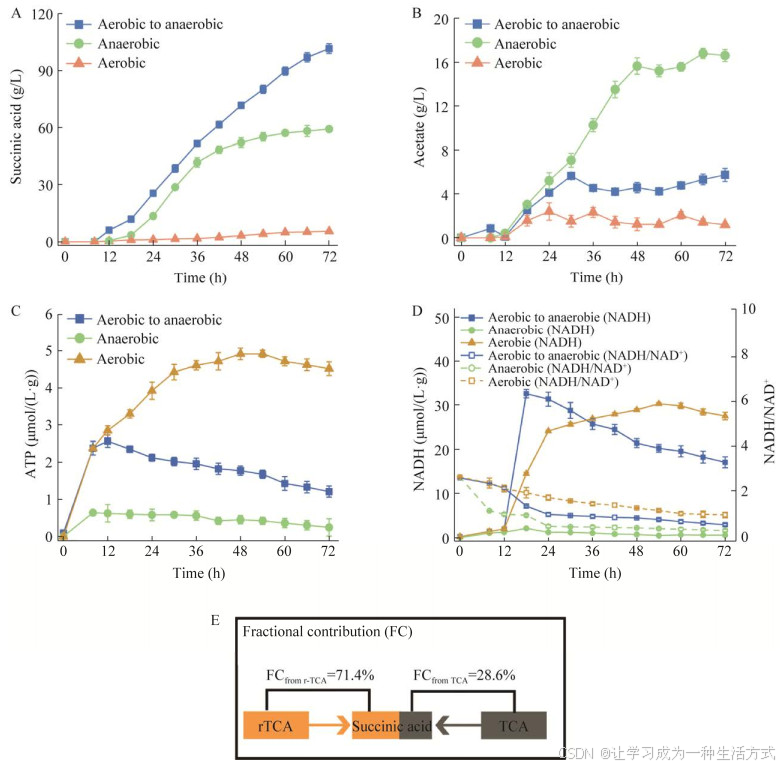
图 1 不同氧气条件下产酸情况与辅因子水平
在有氧发酵、厌氧发酵和两阶段发酵3种不同条件下琥珀酸浓度(A)、乙酸浓度(B)、ATP浓度(C)和NADH浓度(D)变化曲线. E:大肠杆菌利用葡萄糖生产琥珀酸达到理想最大产率条件下的碳通量示意图
|
| (1) |
|
| (2) |
|
| (3) |
|
| (4) |
|
| (5) |
|
| (6) |
2.2 提高胞内ATP水平促进琥珀酸的高效合成
提高胞内ATP含量的代谢工程策略包括减少ATP消耗和强化ATP合成。为了减少ATP消耗,在E. coli FMME-N-26菌株中异源表达来源于运动假单胞菌(Zymomonas mobilis)的葡萄糖扩散蛋白基因(Zmglf),该基因表达的葡萄糖扩散蛋白(glucose facilitated diffusion protein, GLF)能更好地与大肠杆菌的葡萄糖激酶(glucokinase, Glk)配合,在提高葡萄糖利用效率的同时提高琥珀酸的得率,减少了生产琥珀酸过程中ATP的消耗[23-24],另外通过表达Zmglf,也有利于莽草酸[17, 25]生产,在E. coli FMME-N-26中导入带有Zmglf基因的组成型质粒pJ01,得到突变株E. coli FW-1,在两阶段摇瓶发酵中,ATP含量、琥珀酸产量分别为0.92 μmol/(L·g DCW)、69.43 g/L,比出发菌株E. coli FMME-N-26分别提高了17.9%、9.3%,同时,乙酸浓度降低了23.4% (图 2A、2B)。为了强化ATP合成,在菌株E. coli FMME-N-26中通过组成型质粒PJ01过表达磷酸甘油酸变位酶基因pgm和产琥珀酸放线杆菌的磷酸烯醇式丙酮酸激酶基因Aspck。磷酸甘油酸变位酶(PGM)是磷酸甘油酸激酶下游的一种关键糖酵解酶[26],过表达PGM提高3-磷酸甘油醛到1, 3-二磷酸甘油酸的碳通量,从而提高ATP的合成;A. succinogenes的磷酸烯醇式丙酮酸激酶(phosphoenolpyruvate kinase, PCK)可以在催化磷酸烯醇式丙酮酸生成草酰乙酸的同时吸收1 mol CO2和1 mol ATP[18],在大肠杆菌中异源表达编码该酶的Aspck基因可以促进琥珀酸生产[12]。在两阶段摇瓶发酵中,与出发菌株比较,过表达pgm的突变菌株E. coli FW-2胞内ATP含量提高了100% [1.56 μmol/(L·g DCW)],琥珀酸产量无明显差别,但乙酸浓度降低了16.7% (8.22 g/L)。而过表达Aspck的突变菌株E. coli FW-3胞内ATP含量和琥珀酸产量分别为1.15 μmol/(L·g DCW)、67.71 g/L,比出发菌株分别提高了47.4%和6.5%,同时,乙酸浓度降低了33.7% (6.54 g/L)。进一步将Aspck基因整合到菌株E. coli FMME-N-26基因组编码丙酮酸脱氢酶poxB处,构建突变菌株E. coli FW-4,在两阶段摇瓶发酵中,ATP含量和琥珀酸产量分别为1.33 μmol/(L·g DCW)、69.40 g/L,与出发菌株相比,分别提高了57.0%和9.2%。为了进一步提高琥珀酸产量,在突变菌株E. coli FW-4的基础上,借助RBS0034、RBS0031、RBS0011,通过PJ01质粒将pgm和Zmglf基因表达水平控制在高(H)、中(M)、低(L) 3个层级(图 2C、2D),获得了9株突变菌株(图 2E),其中当pgm和Zmglf基因分别处于高水平和中水平表达时,突变菌株E. coli FW-6在两阶段摇瓶发酵中ATP含量和琥珀酸产量分别为1.35 μmol/(L·g DCW)、71.66 g/L,比出发菌株E. coli FMME-N-26分别提高了73.1%和11.6%,此外乙酸浓度降低至5.3 g/L (图 2F–2H)。上述结果表明,提高胞内ATP含量能有效地降低副产物乙酸浓度,进一步提高琥珀酸产量。
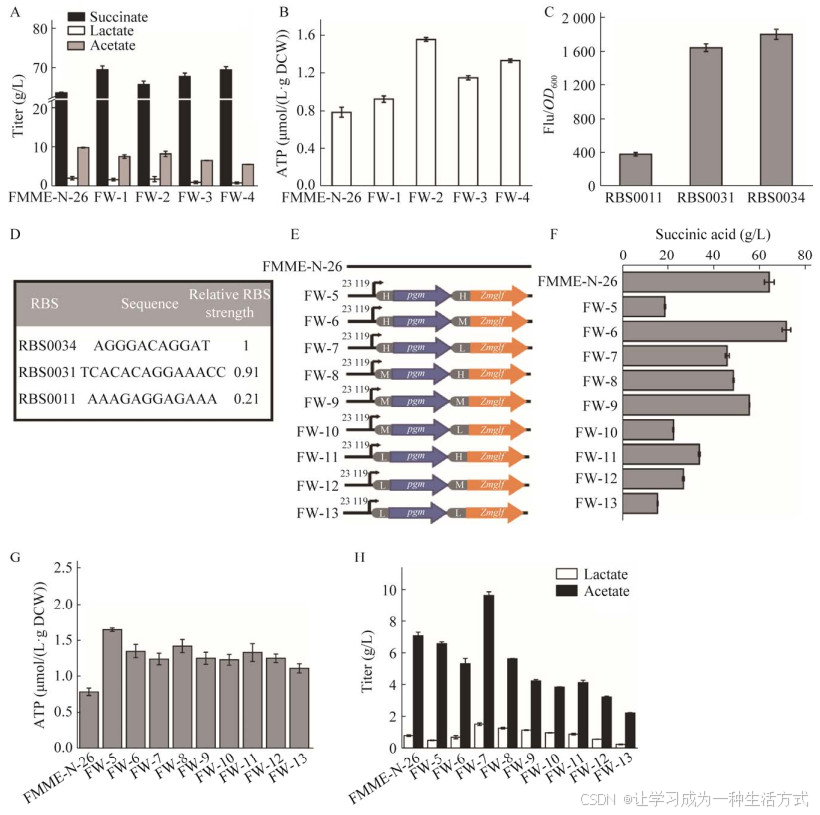
图 2 调节ATP平衡的菌株构建与评估Fig. 2 Construction and evaluation of strains regulating ATP balance.菌株E. coli FMME-N-26、FW-1、FW-2、FW-3、FW-4发酵产酸情况(A)和ATP含量(B). C:3种不同RBS的强度. D:3种不同RBS的序列情况与相对强度. E:不同表达水平的pgm和Zmglf表达组合. pgm和Zmglf不同表达组合的琥珀酸产量(F)、ATP含量(G)、乙酸和乳酸浓度(H)
2.3 调节NADH水平促进琥珀酸的高效合成
提高胞内NADH含量的代谢策略包括阻断NADH竞争途径和构建NADH回补路径。乙醇脱氢酶(ADHE)催化乙酰辅酶A消耗NADH还原为乙醛,在菌株E. coli FW-6中敲除编码乙醇脱氢酶的基因adhE,得到突变菌株E. coli FW-14 (图 3A、3B),其琥珀酸产量、NADH浓度和NADH/NAD+比值分别比菌株E. coli FMME-N-26提高了18.91% (75.57 g/L)、8.0% [19.76 μmol/(L·g DCW)]和21.4% (0.51),但菌体量降低了10.5% (附图 1)。gapA基因编码的磷酸甘油醛脱氢酶是糖酵解中生成NADH的主要来源,表达此基因在增加了胞内NADH含量的同时也提高了葡萄糖利用率[27-28],在菌株E. coli FW-6中过表达gapA基因,得到菌株E. coli FW-15,其琥珀酸产量和NADH浓度分别提高了21.0% (76.87 g/L)、14.0% [20.12 μmol/(L·g DCW)],但NADH/NAD+比值不发生变化(0.42)。pncB基因编码的烟酸磷酸核糖基转移酶(niacin phosphoribosyltransferase, NAPRTase)是一种NAD(H)合成途径中的限制酶,过表达pncB基因可以提高NAD(H)浓度,使得菌体量和琥珀酸产量在厌氧条件下显著增加[11],在菌株E. coli FW-6中过表达pncB基因,得到突变菌株E. coli FW-16,其琥珀酸产量、NADH浓度和NADH/NAD+比值分别提高了22.35% (77.75 g/L)、22.7% [22.46 μmol/(L·g DCW)]、13.0% (0.55)。为进一步提高琥珀酸产量,在菌株E. coli FW-6中借用RBS0034、RBS0031、RBS0011将gapA和pncB表达水平控制在高(H)、中(M)、低(L) 3个水平,构建了9株基因工程菌(图 3C)。其中当gapA、pncB基因都高水平表达时,突变菌株E. coli FW-17琥珀酸产量、胞内ATP浓度、NADH浓度、NADH/NAD+比值分别为85.67 g/L、2.10 μmol/(L·g DCW)、28.75 μmol/(L·g DCW)和0.65,比对照菌株E. coli FMME-N-26分别提高了34.8%、169.23%、28.75%和54.76%,同时乙酸和乳酸分别下降了72.92%和41.47% (图 3D–3F)。
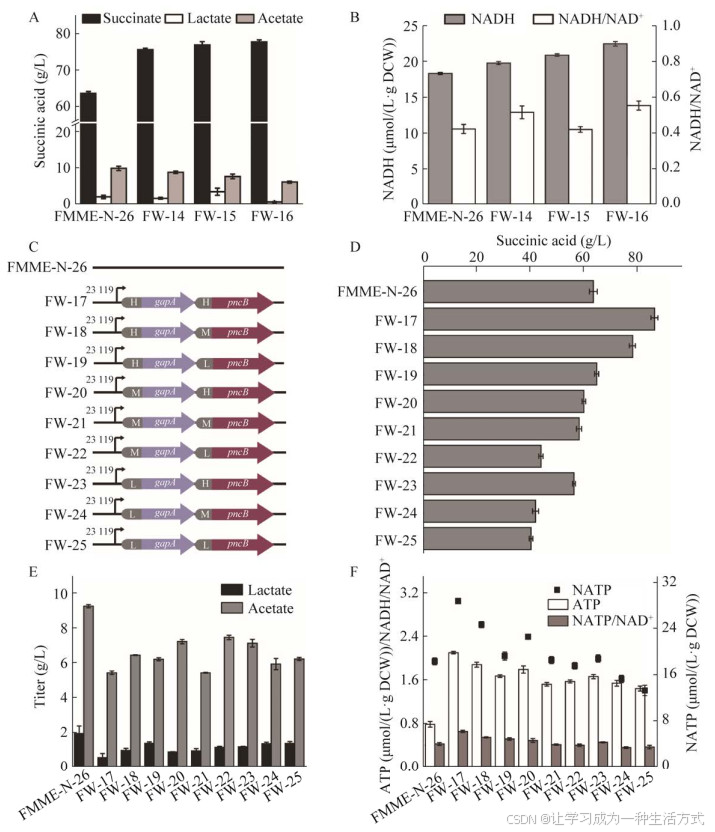
图 3 调节NADH平衡的菌株构建与评估Fig. 3 Construction and evaluation of strains in which the NADH balance is regulated.菌株E. coli FMME-N-26、FW-14、FW-15、FW-16发酵产酸情况(A)、NADH含量与NADH/NAD+比值(B). C:构建了一系列不同表达水平的gapA和pncB表达组合. gapA和pncB不同表达组合的琥珀酸产量(D)、乙酸和乳酸浓度(E)、ATP含量、NADH含量和NADH/NAD+比值(F)
2.4 发酵条件优化提高琥珀酸的产量
最优突变菌株E. coli FW-17在5 L发酵罐中,两阶段发酵罐发酵生产琥珀酸的过程曲线如图 4A所示。发酵72 h后,琥珀酸产量和生产强度为118.52 g/L、1.73 g/(L·h),比出发菌株E. coli FMME-N-26分别提高了16.88%和9.49%,副产物乙酸浓度降低为4.32 g/L (图 4A,表 4)。但从图 4B发现在发酵后期(48 h后)斜率下降了13.3%,这一结果表明菌株代谢能力下降,这也是为何发酵后期残糖升高和乙酸积累的原因。为此,将发酵过程中残糖浓度分别控制在0.0、1.0、5.0 g/L,结果表明将残糖控制在1.0 g/L时琥珀酸浓度提高到139.63 g/L,比优化前提高了17.81%,副产物乙酸浓度降低了67.59% (1.40 g/L),同时发现,副产物乙酸的浓度会随着残糖浓度的升高而逐渐升高(图 4C、4D);发酵液中糖分不足时,pH上升。综上所述,通过调整补糖速率,可以在提高E. coli FW-17琥珀酸生产能力的同时,降低发酵液中的乙酸积累。
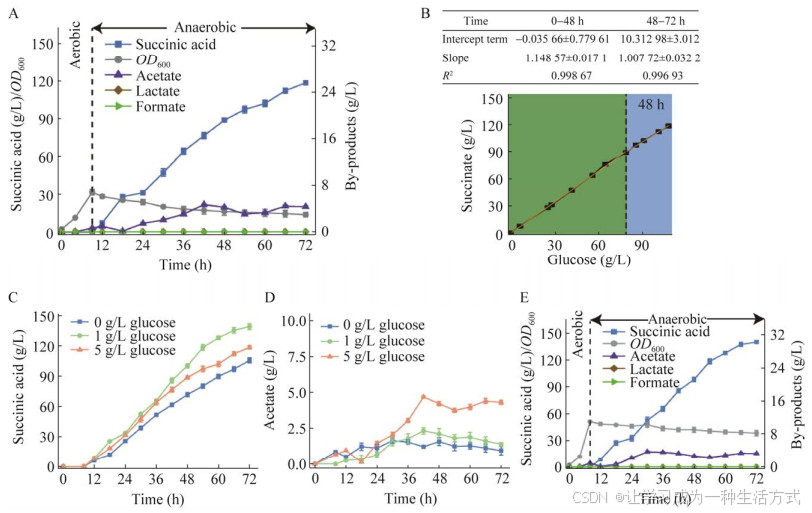
图 4 菌株Escherichia coli FW-17发酵结果Fig. 4 Fermentation profile of Escherichia coli FW-17.A:E. coli FW-17的产酸、OD600与葡萄糖浓度变化曲线. B:菌株E. coli FW-17在发酵过程中琥珀酸与葡萄糖关系图. C:不同糖浓度条件下琥珀酸的生产情况. D:乙酸积累情况. E:菌株E. coli FW-17在1 000 L罐的产酸、OD600与葡萄糖浓度变化曲线
Table 4 Summary of fermentation parameters under different conditions
| Condition | Organic acid concentration (g/L) | Productivity (g/(L·h)) | |||
| Succinate | Acetate | Lactate | Formate | ||
| E. coli FMME-N-26 | 101.40 | 5.50 | ‒ | ‒ | 1.58 |
| E. coli FW-17 | 118.52 | 4.32 | ‒ | ‒ | 1.73 |
| 0 g/L glucose | 105.70 | 1.44 | ‒ | ‒ | 1.47 |
| 1 g/L glucose | 139.52 | 1.40 | ‒ | ‒ | 1.94 |
| 5 g/L glucose | 118.53 | 4.32 | ‒ | ‒ | 1.65 |
| 1 000 L scale-up | 140.20 | 1.38 | ‒ | ‒ | 1.95 |
| ‒ means undetected. | |||||
进一步将菌株E. coli FW-17于1 000 L发酵罐中进行放大实验,结果如图 4E所示。在最优发酵条件下(残糖控制在1.0 g/L的两阶段发酵罐发酵),在发酵72 h后琥珀酸的产量为140.2 g/L,生产强度为1.95 g/(L·h),见图 4E、表 1,相比于原始菌株E. coli FMME-N-26分别提高了38.26%和23.42%,乙酸浓度为1.38 g/L,降低了74.91%。这与5 L罐发酵水平相当,说明了该菌株与发酵工艺具有工业生产潜力。
3 讨论与结论
通过化学计量学分析,有助于从机理角度去分析琥珀酸生产瓶颈,找到提高琥珀酸产量的方法。在发酵生产过程中难免会出现产量提升困难的情况,需要从菌株改造、培养基优化等方面进行综合考虑,找到瓶颈问题。例如,Lin[29]开发了好氧琥珀酸生产系统能够在好氧条件下实现1.0 mol/mol葡萄糖的最大理论琥珀酸产量;Li等[30]采用正交试验设计,以玉米浸膏和酵母浸膏为葡萄糖和氮源,确定最佳浓度,获得维生素B12的产率为530.29 μg/g DCW。其中基于化学计量学分析进行有机酸合成路径改造达到较好的效果[18, 22, 31],但只考虑了单一条件下的情况,往往与实际脱离。在本研究中,为了探究菌株E. coli FMME-N-26琥珀酸能力下降的瓶颈,基于3种不同发酵条件下的产酸情况进行化学计量学分析,得出了一种调节胞内NADH与ATP水平以进一步提高琥珀酸产量的代谢工程策略。
胞内辅因子不足会影响琥珀酸合成,降低琥珀酸产量。在琥珀酸生产中涉及的辅因子主要为ATP与NADH,提高胞内ATP与NADH含量的方法主要为表达生成ATP与NADH相关基因或降低ATP与NADH消耗相关的基因,马江峰等[11]通过表达了烟碱酸磷酸核糖基转移酶(NAPRTase, pncB)和源自乳酸链球菌(Lactococcus lactis)的pyc基因,使NADH的浓度提高了9.8倍,琥珀酸产量提高了8.0倍,达到14.1 g/L,Kim等[12]在PPC突变的菌株中表达产琥珀酸放线杆菌的PCK后,将PEP羧化与ATP生成相结合,琥珀酸的产量较出发菌株提高了6.5倍。但是过表达基因后,容易使胞内氧化还原失衡,缺失了ldhA和pflB基因的大肠杆菌,由于无法再生NAD+,造成氧化还原失衡,无法在厌氧条件下利用葡萄糖[11, 32]。为了解决这一问题,需要调整胞内氧化还原平衡。在本研究中,为了解决菌株E. coli FMME-N-26胞内ATP与NADH含量不足,使得发酵后期生产强度降低这一问题,进一步提高琥珀酸浓度,本研究先表达Aspck、pgm和Zmglf基因提高胞内ATP含量,表达gapA和pncB基因并敲除NADH消耗的adhE基因提高胞内NADH含量;随后通过组合协调pgm、Zmglf、gapA和pncB的表达,在提高ATP与NADH含量的同时,维持胞内氧化还原平衡,将琥珀酸产量提高至118.52 g/L。
通过发酵条件的优化,琥珀酸产量进一步提高。经1 000 L发酵罐的生产,最优菌株FW-17生产琥珀酸浓度为140.2 g/L,生产强度为1.95 g/(L·h)。本研究证明了大肠杆菌生产琥珀酸的工业化潜力。此外,本文还为以大肠杆菌为工业平台的生物基化学品生产的未来发展提供了指导方针。