【文献分享】利用 GeneTEA 对基因描述进行自然语言处理以进行过表达分析
文章目录
- 介绍
- 代码
- 参考
介绍
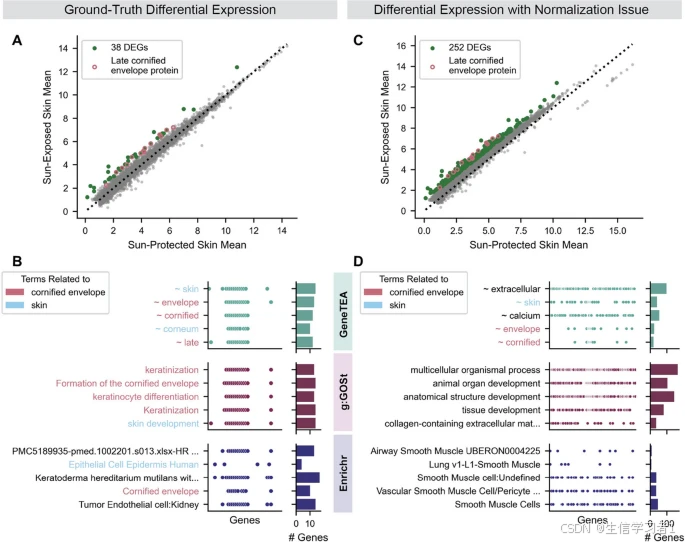
过度表型分析用于识别基因列表中的生物学富集现象。在此,我们介绍了 GeneTEA,这是一个能够接收自由文本基因描述并结合自然语言处理方法来学习稀疏的基因-项嵌入的模型,该嵌入可以被视为一个新的基因集数据库。在与现有过度表型分析工具的基准测试中,只有 GeneTEA 能够正确控制假发现率,同时始终能揭示最相关的生物学信息,并且这样做的冗余度更低。我们表明,同样的方法可以应用于其他生物体的基因组或化合物。此外,我们还提供了训练好的 GeneTEA 模型的交互式应用程序和 API。
技术的进步使得能够转向基于基因组规模、旨在产生假设的实验。因此,过度表达分析(ORA)应运而生,用于从这些高维数据中获取生物学见解。该方法涉及对来自大规模实验的查询基因列表进行测试,以检测编码生物过程、分子功能、表型或其他已有知识的基因集的统计富集情况[1]。关于基因集并没有统一的定义,如今它们在许多数据库中(包括基因本体论(GO)[2]、人类表型本体论(HPO)[3]、分子特征数据库[4]、京都基因与基因组百科全书(KEGG)[5]、维基通路(WP)[6]和反应子数据库(REAC)[7])中是相互独立定义的。
已经开发出了多种工具,用于同时在这些数据库上运行 ORA,其中 g:Profiler 的 g:GOSt [8] 和 Enrichr [9] 是最受欢迎的工具。然而,这种方法也存在诸多问题:
基因集数据库的大量增长导致出现了许多重复、相互矛盾以及定义不清的基因集[10,11,12]。
这些数据库内部及之间的基因集之间存在高度重叠的情况,已被证实会降低 ORA 的特异性[13]。
显著值的大小与所查询的基因集合库的规模直接相关,这使得从那些跨多个数据库进行汇总的工具中得出的结果难以解读[14]。
许多工具都存在较高的假阳性率问题,这通常是因为背景定义不当以及对所进行的并行测试数量的低估所致[15]。
综合来看,这些问题表明,尽管 ORA 已得到广泛应用,但仍存在有待改进的空间。

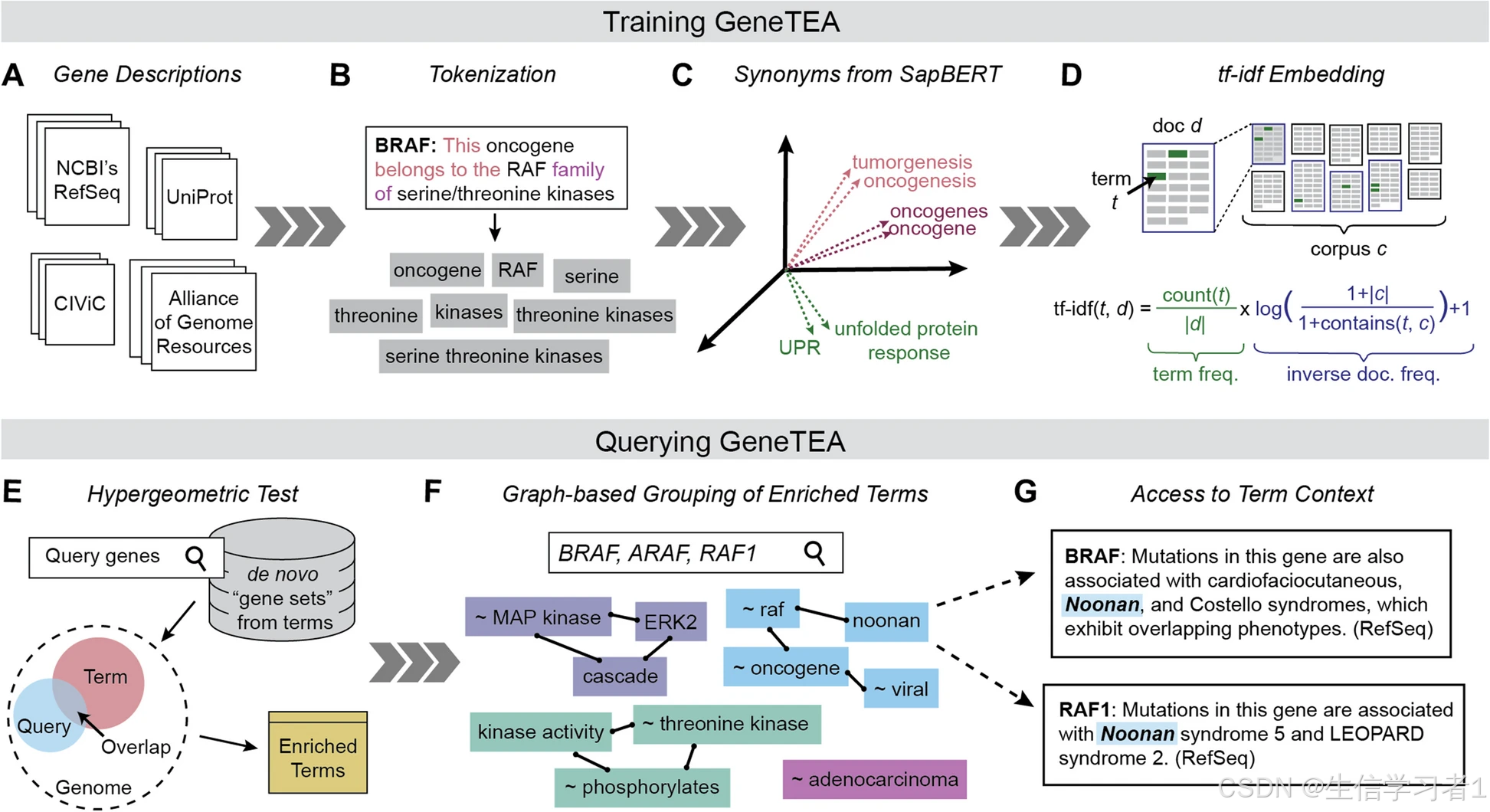
基因TEA 模型概述。A 该训练语料库由自由文本的基因描述构建而成。B 词的分词示例。C 对于每个词的 SapBERT 嵌入的表示,根据所分配的同义词集进行着色。D 表示词频 - 逆文档频率(tf-idf)嵌入的图表和方程。E 表示使用超几何检验来识别富集术语的图形。F 与查询“BRAF、ARA F、RAF1”相关的术语组示例。G 引用“诺南”一词在 BRAF 和 RAF1 中的文本摘录。
代码
https://github.com/broadinstitute/GeneTEA
参考
- Natural language processing of gene descriptions for overrepresentation analysis with GeneTEA
- https://github.com/broadinstitute/GeneTEA
