Nature Methods | GHIST vs LOKI:HE→表达的两条路径 | 单细胞级重建 vs spot级预测
引言:空间转录组与HE图像的融合
空间转录组技术能够在二维或三维组织中解析基因表达的空间分布,但现有平台成本高、实验流程复杂,限制了其广泛应用。而病理学常用的HE(hematoxylin–eosin)染色切片廉价、易获取,并含有反映细胞及组织结构的丰富信息。如果能够利用HE图像预测空间基因表达,便可在无需额外测序的情况下,为大量既存样本赋予新的“空间转录组”层次,助力肿瘤异质性分析和生物标志物发现。之前介绍了LOKI这个平台,这次继续学习GHIST这个平台方法。

围绕用 H&E 推断分子层信息,GHIST 面向单细胞分辨率的表达重建,推理时只依赖 H&E;而 LOKI(基于 OmiCLIP)提供对齐、注释、分解、检索等模块,其中 PredEx 能在 Visium 斑点尺度从 H&E 预测基因表达,适合多任务工作流。两者共同降低了对昂贵空间测序的依赖,但分辨率与输出形态不同,应据研究目的选择。
GHIST工具:多任务学习链接多层信息
GHIST(Gene expression from HISTology)是9月发表在Nature Methods上的一种深度学习框架,它通过利用训练阶段的亚细胞空间转录组(SST)数据,将HE图像映射为单细胞空间基因表达图像。其核心特征包括:
-
四重信息协同学习:GHIST不仅处理HE图像,还同时学习细胞类型、邻域细胞组成、核形态和单细胞RNA表达四种信息。通过多任务架构和互相约束的损失函数,这些信息在不同预测头之间传递,使模型捕捉到细胞类型难以区分或受邻居影响时的表达特征,从而提高预测准确性。
-
单细胞级空间分辨率:以往方法多基于10× Visium等斑点平台,只能预测几十微米的斑块中的混合表达,难以解析单细胞异质性。GHIST利用亚细胞分辨的空间转录组数据训练,能在推理阶段仅依据HE图像预测每个细胞的数百个基因表达值,并生成与细胞定位一致的多通道表达图像。
-
无需匹配的单细胞RNA参照:训练时可使用公开单细胞RNA测序数据提供的细胞类型信息,而这些单细胞数据无需与输入的HE图像配对。模型训练完成后,在推理时不再需要空间转录组或其他组学数据,这一点使其具有实际应用潜力。
-
优于斑点级方法的性能:在HER2+乳腺肿瘤等数据集上,GHIST在Pearson相关系数(PCC)和结构相似度(SSIM)等指标上均明显优于ST‑Net、Hist2ST、DeepPT等方法。尤其是在高变异基因和空间差异基因(SVG)上,GHIST的PCC分别达到0.20和0.27,高于其他方法。
下面的图展示了GHIST的总体框架和示例预测效果。
GHIST框架示意
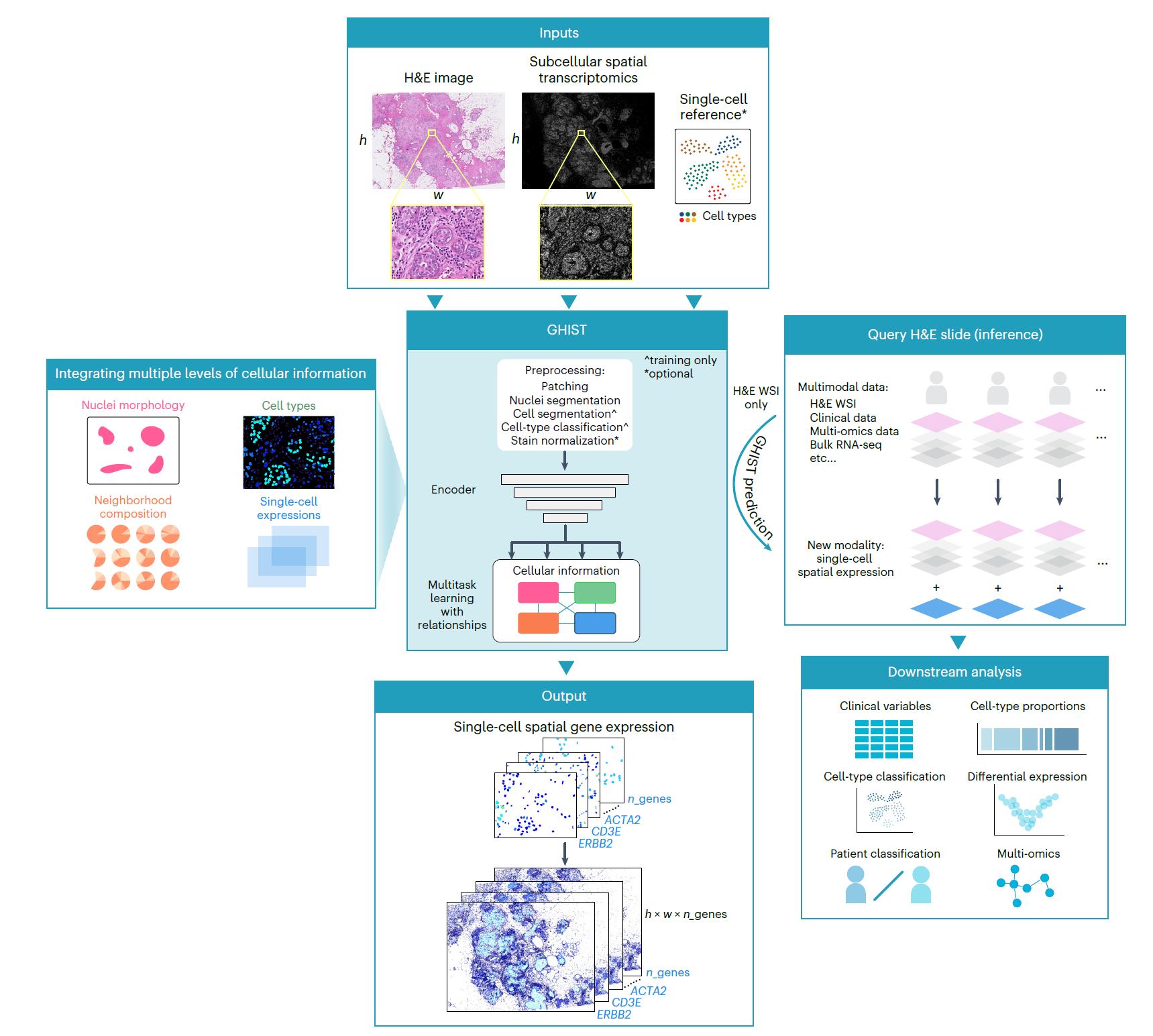
图中,模型利用HE图像、亚细胞空间转录组和单细胞参考信息作为输入,通过核形态、邻域组成、细胞类型和单细胞表达四个预测头在训练阶段联合优化;模型经训练后即可仅凭HE全景图像推断出单细胞空间基因表达,并可进一步用于临床变量关联、差异表达分析、患者分层和多组学整合等分析场景。
两个乳腺癌样本的GHIST预测结果
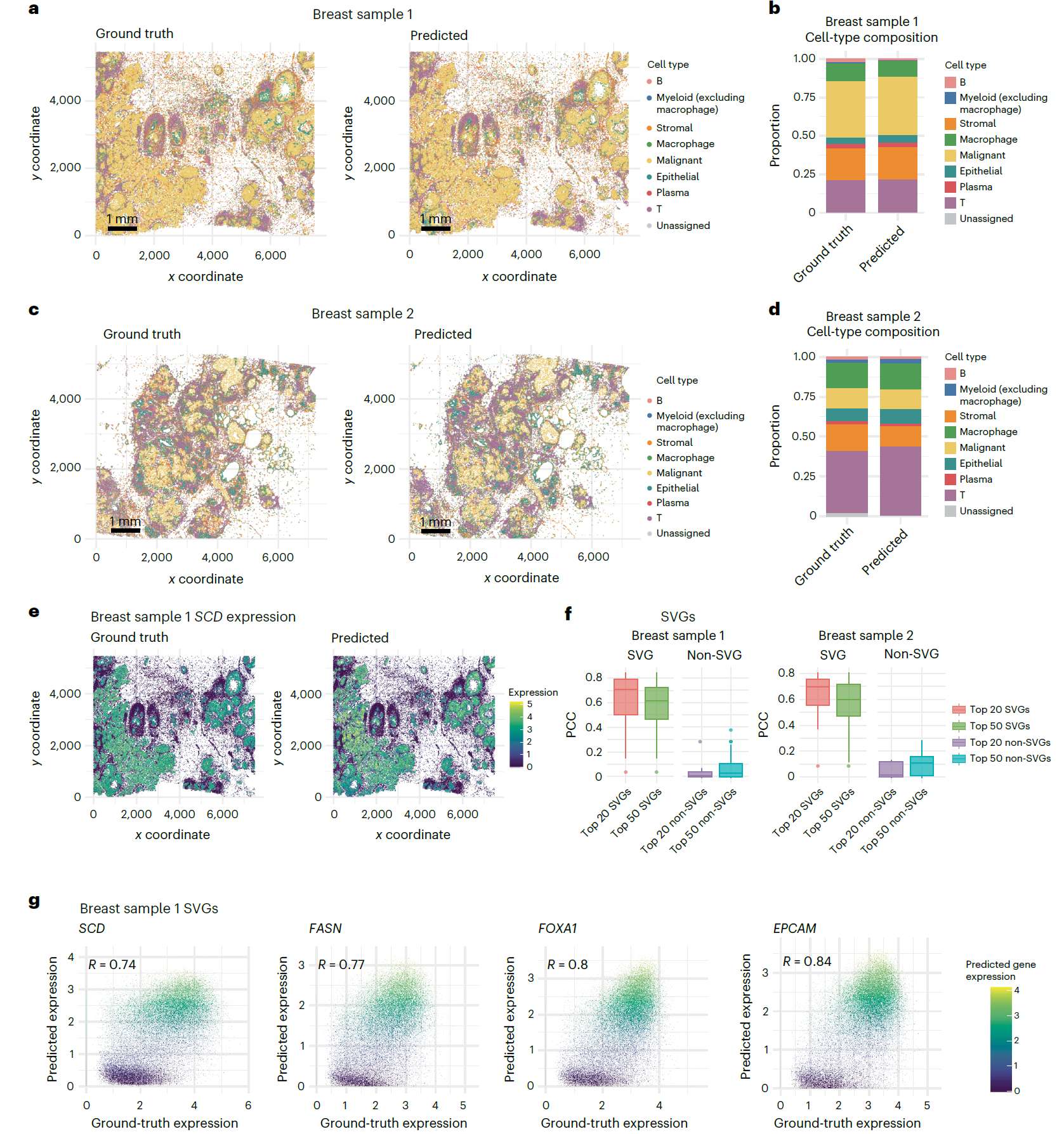
面板a-d比较了BreastCancer1和BreastCancer2样本的实际单细胞类型分布与GHIST预测结果,预测与实测的细胞类型分布高度一致。面板e展示了脂肪酸合酶基因SCD在BreastCancer1样本中的真实表达和GHIST预测值;预测值与实测值具有显著相关性。箱线图(f)显示,GHIST在预测空间差异基因(SVGs)和高变异基因(HVGs)时的相关系数显著高于非差异基因,这说明模型能捕捉生物学意义强的基因表达。面板g中的散点图进一步展示了部分基因(如FASN、FOXA1、EPCAM)的预测值与实测值之间的高相关性,表明GHIST在乳腺癌样本中准确回归关键基因表达。
与LOKI平台的简要比较
为更好理解GHIST的特点,与之前发表的Loki平台进行对比。Loki是基于视觉语言双编码模型OmiCLIP的多模态分析平台,旨在统一处理组织学图像和转录组数据。二者的主要异同包括:
| 特性 | GHIST | Loki平台 |
|---|---|---|
| 核心目标 | 从HE图像预测单细胞空间基因表达,重点提升分辨率与生物学解释性 | 构建视觉‑转录组基础模型,为多模态分析提供底座,模块化实现对齐、注释、分解、检索和斑点基因预测 |
| 输入数据 | 训练时需要HE图像与亚细胞空间转录组匹配样本;推理阶段仅需HE图像 | 训练OmiCLIP时使用来自32个器官的220万对图像‑Visium斑点数据,将转录组转换为“基因句子”进行对比学习 |
| 信息整合策略 | 多任务架构同时学习细胞类型、邻域组成、核形态和RNA表达,并在预测头之间共享信息 | 基于CLIP的双编码模型在嵌入空间对齐图像和转录组,提供对齐、注释、细胞类型分解和检索等模块 |
| 分辨率 | 可直接输出单细胞级空间基因表达;也能适配斑点数据 | 主要针对斑点级ST数据,利用预训练嵌入预测斑块基因表达(Loki PredEx模块) |
| 应用范围 | 对肿瘤及其他组织的HE图像进行空间转录组“赋能”,生成可用于细胞类型分析、差异表达和多组学整合的单细胞表达图像 | 提供五个模块:Loki Align用于多切面对齐,Loki Annotate用于多模态注释,Loki Decompose用于细胞类型分解,Loki Retrieve用于图像‑转录组检索,Loki PredEx用于斑点基因预测 |
| 优势与局限 | 优势:能够捕捉细胞微环境信息,实现单细胞分辨率预测,并在乳腺癌、肺腺癌、黑色素瘤等数据中表现优异;局限:训练依赖高分辨空间转录组数据,不易获得 | 优势:利用大规模预训练模型对多种任务进行统一处理,适应不同器官和疾病;局限:目前预测模块以斑点分辨率为主,无法直接输出单细胞表达 |
总的来说,GHIST更聚焦于利用亚细胞空间转录组训练出高分辨率的预测模型,而Loki平台则提供一个多任务的基础设施,可在多种场景中迁移使用。在实际应用中,两者可互补:GHIST生成的单细胞表达图像可作为Loki平台进行下游分析的输入,而Loki的对齐和注释模块也可用于提高GHIST训练数据的质量。
文章主要内容与应用场景
文章的主体包括以下几个部分,分别对应了GHIST的设计、验证及潜在应用场景:
-
方法开发与框架设计:介绍GHIST的总体结构(见图1),包括输入端(HE图像、亚细胞SST、单细胞参考)、多任务编码器以及四个预测头,阐述如何利用多种生物学信息进行联合学习。
-
模型验证与性能评估:作者使用乳腺癌、肺腺癌、黑色素瘤等公开数据集验证GHIST的性能。通过比较预测的细胞类型分布与真实分布、计算空间差异基因的相关系数、评估在斑点数据上的PCC/SSIM等指标,证明GHIST在单细胞和斑点层面均优于现有方法。
-
肿瘤微环境解析:GHIST能够准确预测肿瘤细胞和免疫、间质细胞的空间分布(图2a‑d)。在乳腺癌样本中,模型预测的细胞比例与真实数据高度一致,表明其可用于量化肿瘤微环境中的细胞组成和异质性。
-
关键基因表达回归:文章展示了GHIST对脂肪酸合酶*(FASN)、脂肪酸不饱和酶SCD*、转录因子FOXA1及上皮标志物EPCAM等关键基因的预测结果,预测值与实测单细胞表达具有显著相关性。因此,GHIST可以用于挖掘驱动肿瘤进展的基因或药物靶点。
-
适配斑点平台的扩展:作者将GHIST适配到Visium等斑点平台,通过评估HER2+乳腺癌数据集,展示了模型在斑点层面也明显优于ST‑Net、Hist2ST、DeepPT等方法。这一扩展表明GHIST不仅适用于亚细胞分辨的空间转录组,也可服务于传统斑点数据。
-
下游分析与应用展望:GHIST生成的单细胞空间表达可用于多种生物医学分析,包括:
- 细胞类型注释与定量:在没有空间转录组的情况下,通过GHIST预测的表达与公开单细胞数据比对,快速标注细胞类型,量化不同细胞亚群的空间分布。
- 差异表达与基因富集分析:在空间维度上识别肿瘤与正常区域或不同细胞类型间的差异表达基因,揭示潜在的机制及候选治疗靶点。
- 多组学整合和患者分层:将预测的单细胞表达与临床信息、突变数据或其他组学数据结合,进行患者预后模型构建和精准治疗决策。作者在HER2+乳腺癌队列中展示了利用预测的表达进行生存分析的例子。
- 新实验设计与假设生成:通过对大规模HE切片进行预测,研究者可发现新的空间表达模式和细胞相互作用,为后续的空间转录组实验提供假设和区域选点建议。
资源与权重获取
代码仓库(官方)
https://github.com/SydneyBioX/GHIST
仓库内提供安装说明与 3 类教程(数据预处理、训练与验证、仅用 H&E 的无真值预测)。Releases 显示“无发布”,作者未提供预训练权重;如需在自己的组织/面板上使用 GHIST,需要按教程自行训练模型。推理阶段可仅基于 H&E 切片运行。
环境与依赖(简要提示)
- 官方建议 ≥24 GB 显存 GPU(文中为 RTX 4090);
python=3.10;按requirements.txt安装依赖。 - 需额外安装
stainlib用于染色归一化(如 Macenko、Reinhard、Vahadane)。 - 仓库
tutorials/目录含训练与无真值预测示例,可直接对照改参数跑你自己的切片。([GitHub][1])
(按 tutorials/Training and validation 自训得到与组织/基因面板匹配的 checkpoint;推理可以参考 tutorials/Prediction on data without ground truth。)
使用注意
-
无现成权重:需要用“配对的 H&E + 亚细胞/单细胞空间转录组(如 Xenium/CosMx/MERFISH)”数据自行训练;可预测的基因集合取决于训练面板。
-
颜色与尺度:跨批次/跨中心的染色差异与分辨率不匹配会显著影响泛化;需要进行染色归一化并保持与训练一致的空间尺度。具体染色归一,作者在GitHub中也给出了示例。
结论与前瞻
GHIST的出现拓展了空间转录组学的应用边界。它通过深度学习整合HE图像中的形态信息和空间转录组数据的分子信息,使得在没有空间测序的情况下也能获得接近单细胞分辨率的基因表达图像。
与Loki等多模态平台相比,GHIST的优势在于高分辨率和生物学解释性强,但其训练依赖于高质量的亚细胞空间转录组数据,未来需构建更大规模的训练集以提升泛化能力。
简单来说,GHIST 在训练阶段利用与 H&E 配对的亚细胞/单细胞空间转录组(SST)学习跨模态映射;一旦训练完成,推理时仅需 H&E 即可进行单细胞层面的表达估计。当前官方仓库无发布版权重,需依教程自训模型再应用到个人的 H&E 切片。
展望未来,该方法可进一步结合三维组织切片、实时成像及多模态组学,实现更精细的肿瘤微环境重构与动态观察。随着生成式AI和跨模态大模型的发展,GHIST或能与Loki等平台融合,为精准医学提供更为全面、实时的空间生物信息。
参考文献
- Fu X.等. Spatial gene expression at single‑cell resolution from histology using deep learning with GHIST. Nature Methods 22, 1900–1910 (2025)。
👋 欢迎关注我的生信学习专栏~ 如果觉得文章有帮助,别忘了点赞、关注、评论,一起学习
