【科研绘图系列】R语言绘制模型预测与数据可视化
禁止商业或二改转载,仅供自学使用,侵权必究,如需截取部分内容请后台联系作者!
文章目录
-
- 介绍
- 加载R包
- 数据下载
- 函数
- 导入数据
- 数据预处理
- 画图
- 总结
- 系统信息
介绍
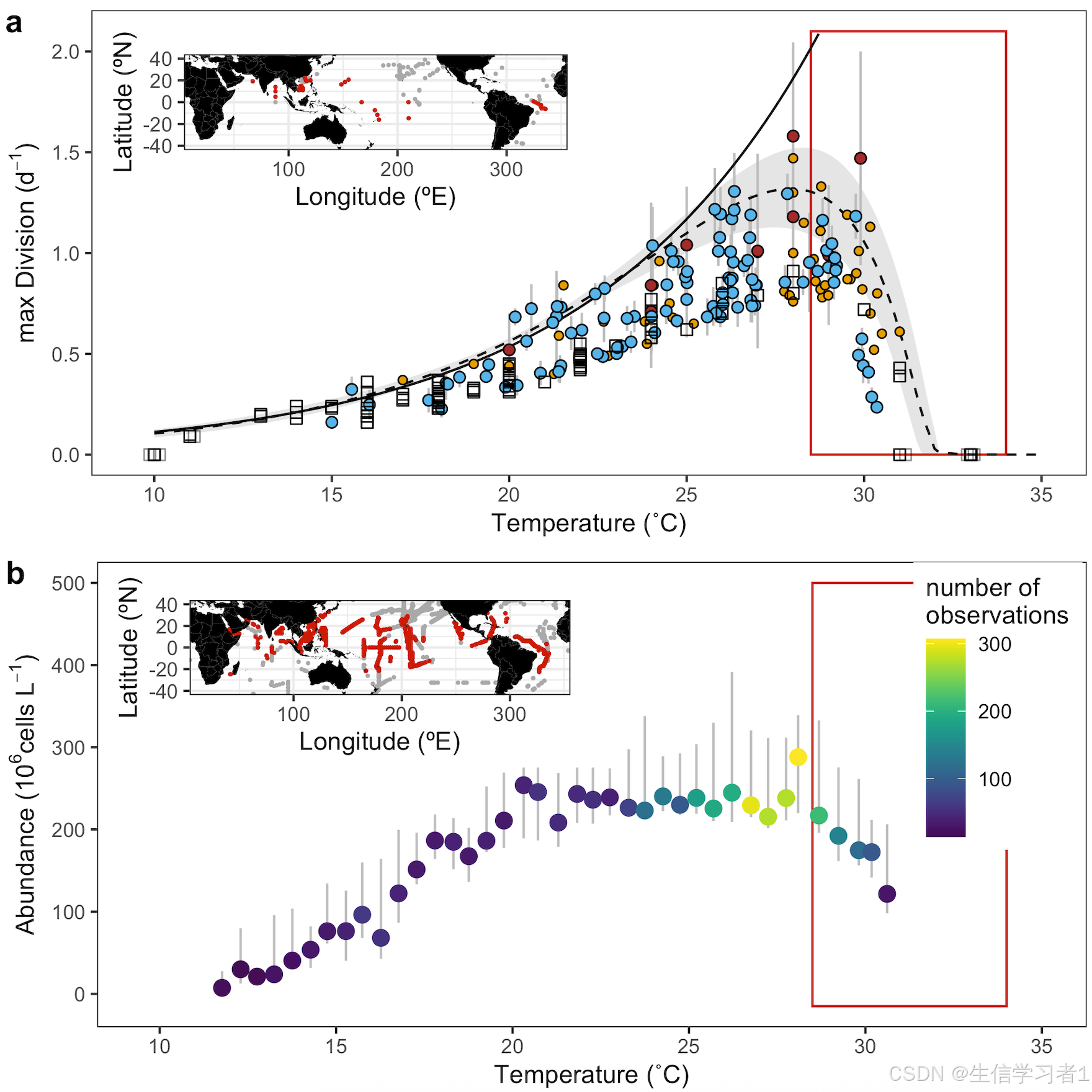
本文介绍了一种利用R语言进行海洋微生物群落动态分析的方法,该方法通过构建多个统计模型来预测不同环境条件下微生物群落的变化。文章首先介绍了五种不同的微生物生长模型,包括指数增长模型(Eppley模型)、带有感染的Eppley-Norberg模型、最佳生长温度模型(Cardinal Temperature Model)、指数增长与感染模型(Thomas模型)以及基于营养的最优生长温度的交互双指数模型(IDE模型)。这些模型考虑了温度、营养盐浓度和光照等环境因素对微生物生长速率的影响。
接着,文章描述了如何从多个数据源加载和预处理数据,包括实验室培养实验、稀释实验、细胞周期分析以及现场观测数据。通过整合这些数据,研究者能够构建一个全面的微生物群落数据集,用于后续的模型预测和分析。
文章的核心部分是利用单细胞转录组学技术对微生物群落进行分析,揭示了微生物群落在不同环境条件下的功能异质性。研究者使用了Bootstrap方法来评估模型预测的不确定性,并计算了Prochlorococcus属的最佳生长温度。此外,文章还展示了如何使用R语言中的tidyverse和ggpubr包进行数据可视化,包括绘制微生物群落对环境变化的响应图和微生物丰度分布图。
最后,文章讨论了单细胞转录组学技术在微生物学研究中的潜力,以及如何通过结合其
