酶 EC number 预测工具CLEAN的安装和使用
写在前面
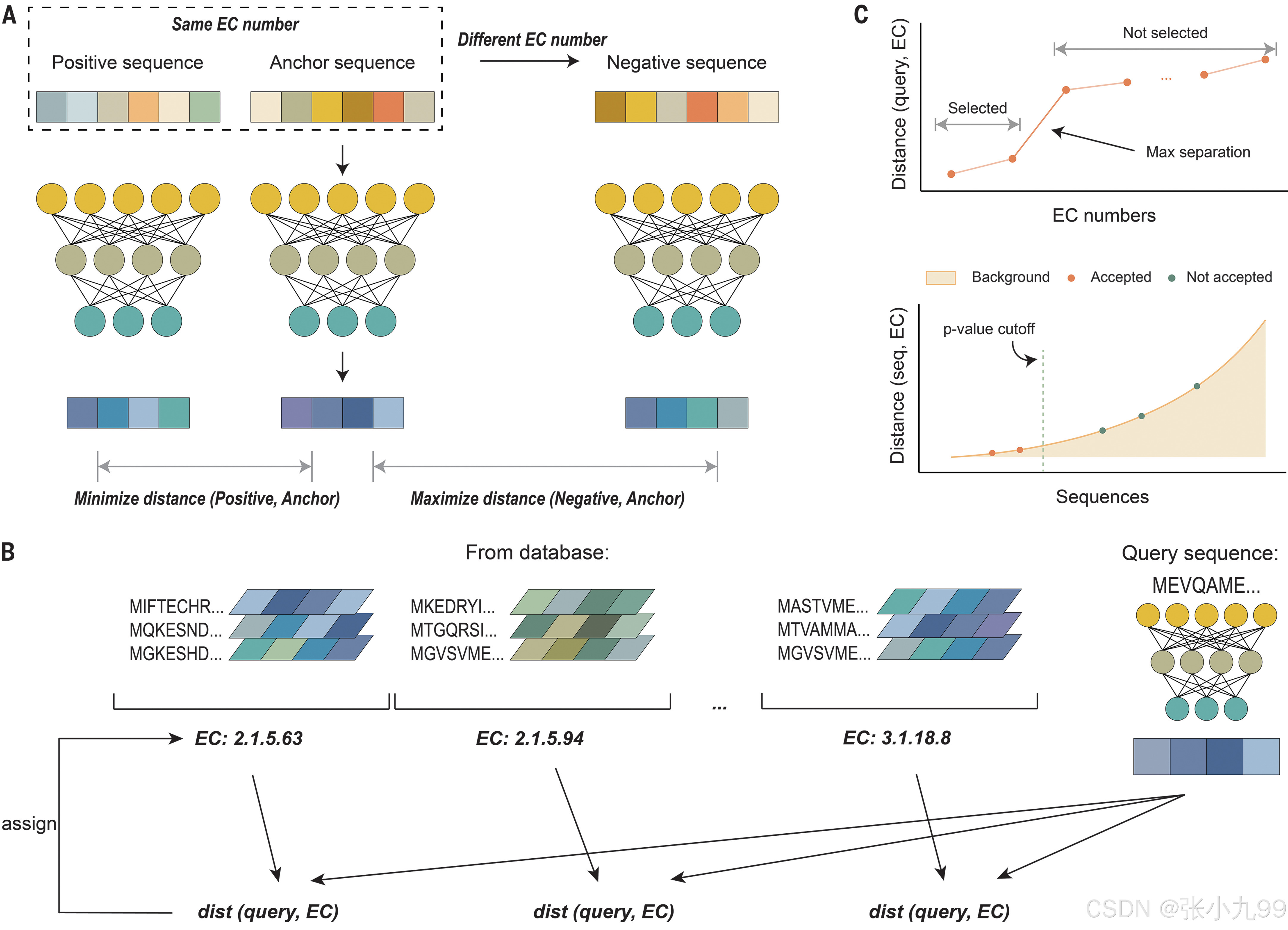
酶往往根据其可以催化的化学反应进行分类,最广为人知的酶催化功能数字分类方案是酶委员会(EC)编号,每个EC编号都与酶催化的化学反应相关,所有催化相同反应的酶都具有相同的EC编号。因此,EC编号是到目前为止最常用的酶分类方案,它使用四位数字来确定酶的催化功能,通过预测EC编号,可以用来完全确定酶的催化功能。针对现有模型无法对研究较少的蛋白质,或者具有未表征功能,或者多种活性的蛋白质的功能进行准确注释的问题,研究人员基于对比学习机制开发了一种酶蛋白功能预测的AI深度学习框架CLEAN。CLEAN 基于 UniProt 的高质量数据库进行训练,通过输入氨基酸序列就可得到酶功能编号(EC, Enzyme commission,指示酶催化哪种反应的 ID 代码)的可能性排序列表。为了验证 CLEAN 的准确性和稳健性,作者团队不仅进行了大量的计算机运算实验,还使用 CLEAN 为 36 个分类的卤化酶的进行功能注释 EC 编号,结合实际实验验证结果,综合表明 CLEAN 在这些酶功能预测任务中比其他工具更具优越性。
一、安装教程
1. 克隆项目代码
git clone https://github.com/tttianhao/CLEAN.git
cd CLEAN/app
2. 创建 Conda 环境
确保你已经安装了 Anaconda 或 Miniconda。
conda create -n clean python==3.10.4 -y
conda activate clean说明:这里我们新建了一个名为 clean 的虚拟环境,Python 版本为 3.10.4。
3. 安装依赖
进入项目目录后,使用 pip 安装所需依赖:
pip install -r requirements.txt4. 安装 PyTorch + CUDA 11.3
# CUDA 11.3
conda install pytorch==1.11.0 torchvision==0.12.0 torchaudio==0.11.0 cudatoolkit=11.3 -c pytorch
5. 下载预训练权重
-
下载并解压官方提供的预训练模型(70% / 100% split),以及 cluster embeddings 文件。
-
把解压后的内容放到:
data/pretrained/(没有下载这部分文件的话,推理会报错)
unzip pretrained.zip6. 构建依赖模块
python build.py install这是 CLEAN 的自定义构建步骤,必须执行。
6. 安装 Facebook ESM(蛋白质语言模型依赖)
git clone https://github.com/facebookresearch/esm.git然后把 esm/ 路径加到 PYTHONPATH,或者直接安装:
cd esm pip install -e .7. 准备 ESM 数据目录
mkdir data/esm_data二、使用教程
1. 执行示例推理脚本
python CLEAN_infer_fasta.py --fasta_data price执行上述脚本你的时候会默认下载esm的模型权重:esm1b_t33_650M_UR50S,但是由于网络等原因可能遇到下面的问题,解决方案就是下载到本地在传输到服务器上即可;
RuntimeError: unexpected EOF, expected ... The file might be corrupted.
显示以下的信息说明执行成功:
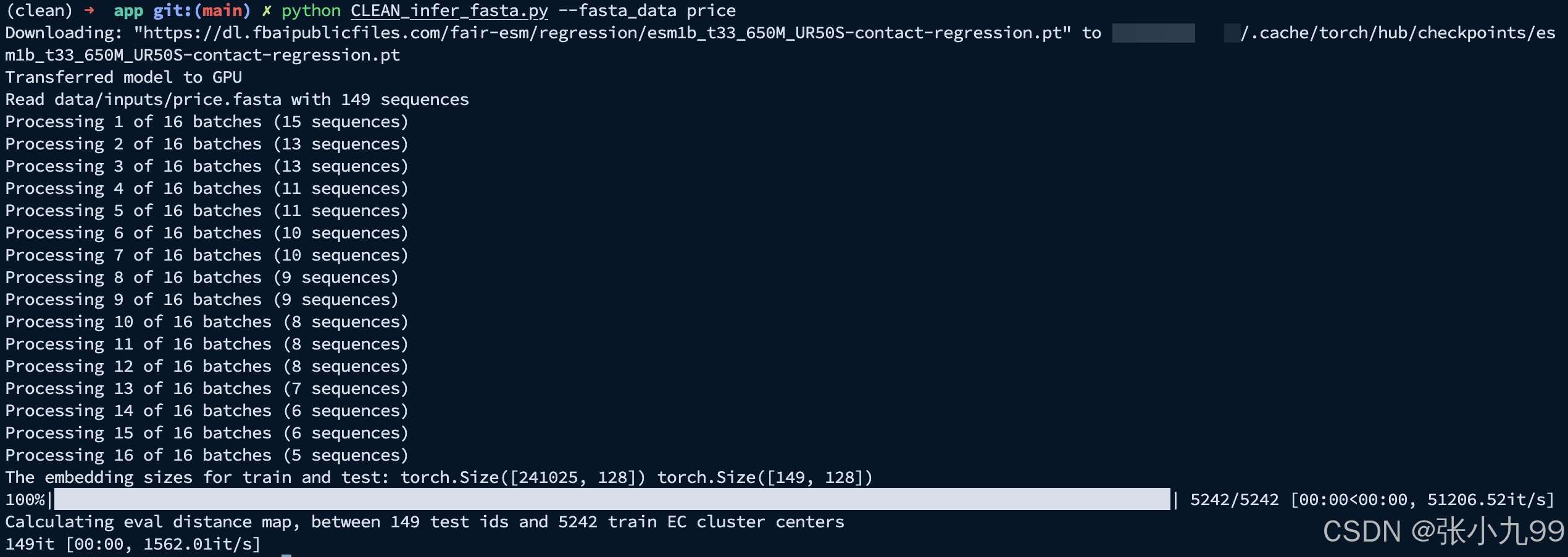
结果将保存在以下目录中:results/inputs/price_maxsep.csv
2. 通用推理脚本
python CLEAN_infer_fasta.py --fasta_data <name>-
只接收一个参数:
--fasta_data(字符串)。 -
语义:不带扩展名的 FASTA 基名。
示例:--fasta_data test→ 期望存在data/inputs/test.fasta。 -
推理结果:会保存在以下路径中
results/<name>.csv
脚本会自己去找 data/inputs/<name>.fasta,传入 test 而不是 test.fasta;否则最后清理时会去删 test.fasta.csv 这种不存在的路径
