帝国理工学院团队研发:Missense3D-PTMdb—— 解析遗传变异与翻译后修饰的交互式工具

前言概述
Missense3D-PTMdb 是一个交互式网络资源,旨在利用 AlphaFold 3D 模型探索和可视化遗传变异与翻译后修饰位点(PTMs),为研究人员提供蛋白质序列与结构的关联分析工具。该工具覆盖 20,235 种人类蛋白质、11,544,303 个天然错义变异及 203,775 个 PTM 位点,可自由访问(网址:https://missense3d.bc.ic.ac.uk/ptmdb)。
核心功能
![]()
![]()
序列 - 结构映射
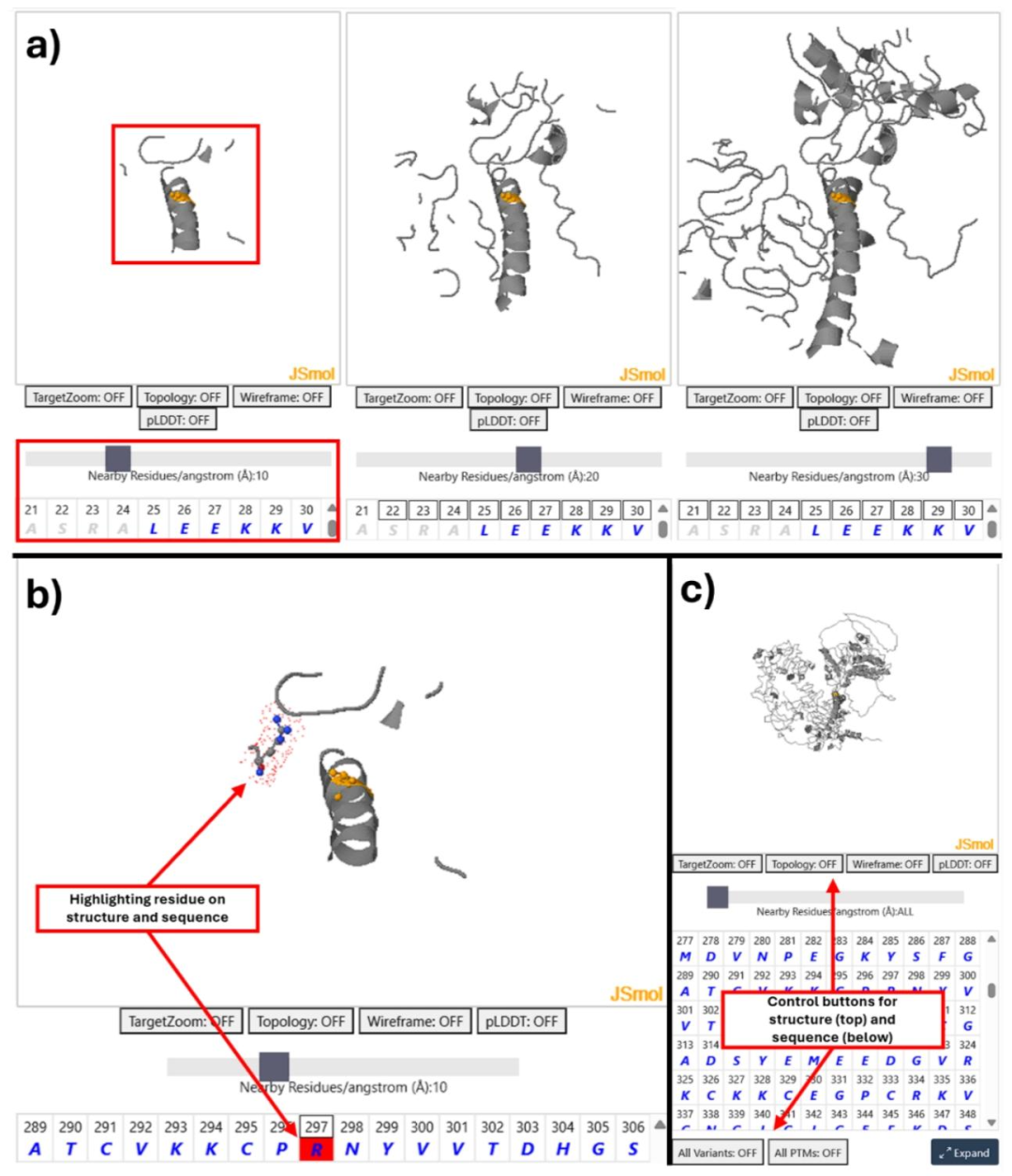
将错义变异和 PTM 位点映射到 AlphaFold 生成的蛋白质 3D 模型,支持查看变异与 PTM 位点在序列和空间上的邻近关系
![]()
可视化工具
通过 JSmol 实现 3D 结构交互可视化,用户可调整距离滑块(5Å 至 30Å)显示邻近残基,并同步高亮序列视图中的对应位置。
![]()
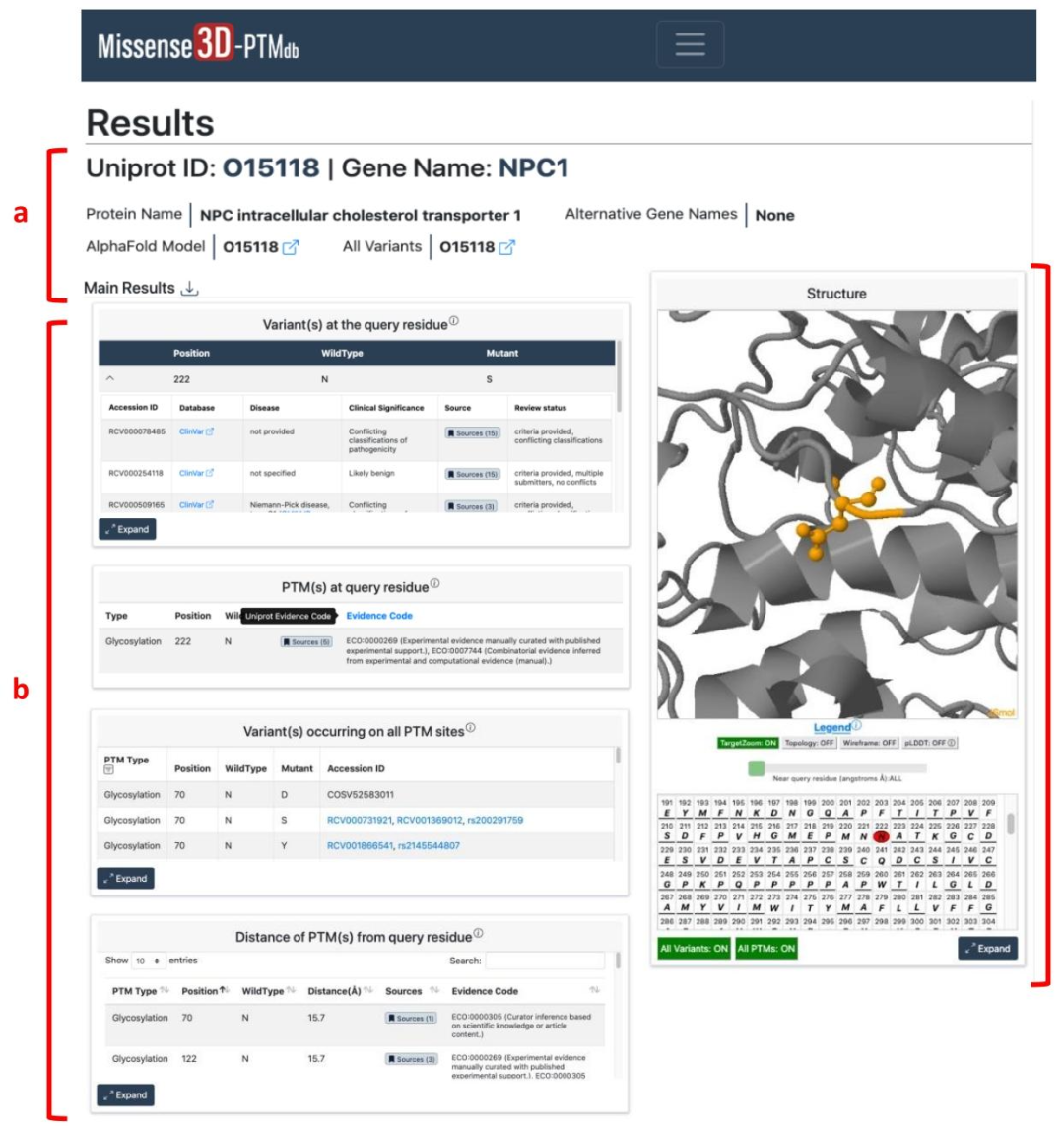
数据查询与下载
支持通过 UniProt ID、基因名、残基位置等检索,结果包含变异的临床意义、PTM 证据来源等,可导出为 Excel 文件。
数据亮点
PTM 类型分布
磷酸化位点占比最高(64%,130,269 条记录),其次为 SUMOylation(20%)、糖基化等,共涵盖 63 种 PTM 类型。
变异与 PTM 关联
1.7% 的错义变异(205,645 个)位于 PTM 残基上,其中 66% 涉及磷酸化位点;1.8% 的临床注释变异(10,510 个)位于 PTM 残基。
总结
该资源填补了 PTM 与变异 3D 结构关联分析的空白,为非结构生物学专家提供了直观工具,助力研究遗传变异对蛋白质功能的影响。
