机器学习——DBSCAN
一·概念介绍
有监督学习有外部结果y,无监督学习无外部结果y,此为二者核心区别。
概念:
基于密度的带噪声的空间聚类应用算法,它是将簇定义为密度相连的点的最大集合,能够把具有足够高密度的区域划分为簇,并在噪声的空间数据集中发现任意形状的聚类。

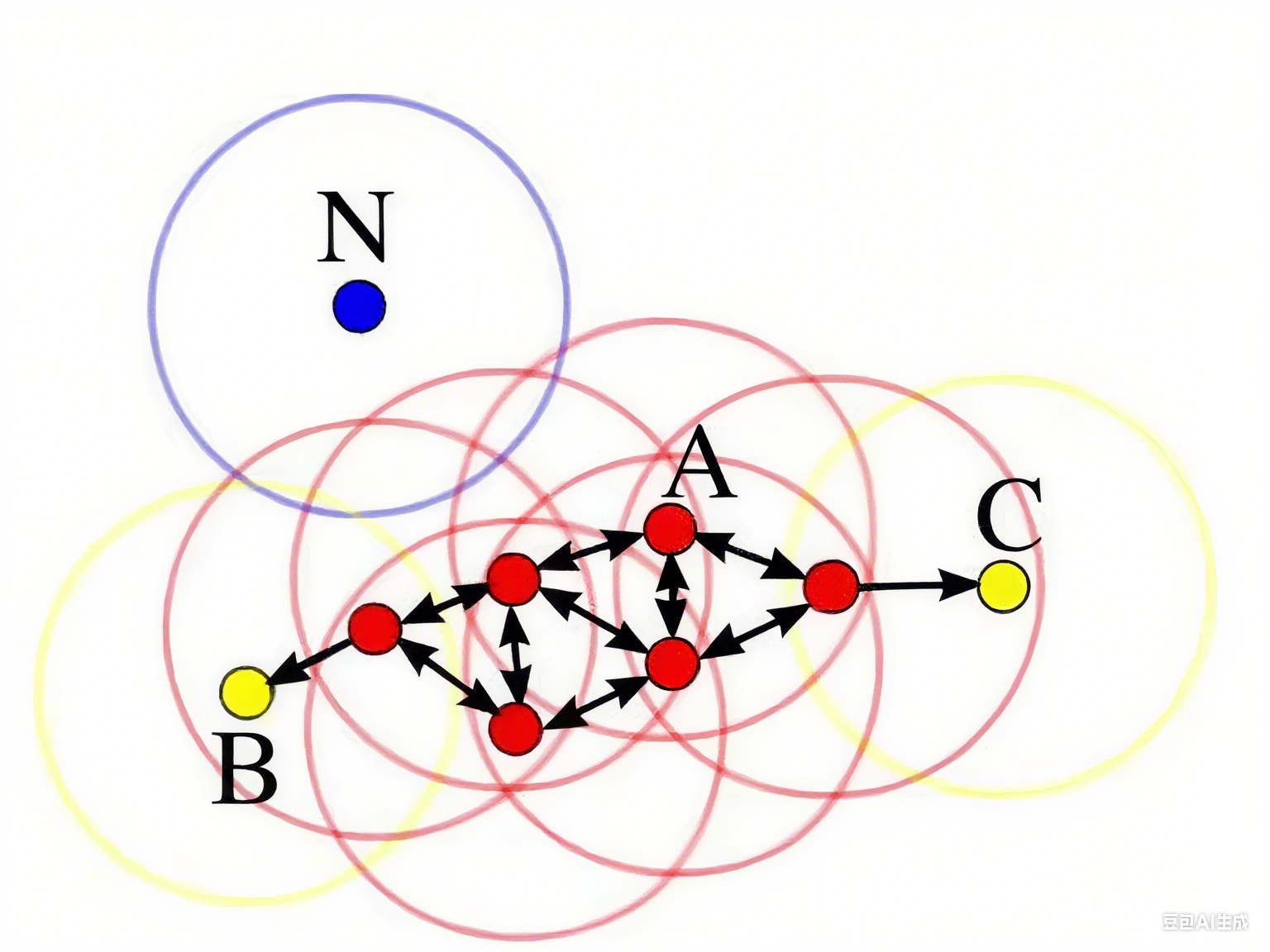
要点:
1. 核心对象:A 点
2.E 邻域:给定对象半径为 E 内的区域
3. 直接密度可达:
4. 密度可达:
5. 边界点:B 点、C 点
6. 离群点:N 点
实现过程
- 输入数据集
- 指定半径;
- 指定密度阈值;
二·参数
1. 初始化参数
python
class sklearn.cluster.DBSCAN(eps=0.5, min_samples=5, metric='euclidean', metric_params=None, algorithm='auto', leaf_size=30, p=None, n_jobs=None
)2. 各参数详细说明
eps:DBSCAN 算法中 ε- 邻域的距离阈值。样本距离超过eps的样本点不在 ε- 邻域内,默认值 0.5 ,需通过多组值选合适阈值。eps过大,更多点落入核心对象 ε- 邻域,类别数可能减少;过小则类别数可能增大,原本一类的样本会被分开。min_samples:样本点成为核心对象所需的 ε- 邻域样本数阈值,默认值 5 ,常和eps一起调参。min_samples过大,核心对象过少,簇内样本可能被标为噪音点,类别数变多;过小会产生大量核心对象,可能导致类别数过少。metric:最近邻距离度量参数,常用默认欧式距离(p=2的闵可夫斯基距离 )即可满足需求,可选值还有:- 欧式距离
“euclidean” - 曼哈顿距离
“manhattan” - 切比雪夫距离
“chebyshev”
- 欧式距离
metric_params:距离度量的额外参数,一般用默认值None即可。algorithm:最近邻搜索算法参数,有三种实现方式及四种可选输入:'brute':蛮力实现'kd_tree':KD 树实现'ball_tree':球树实现'auto':在上述三种算法中权衡选最优。若输入样本特征稀疏,最终会用'brute'。一般默认'auto'即可,数据量大 / 特征多且'auto'建树慢时,可按需调整。
leaf_size:构建 KD 树或球树时的叶节点大小,默认值 30 ,会影响树的构建速度和查询效率,一般用默认值。p:闵可夫斯基距离中p的值,p=1对应曼哈顿距离,p=2对应欧式距离,默认None(此时用默认欧式距离 )。n_jobs:用于并行计算的 CPU 核心数,None表示用 1 个核心,-1表示用所有可用核心,可加速最近邻搜索等过程。
3. 属性
labels_:训练后,存储每个样本点的分类标签,用于标识样本所属的簇(包括噪音点,噪音点标签通常为-1)。
三·代码
import pandas as pd
from sklearn.cluster import DBSCAN
from sklearn import metrics# 读取文件
beer = pd.read_table("data.txt", sep=' ', encoding='utf8', engine='python')
# 传入变量(列名)
X = beer[["calories", "sodium", "alcohol", "cost"]]
# DBSCAN聚类分析
"""
eps:半径
min_samples: 最小密度 【就是圆内最少有几个样本点】
labels: 分类结果 【自动分类,-1为离群点】
"""
db = DBSCAN(eps=20, min_samples=2).fit(X)#归一化,
labels = db.labels_# 添加结果至原数据框
"""
metrics.silhouette_score轮廓评价函数,它是聚类模型优劣的一种评估方式,可用于对聚类结果进行评
X:数据集 scaled_cluster: 聚类结果
score: 非标准化聚类结果的轮廓系数->聚类
"""score = metrics.silhouette_score(X, beer.cluster_db)
print(score)#作业: KNN的寝室。 y 暂时不用y,用kmens或dbscan来训练。 将一部分作为测试集,测试一下效果1. 导入所需库
首先导入了进行数据分析和聚类所需的库:
pandas:用于数据读取和处理DBSCAN:来自 scikit-learn 的密度聚类算法metrics:来自 scikit-learn,用于聚类结果评估
python
运行
import pandas as pd
from sklearn.cluster import DBSCAN
from sklearn import metrics
2. 读取数据文件
使用 pandas 的read_table函数读取数据文件:
- 数据文件名为 "data.txt"
- 使用空格作为分隔符(sep=' ')
- 指定编码为 utf8
- 使用 python 引擎解析文件
python
运行
# 读取文件
beer = pd.read_table("data.txt", sep=' ', encoding='utf8', engine='python')
3. 准备特征数据
从读取的数据中选取需要用于聚类分析的特征列:
- 选取了 "calories"(卡路里)、"sodium"(钠含量)、"alcohol"(酒精含量)和 "cost"(成本)这四个特征
- 这些特征数据将作为 DBSCAN 算法的输入
python
运行
# 传入变量(列名)
X = beer[["calories", "sodium", "alcohol", "cost"]]
4. 执行 DBSCAN 聚类
使用 DBSCAN 算法进行聚类分析:
eps=20:设置聚类的半径为 20min_samples=2:设置每个聚类的最小样本数为 2(即一个聚类至少需要包含 2 个样本)fit(X):对特征数据 X 执行聚类labels_:获取聚类结果标签,其中 - 1 表示离群点
python
运行
# DBSCAN聚类分析
"""
eps:半径
min_samples: 最小密度 【就是圆内最少有几个样本点】
labels: 分类结果 【自动分类,-1为离群点】
"""
db = DBSCAN(eps=20, min_samples=2).fit(X)#归一化,
labels = db.labels_
5. 评估聚类结果
使用轮廓系数(silhouette score)评估聚类效果:
- 轮廓系数取值范围为 [-1, 1]
- 接近 1 表示聚类效果好,样本与自身聚类中的样本更相似
- 接近 - 1 表示聚类效果差,样本更应该属于其他聚类
python
运行
# 计算轮廓系数评估聚类结果
"""
metrics.silhouette_score轮廓评价函数,它是聚类模型优劣的一种评估方式,可用于对聚类结果进行评
X:数据集 labels: 聚类结果
score: 聚类结果的轮廓系数
"""
score = metrics.silhouette_score(X, labels)
print(score)
注意事项
原代码中有一处笔误,beer.cluster_db应该改为labels,因为聚类结果存储在labels变量中,而不是beer数据框的cluster_db列中(该列尚未创建)。上面的代码已经修正了这一问题。
如果需要将聚类结果添加到原数据框中,可以添加以下代码:
python
运行
# 添加聚类结果到原数据框
beer['cluster_db'] = labels上面呢是轮廓系数,下面的是分类指标做的
总代码
import os
import pandas as pdfile_path = "datingTestSet2.txt"
if not os.path.exists(file_path):print(f"Error: File '{file_path}' not found")exit(1)try:data = pd.read_csv(file_path, sep=None, engine='python')
except Exception as e:print(f"File read error: {e}")exit(1)X = data.iloc[:, :-1]
y_true = data.iloc[:, -1].astype(int)import numpy as np
from scipy.optimize import linear_sum_assignmentdef align_labels(true_labels, cluster_labels):unique_true = np.unique(true_labels)unique_cluster = np.unique(cluster_labels)n_true = len(unique_true)n_cluster = len(unique_cluster)# 取较小的类别数作为矩阵维度,避免不匹配n = min(n_true, n_cluster)cost_matrix = np.zeros((n, n))for i in range(n):for j in range(n):cost_matrix[i, j] = -np.sum((true_labels == unique_true[i]) & (cluster_labels == unique_cluster[j]))row_ind, col_ind = linear_sum_assignment(cost_matrix)label_map = {}# 只映射存在的索引,避免越界for i, j in zip(row_ind, col_ind):if j < len(unique_cluster) and i < len(unique_true):label_map[unique_cluster[j]] = unique_true[i]aligned_labels = np.array([label_map.get(c, -1) for c in cluster_labels])return aligned_labelsfrom sklearn.cluster import KMeans
import matplotlib.pyplot as plt
from sklearn.metrics import accuracy_score, precision_score, recall_score, f1_scorek_values = [2, 3, 4, 5]
metrics_kmeans = {'accuracy': [], 'precision': [], 'recall': [], 'f1': []}for k in k_values:kmeans = KMeans(n_clusters=k, random_state=42).fit(X)aligned_labels = align_labels(y_true, kmeans.labels_)mask = aligned_labels != -1if np.sum(mask) > 0:acc = accuracy_score(y_true[mask], aligned_labels[mask])prec = precision_score(y_true[mask], aligned_labels[mask], average='macro', zero_division=0)rec = recall_score(y_true[mask], aligned_labels[mask], average='macro', zero_division=0)f1 = f1_score(y_true[mask], aligned_labels[mask], average='macro', zero_division=0)metrics_kmeans['accuracy'].append(acc)metrics_kmeans['precision'].append(prec)metrics_kmeans['recall'].append(rec)metrics_kmeans['f1'].append(f1)print(f"KMeans(k={k}) - Acc: {acc:.3f}, Prec: {prec:.3f}, Rec: {rec:.3f}, F1: {f1:.3f}")plt.figure(figsize=(12, 8))
for i, metric in enumerate(metrics_kmeans.keys()):plt.subplot(2, 2, i + 1)plt.plot(k_values, metrics_kmeans[metric], 'o-')plt.xlabel('k')plt.ylabel(metric)plt.title(f'KMeans {metric}')plt.tight_layout()
plt.show()from sklearn.cluster import DBSCANdb = DBSCAN(eps=0.5, min_samples=5).fit(X)
db_labels = db.labels_mask_noise = db_labels != -1
if np.sum(mask_noise) > 0:aligned_db_labels = align_labels(y_true[mask_noise], db_labels[mask_noise])acc_db = accuracy_score(y_true[mask_noise], aligned_db_labels)prec_db = precision_score(y_true[mask_noise], aligned_db_labels, average='macro', zero_division=0)rec_db = recall_score(y_true[mask_noise], aligned_db_labels, average='macro', zero_division=0)f1_db = f1_score(y_true[mask_noise], aligned_db_labels, average='macro', zero_division=0)print(f"\nDBSCAN - Acc: {acc_db:.3f}, Prec: {prec_db:.3f}, Rec: {rec_db:.3f}, F1: {f1_db:.3f}")
else:print("\nDBSCAN: Too many noise points, cannot calculate metrics")data['db_cluster'] = db_labels
print("\nSample results:")
print(data.head(10))
这段代码是上一个聚类分析代码的改进版本,核心区别在于评估指标的变化:上一个代码使用轮廓系数(聚类专用指标,适用于无真实标签的情况),而本代码因为有真实标签(y_true),所以采用了分类任务中常用的评估指标(准确率、精确率、召回率、F1 值)来更直观地衡量聚类效果与真实类别的匹配程度。
1. 导入基础库与文件处理
首先导入基础工具库,检查并读取数据文件:
os:用于检查文件是否存在pandas:用于数据读取和处理
python
运行
import os
import pandas as pdfile_path = "datingTestSet2.txt"
if not os.path.exists(file_path):print(f"Error: File '{file_path}' not found")exit(1)try:data = pd.read_csv(file_path, sep=None, engine='python') # 自动识别分隔符读取数据
except Exception as e:print(f"File read error: {e}")exit(1)
2. 准备特征数据与真实标签
从读取的数据中拆分出特征和真实标签(用于后续评估聚类效果):
X:所有行,除最后一列外的列(特征数据)y_true:所有行的最后一列(真实类别标签,转为整数类型)
python
运行
X = data.iloc[:, :-1] # 特征数据(所有特征列)
y_true = data.iloc[:, -1].astype(int) # 真实标签(最后一列)
3. 导入标签对齐所需库
聚类算法输出的标签仅代表类别编号(与真实标签的编号可能不一致),需要通过标签对齐将聚类标签映射到真实标签:
numpy:用于数值计算linear_sum_assignment:用于找到最优标签映射关系(匈牙利算法)
python
运行
import numpy as np
from scipy.optimize import linear_sum_assignment
4. 定义标签对齐函数
align_labels函数的作用是将聚类输出的标签(cluster_labels)与真实标签(true_labels)进行映射,解决 “类别编号不匹配” 问题:
- 构建成本矩阵:计算聚类标签与真实标签的匹配程度
- 用匈牙利算法找到最优映射关系
- 将聚类标签转换为与真实标签一致的编号(无法映射的标签记为 - 1)
python
运行
def align_labels(true_labels, cluster_labels):unique_true = np.unique(true_labels) # 真实标签的唯一值unique_cluster = np.unique(cluster_labels) # 聚类标签的唯一值n_true = len(unique_true)n_cluster = len(unique_cluster)# 取较小的类别数作为矩阵维度,避免不匹配n = min(n_true, n_cluster)cost_matrix = np.zeros((n, n))# 构建成本矩阵(值越小表示匹配度越高)for i in range(n):for j in range(n):# 计算聚类标签j与真实标签i的重叠数量(取负号转为成本)cost_matrix[i, j] = -np.sum((true_labels == unique_true[i]) & (cluster_labels == unique_cluster[j]))# 用匈牙利算法找到最优映射row_ind, col_ind = linear_sum_assignment(cost_matrix)label_map = {}# 构建映射关系(聚类标签→真实标签)for i, j in zip(row_ind, col_ind):if j < len(unique_cluster) and i < len(unique_true):label_map[unique_cluster[j]] = unique_true[i]# 将聚类标签转换为映射后的真实标签格式aligned_labels = np.array([label_map.get(c, -1) for c in cluster_labels])return aligned_labels
5. 导入 KMeans 聚类与评估库
导入 KMeans 算法、绘图工具和分类评估指标:
KMeans:聚类算法matplotlib.pyplot:用于绘制指标变化图- 分类评估指标:
accuracy_score(准确率)、precision_score(精确率)等
python
运行
from sklearn.cluster import KMeans
import matplotlib.pyplot as plt
from sklearn.metrics import accuracy_score, precision_score, recall_score, f1_score
6. 执行 KMeans 聚类与评估
尝试不同的聚类数(k=2,3,4,5),对每个k进行聚类并评估:
- 聚类后通过
align_labels对齐标签 - 过滤无法映射的标签(
-1),计算评估指标 - 存储指标结果并打印
python运行
k_values = [2, 3, 4, 5] # 尝试的聚类数
metrics_kmeans = {'accuracy': [], 'precision': [], 'recall': [], 'f1': []} # 存储各指标结果for k in k_values:# 训练KMeans模型kmeans = KMeans(n_clusters=k, random_state=42).fit(X)# 对齐聚类标签与真实标签aligned_labels = align_labels(y_true, kmeans.labels_)# 过滤无法映射的标签(仅保留有效标签)mask = aligned_labels != -1if np.sum(mask) > 0:# 计算评估指标acc = accuracy_score(y_true[mask], aligned_labels[mask])prec = precision_score(y_true[mask], aligned_labels[mask], average='macro', zero_division=0)rec = recall_score(y_true[mask], aligned_labels[mask], average='macro', zero_division=0)f1 = f1_score(y_true[mask], aligned_labels[mask], average='macro', zero_division=0)# 存储指标metrics_kmeans['accuracy'].append(acc)metrics_kmeans['precision'].append(prec)metrics_kmeans['recall'].append(rec)metrics_kmeans['f1'].append(f1)# 打印结果print(f"KMeans(k={k}) - Acc: {acc:.3f}, Prec: {prec:.3f}, Rec: {rec:.3f}, F1: {f1:.3f}")
7. 绘制 KMeans 指标变化图
用子图展示不同k值下各评估指标的变化趋势,直观对比聚类效果:
python运行
plt.figure(figsize=(12, 8))
for i, metric in enumerate(metrics_kmeans.keys()):plt.subplot(2, 2, i + 1) # 2行2列子图plt.plot(k_values, metrics_kmeans[metric], 'o-') # 绘制折线图plt.xlabel('k') # x轴:聚类数kplt.ylabel(metric) # y轴:评估指标plt.title(f'KMeans {metric}') # 子图标题plt.tight_layout() # 调整布局
plt.show() # 显示图像
8. 执行 DBSCAN 聚类与评估
使用 DBSCAN 算法进行聚类,同样通过标签对齐和分类指标评估:
- 先过滤噪声点(DBSCAN 中标签为
-1的样本) - 对有效样本的标签进行对齐,计算评估指标并打印
- python运行
from sklearn.cluster import DBSCAN# 训练DBSCAN模型(eps:半径,min_samples:最小样本数)
db = DBSCAN(eps=0.5, min_samples=5).fit(X)
db_labels = db.labels_ # 获取聚类标签(-1为噪声点)# 过滤噪声点
mask_noise = db_labels != -1
if np.sum(mask_noise) > 0:# 对齐有效样本的标签aligned_db_labels = align_labels(y_true[mask_noise], db_labels[mask_noise])# 计算评估指标acc_db = accuracy_score(y_true[mask_noise], aligned_db_labels)prec_db = precision_score(y_true[mask_noise], aligned_db_labels, average='macro', zero_division=0)rec_db = recall_score(y_true[mask_noise], aligned_db_labels, average='macro', zero_division=0)f1_db = f1_score(y_true[mask_noise], aligned_db_labels, average='macro', zero_division=0)# 打印结果print(f"\nDBSCAN - Acc: {acc_db:.3f}, Prec: {prec_db:.3f}, Rec: {rec_db:.3f}, F1: {f1_db:.3f}")
else:print("\nDBSCAN: Too many noise points, cannot calculate metrics")
9. 展示聚类结果样本
将 DBSCAN 的聚类结果添加到原始数据中,打印前 10 行样本,直观查看聚类标签:python
运行
data['db_cluster'] = db_labels # 添加DBSCAN聚类结果到数据框
print("\nSample results:")
print(data.head(10)) # 显示前10行数据及聚类标签
