地理空间视角下的 SIR 传染病模型模拟与可视化
在当今世界,传染病的传播模拟已成为公共卫生领域的重要研究工具。传统的传染病模型往往忽略了空间因素,将整个群体视为一个均质的整体。然而,现实中的疾病传播与地理空间位置密切相关,相邻区域之间的传播风险远高于远距离区域。今天,我们将通过一个结合地理信息系统 (GIS) 和随机模拟算法的案例,深入探讨如何在空间维度上模拟传染病的传播过程。
引言:当传染病模型遇上地理空间
传染病的传播从来都不是抽象的数字游戏,它实实在在地发生在我们生活的城市空间中。想象一下,在伦敦这样的大都市中,不同区域之间的人口流动、空间邻近性如何影响疾病的传播速度和范围?这正是我们今天要探索的主题。
我们将使用经典的 SIR 传染病模型,但会为其添加 "地理空间" 这一重要维度。SIR 模型将人群分为三类:易感者 (Susceptible)、感染者 (Infectious) 和康复者 (Recovered)。通过引入空间权重矩阵,我们可以量化不同区域之间的邻近关系,使模型能够更真实地反映疾病在地理空间上的传播过程。
数据准备:加载伦敦的地理空间数据
首先,我们需要准备地理空间数据。代码中加载了伦敦市 (City of London) 和卡姆登区 (Camden) 的低级统计区 (LSOA) 边界数据。这些数据是英国国家统计数据的一部分,能够帮助我们精确界定不同的地理单元。
# 加载伦敦市和卡姆登区的shapefile
lon = gpd.read_file("LSOA_2011_BFE_City_of_London.shp")
cam = gpd.read_file("LSOA_2011_BFE_Camden.shp")
# 合并两个GeoDataFrame
df = pd.concat([lon, cam], ignore_index=True)地理数据通常有不同的坐标参考系统 (CRS)。为了确保后续计算的准确性,我们需要进行坐标转换。我们先保留一个 WGS84 (经纬度) 坐标系的副本用于地图绘制,然后将数据转换为等距坐标系 (EPSG:3857) 用于质心计算和距离测量。
# 保留WGS84副本用于绘制经纬度地图
df_ll = df.to_crs(epsg=4326)# 在进行质心计算前重新投影到等距CRS(EPSG:3857)
df = df_ll.to_crs(epsg=3857)坐标系统的转换是地理空间分析中非常关键的一步。想象一下,如果我们在经纬度坐标系中直接计算两点之间的距离,由于地球曲率的影响,结果会有很大误差。而转换到等距坐标系后,我们就可以像在平面上一样准确计算距离和面积了 📐。

接下来,我们计算每个区域的质心,并获取它们在两种坐标系中的坐标:
# 计算两种CRS下的质心
centroids_3857 = df.geometry.centroid
coords_3857 = np.vstack([centroids_3857.x, centroids_3857.y]).T
centroids_ll = df_ll.geometry.centroid
coords_ll = np.vstack([centroids_ll.x, centroids_ll.y]).T空间权重矩阵:定义区域间的邻近关系
在空间分析中,"邻近性" 是一个关键概念。如何定义两个区域是否相邻?在代码中,我们使用了 "Queen contiguity" 规则,这是空间统计中常用的一种邻近性定义。Queen contiguity 认为,如果两个区域共享一条边或一个角,则它们是相邻的。
# 使用Queen邻接构建空间权重
w = libpysal.weights.Queen.from_dataframe(df, use_index=True)# 转换为权重矩阵
n = len(df)
W = np.zeros((n, n), dtype=int)
for i, neighbors in w.neighbors.items():for j in neighbors:W[i, j] = 1W[j, i] = 1 # 对称矩阵这种邻接关系定义很有意思,它模拟了一种 "棋盘式" 的邻近性。想象一下国际象棋中的皇后可以沿对角线移动,Queen contiguity 就是用这种方式定义邻近性的 � chess。与只考虑共享边的 "Rook contiguity" 相比,它考虑了更多的邻近关系。
为了更直观地理解我们构建的权重矩阵,我们可以绘制权重分布的直方图:
# 正权重的直方图
dist = W[W > 0]
plt.figure()
plt.hist(dist, bins=20)
plt.title('Distribution of Weights > 0')
plt.show()由于我们使用的是二元邻接矩阵 (相邻为 1,不相邻为 0),这个直方图应该会呈现出明显的双峰分布,其中大部分权重为 0,而相邻区域的权重为 1。不过,在后续步骤中,我们还会引入阈值处理,让邻接关系变得更加 "模糊"。
构建空间网络:阈值化邻接关系
有时候,我们可能需要考虑更灵活的邻近关系定义。例如,不仅考虑直接相邻的区域,还可以根据距离或其他因素定义一个 "影响范围"。在代码中,我们通过设置一个阈值,将权重矩阵转换为一个二元邻接矩阵:
# 阈值邻接
threshold = 0.16
adj = (W > threshold).astype(int)这里的阈值设置是一个关键参数,它决定了哪些区域会被视为 "相邻"。阈值越高,邻接关系越严格,网络中的连接越少;阈值越低,邻接关系越宽松,网络中的连接越多。这个参数的设置需要根据具体问题和数据特点来调整 🧩。
我们可以将这些邻接关系可视化,看看在地理空间上区域之间是如何连接的:
# 绘制高于阈值的边
g_df = df_ll.copy()
plt.figure(figsize=(6, 6))
g_df.plot(facecolor='none', edgecolor='gray')
plt.scatter(coords_ll[:, 0], coords_ll[:, 1], s=10, color='black')
for i in range(n):for j in range(i + 1, n):if adj[i, j]:plt.plot([coords_ll[i, 0], coords_ll[j, 0]],[coords_ll[i, 1], coords_ll[j, 1]],color='blue', linewidth=0.5)
plt.title('Network Edges (thresholded)')
plt.show()
看着这些蓝色的连接线在地图上延伸,我们仿佛看到了疾病可能传播的 "高速公路"。这些连接线构成了一个空间网络,疾病将在这个网络上进行传播 🌐。
SIR 模型:从经典到空间扩展
现在,我们来介绍 SIR 模型的基本原理。SIR 模型是一个经典的传染病模型,它将人群分为三个状态:
- S (Susceptible): 易感者,尚未感染但可能被感染的人
- I (Infectious): 感染者,具有传染性的人
- R (Recovered): 康复者,已经康复并获得免疫力的人
模型的核心是两个速率参数:
- β(beta): 感染率,表示易感者与感染者接触后被感染的概率
- γ(gamma): 康复率,表示感染者康复的概率
在传统的 SIR 模型中,通常假设人群是完全混合的,即每个人与其他人接触的概率是相等的。但在我们的空间扩展版本中,这个假设被打破了。我们考虑了空间邻近性,即一个区域的感染状态会受到其相邻区域感染状态的影响。
在代码中,我们设置了感染率和康复率参数:
# SIR参数
beta = 3.0 # 感染率
gamma = 1.0 # 康复率这些参数的值会显著影响疾病传播的动态。例如,较高的感染率会导致疾病更快地传播,而较高的康复率会缩短感染期,减缓疾病的传播速度 ⚙️。
初始状态设置:疫情的起点
每个传染病模拟都需要一个起点。在代码中,我们随机选择一个区域作为初始感染区域:
# 初始化状态: 'S', 'I', 'R'
status = np.array(['S'] * n)
i0 = random.randrange(n)
status[i0] = 'I'这个初始感染点的选择虽然是随机的,但在现实中,疫情的起点可能与人口密度、交通枢纽等因素有关。如果我们有更详细的数据,我们可以根据实际情况选择更合理的初始感染点 🦠。
Gillespie 随机模拟算法:捕捉传染病的随机性
传染病的传播本质上是一个随机过程,因为每个接触事件是否导致感染都是概率性的。为了准确捕捉这种随机性,我们使用了 Gillespie 随机模拟算法,这是一种在分子尺度上模拟随机过程的强大方法。
Gillespie 算法的核心思想是:
- 计算系统中所有可能事件的发生速率
- 根据这些速率计算每个事件发生的概率
- 随机选择一个事件发生
- 计算到下一个事件发生的时间间隔
- 重复上述过程
在我们的 SIR 模型中,有两种可能的事件:
- 感染事件:易感者被感染者感染
- 康复事件:感染者康复
让我们看看代码中是如何实现这个算法的:
# 存储模拟结果
T_max = 10.0 # 最大模拟时间
times = [0.0] # 时间点列表
states = [status.copy()] # 每个时间点的状态列表
adj_list = {i: list(np.where(adj[i] == 1)[0]) for i in range(n)} # 邻接列表
current_time = 0.0 # 当前时间# Gillespie模拟
i = 0
while current_time < T_max:# 找出所有感染者I_indices = np.where(status == 'I')[0]# 计算每个感染者的康复速率rec_rates = {idx: gamma for idx in I_indices}# 找出所有易感者S_indices = np.where(status == 'S')[0]# 计算每个易感者的感染速率,与相邻感染者数量有关inf_rates = {idx: beta * sum(status[j] == 'I' for j in adj_list[idx]) for idx in S_indices}# 合并所有事件及其速率all_rates = {**rec_rates, **inf_rates}total_rate = sum(all_rates.values())# 如果没有事件发生,结束模拟if total_rate <= 0:break# 提取事件节点和速率nodes, rates = zip(*all_rates.items())# 计算每个事件发生的概率probs = np.array(rates) / total_rate# 随机选择一个事件event_node = np.random.choice(nodes, p=probs)# 更新状态if status[event_node] == 'S':status[event_node] = 'I' # 易感者被感染else:status[event_node] = 'R' # 感染者康复# 计算到下一个事件的时间间隔dt = np.random.exponential(1.0 / total_rate)current_time += dt# 存储当前时间和状态times.append(current_time)states.append(status.copy())i += 1Gillespie 算法的一个重要特点是它精确地模拟了随机过程的时间演化,而不是像欧拉法那样使用固定的时间步长。这使得它在处理具有不同时间尺度的事件时特别有效 ⏳。
在我们的实现中,每个易感者的感染速率不仅取决于感染率 β,还取决于其相邻区域中有多少个感染区域。这正是空间因素被引入模型的关键所在 🌍。
结果分析:传染病的时间动态
模拟结束后,我们可以分析传染病在时间上的传播动态。首先,我们创建一个时间序列数据框,记录每个时间点上易感者、感染者和康复者的数量:
# 时间序列DataFrame
df_counts = [{'time': t,'S': int((st == 'S').sum()),'I': int((st == 'I').sum()),'R': int((st == 'R').sum())}for t, st in zip(times, states)]
df_ts = pd.DataFrame(df_counts)然后,我们可以绘制这三类人群随时间变化的曲线:
plt.figure()
for compartment in ['S', 'I', 'R']:plt.plot(df_ts['time'], df_ts[compartment], label=compartment)
plt.xlabel('Time')
plt.ylabel('Count')
plt.legend()
plt.title('SIR Dynamics Over Time')
plt.show()
典型的 SIR 模型时间曲线应该呈现出这样的模式:
- 易感者数量 (S) 随时间单调递减
- 感染者数量 (I) 先增加后减少,形成一个峰值
- 康复者数量 (R) 随时间单调递增
看着这些曲线的变化,我们可以直观地理解传染病的传播过程:从少数人感染开始,逐渐扩散到高峰,然后随着越来越多的人康复,感染人数逐渐减少 📈📉。
空间传播动画:疾病在地理空间上的扩散
最令人兴奋的部分莫过于将疾病的空间传播过程可视化。通过创建动画,我们可以直观地看到疾病如何从初始感染点开始,沿着空间网络向周围区域扩散。
首先,我们准备动画所需的数据:
# 准备DataFrame用于动画
records = []
for step, st in enumerate(states):for idx, s in enumerate(st):records.append({'time_step': step,'node': idx,'status': s,'x': coords_ll[idx, 0],'y': coords_ll[idx, 1]})
anim_df = pd.DataFrame(records)然后,我们实现动画生成函数:
# 使用PillowWriter生成动画
def animate_sir_map(output_file='london.gif', fps=5):fig, ax = plt.subplots(figsize=(6, 6))def update(frame):ax.clear()df_ll.plot(ax=ax, facecolor='lightgray', edgecolor='white')df_step = anim_df[anim_df['time_step'] == frame]for label, color in zip(['S', 'I', 'R'], ['blue', 'red', 'green']):sub = df_step[df_step['status'] == label]ax.scatter(sub['x'], sub['y'], label=label, s=20)ax.set_title(f"Time step: {frame}")ax.legend(loc='upper right', fontsize='small')ax.set_axis_off()writer = PillowWriter(fps=fps)anim = FuncAnimation(fig, update, frames=len(states), interval=200)anim.save(output_file, writer=writer)print(f"Animation saved as {output_file} using PillowWriter")# 生成动画
animate_sir_map()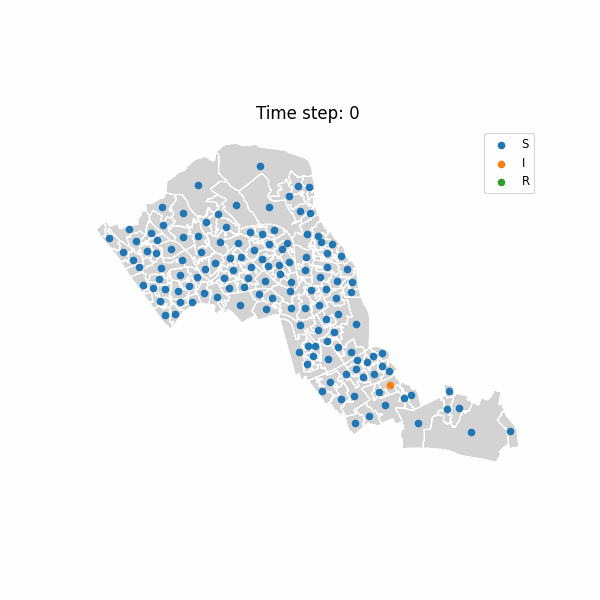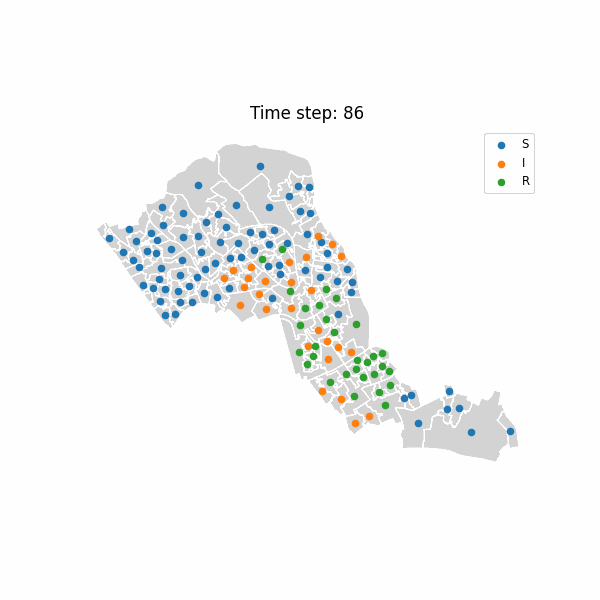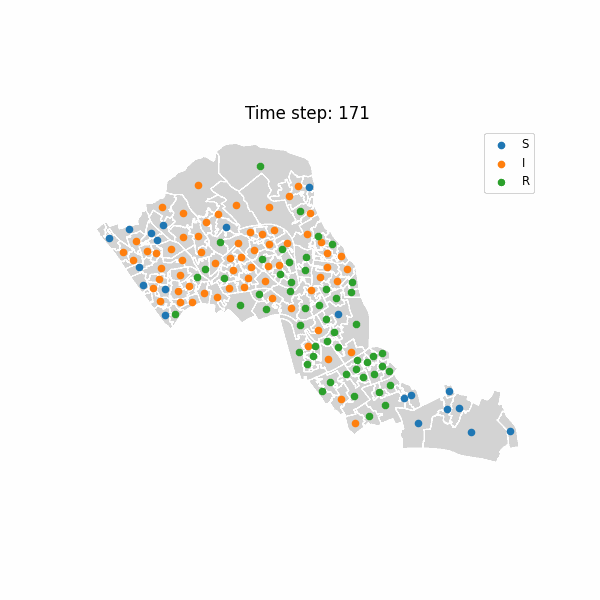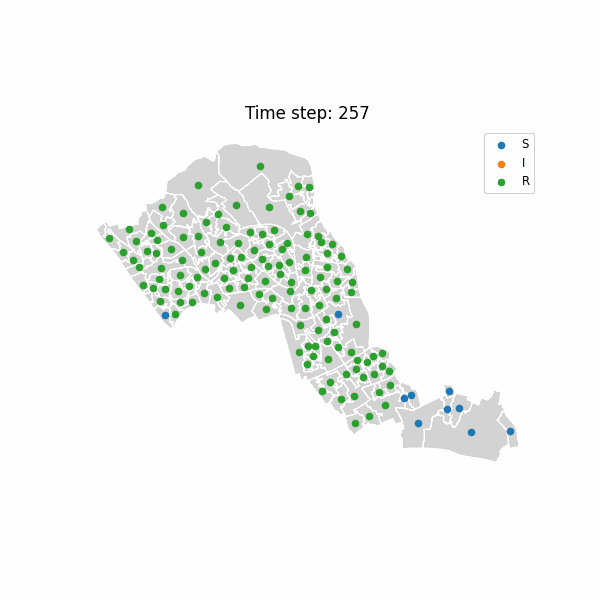
当我们播放这个动画时,可以看到红色的感染区域如何从一个点开始,逐渐扩散到周围的蓝色易感区域,然后变成绿色的康复区域。这种时空动态直观地展示了空间邻近性如何影响疾病的传播模式 🌍→🦠→💊。
模型的局限性与改进方向
尽管我们的空间 SIR 模型比传统模型更贴近现实,但它仍然有一些局限性:
-
空间权重的简化:我们使用了简单的二元邻接矩阵来表示空间关系,而现实中的疾病传播可能受到多种因素影响,如人口流动、交通网络等。
-
同质性假设:模型假设每个区域的人口特征是相同的,而实际上不同区域的人口密度、年龄结构等可能差异很大。
-
确定性参数:我们使用了固定的感染率和康复率,而在现实中这些参数可能随时间和空间变化。
针对这些局限性,我们可以考虑以下改进方向:
-
更复杂的空间权重:可以基于实际交通数据或人口流动数据构建更真实的空间权重矩阵。
-
异质性建模:可以为不同区域设置不同的人口特征和疾病传播参数。
-
引入行为干预:可以模拟社交距离、封锁等干预措施对疾病传播的影响 🛡️。
-
考虑人口流动:可以引入更复杂的人口流动模型,模拟通勤等日常活动对疾病传播的影响。
结语:空间视角下的传染病防控
通过这个案例,我们看到了地理空间视角在传染病建模中的重要性。空间邻近性、区域连接性等因素显著影响着疾病的传播模式和速度。在公共卫生政策制定中,考虑这些空间因素可以帮助我们更精准地分配资源、实施干预措施。
想象一下,当我们能够在地图上实时看到疾病的传播路径和高风险区域时,公共卫生部门就可以更有针对性地部署医疗资源、实施隔离措施,从而最大限度地减少疾病的影响 🏥。
这个模型不仅是一个学术研究工具,也为我们理解现实世界中的传染病传播提供了一个窗口。随着地理信息技术和计算能力的不断发展,我们有理由相信,未来的传染病模型将更加精确、更加贴近现实,为全球公共卫生安全提供更强有力的支持 🌟。
如果你对这个模型感兴趣,不妨尝试调整参数看看会发生什么变化:增加或减少感染率 β,看看疾病传播速度如何变化;调整空间权重的阈值,看看空间网络结构如何影响传播模式。探索不同场景下的疾病传播动态,也许你会有新的发现哦! 🧪
完整代码(省流版)
import random
import numpy as np
import pandas as pd
import geopandas as gpd
import libpysal
import matplotlib.pyplot as plt
from matplotlib.animation import FuncAnimation, PillowWriter# Reproducibility
random.seed(33333334)
np.random.seed(33333334)# Load shapefiles for City of London and Camden
lon = gpd.read_file("LSOA_2011_BFE_City_of_London.shp")
cam = gpd.read_file("LSOA_2011_BFE_Camden.shp")
# Combine GeoDataFrames
df = pd.concat([lon, cam], ignore_index=True)# Keep a WGS84 copy for plotting maps in lon/lat
df_ll = df.to_crs(epsg=4326)# Re-project to a metric CRS before centroid calculations (EPSG:3857)
df = df_ll.to_crs(epsg=3857)# Compute centroids in both CRS
centroids_3857 = df.geometry.centroid
coords_3857 = np.vstack([centroids_3857.x, centroids_3857.y]).T
centroids_ll = df_ll.geometry.centroid
coords_ll = np.vstack([centroids_ll.x, centroids_ll.y]).T# Build spatial weights using Queen contiguity
w = libpysal.weights.Queen.from_dataframe(df, use_index=True)# Convert to weight matrix
n = len(df)
W = np.zeros((n, n), dtype=int)
for i, neighbors in w.neighbors.items():for j in neighbors:W[i, j] = 1W[j, i] = 1 # symmetric# Plot base map and centroids
plt.figure(figsize=(6, 6))
df_ll.plot(facecolor='none', edgecolor='black')
plt.scatter(coords_ll[:, 0], coords_ll[:, 1], s=10)
plt.title('Polygons and Centroids (WGS84)')
plt.show()# Histogram of positive weights
dist = W[W > 0]
plt.figure()
plt.hist(dist, bins=20)
plt.title('Distribution of Weights > 0')
plt.show()# Threshold adjacency
threshold = 0.16
adj = (W > threshold).astype(int)# Plot edges above threshold
g_df = df_ll.copy()
plt.figure(figsize=(6, 6))
g_df.plot(facecolor='none', edgecolor='gray')
plt.scatter(coords_ll[:, 0], coords_ll[:, 1], s=10, color='black')
for i in range(n):for j in range(i + 1, n):if adj[i, j]:plt.plot([coords_ll[i, 0], coords_ll[j, 0]],[coords_ll[i, 1], coords_ll[j, 1]],color='blue', linewidth=0.5)
plt.title('Network Edges (thresholded)')
plt.show()# SIR parameters
beta = 3.0
gamma = 1.0# Initialize status: 'S', 'I', 'R'
status = np.array(['S'] * n)
i0 = random.randrange(n)
status[i0] = 'I'# Storage
T_max = 10.0
times = [0.0]
states = [status.copy()]
adj_list = {i: list(np.where(adj[i] == 1)[0]) for i in range(n)}
current_time = 0.0# Gillespie simulation
i = 0
while current_time < T_max:I_indices = np.where(status == 'I')[0]rec_rates = {idx: gamma for idx in I_indices}S_indices = np.where(status == 'S')[0]inf_rates = {idx: beta * sum(status[j] == 'I' for j in adj_list[idx]) for idx in S_indices}all_rates = {**rec_rates, **inf_rates}total_rate = sum(all_rates.values())if total_rate <= 0:breaknodes, rates = zip(*all_rates.items())probs = np.array(rates) / total_rateevent_node = np.random.choice(nodes, p=probs)if status[event_node] == 'S':status[event_node] = 'I'else:status[event_node] = 'R'dt = np.random.exponential(1.0 / total_rate)current_time += dttimes.append(current_time)states.append(status.copy())i += 1# Time series DataFrame
df_counts = [{'time': t,'S': int((st == 'S').sum()),'I': int((st == 'I').sum()),'R': int((st == 'R').sum())}for t, st in zip(times, states)]
df_ts = pd.DataFrame(df_counts)plt.figure()
for compartment in ['S', 'I', 'R']:plt.plot(df_ts['time'], df_ts[compartment], label=compartment)
plt.xlabel('Time')
plt.ylabel('Count')
plt.legend()
plt.title('SIR Dynamics Over Time')
plt.show()# Prepare DataFrame for animation
records = []
for step, st in enumerate(states):for idx, s in enumerate(st):records.append({'time_step': step,'node': idx,'status': s,'x': coords_ll[idx, 0],'y': coords_ll[idx, 1]})
anim_df = pd.DataFrame(records)# Animate with PillowWriter
def animate_sir_map(output_file='london.gif', fps=5):fig, ax = plt.subplots(figsize=(6, 6))def update(frame):ax.clear()df_ll.plot(ax=ax, facecolor='lightgray', edgecolor='white')df_step = anim_df[anim_df['time_step'] == frame]for label, color in zip(['S', 'I', 'R'], ['blue', 'red', 'green']):sub = df_step[df_step['status'] == label]ax.scatter(sub['x'], sub['y'], label=label, s=20)ax.set_title(f"Time step: {frame}")ax.legend(loc='upper right', fontsize='small')ax.set_axis_off()writer = PillowWriter(fps=fps)anim = FuncAnimation(fig, update, frames=len(states), interval=200)anim.save(output_file, writer=writer)print(f"Animation saved as {output_file} using PillowWriter")animate_sir_map()
