如何获得更多高质量MAG?
上一期内容凌恩生物给大家介绍了2两款子刊推荐的宏基因组组装软件(25年了,宏基因组组装还在用metawrap?),有些朋友觉得意犹未尽,那今天我们接着探究在繁多的宏基因组组装软件中,谁才是最优解?
背景知识
CAMI是一个国际性的研究计划,冒在通过提供标准化的真实和模拟宏基因组数据集,推动宏基因组数据分析工具(如物种注释、功能预测、组装、分箱等)的透明评估和性能比较,从而促进工具开发者改进方法。CAMI数据集包括来自不同环境(如海洋、根系、临床环境等)的复杂宏基因组数据集,这些数据集包括长读长和短读长的数据。
本研究利用CAMI2的海洋、株菌级别、植物相关的数据集对最常用到的宏基因组组装软件——McDevol、VAMB、COMBin、GenomeFace、MetaBAT2、CONCOCT、MaxBin2、MetaWRAP进行全面评估。
研究结果
(1)多种组装软件联合使用是个好办法!
MetaWRAP作为一个集成化工具(包括MetaBAT2, MaxBin2和CONCOCT),获得bin的数量最多,说明多个组装软件联合使用可以提高获得bins的数量。
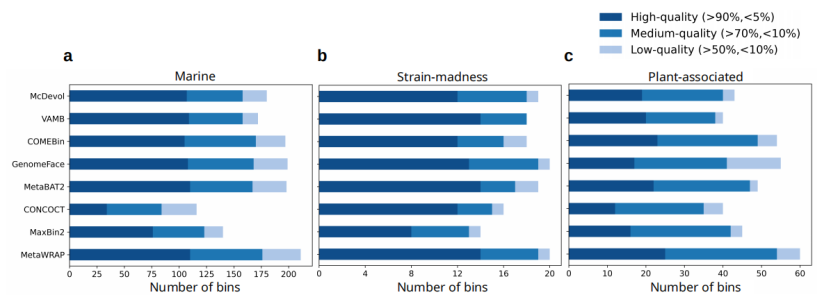
图1 8种工具在不同数据集中组装获得的bins数量展示
(2)重新组装是否可以提升组装质量?
使用CheckM2 评估重新组装对来自共组装多样本分类的基因组分类的影响。发现在海洋和植物相关数据集中,重新组装可以小范围提高组装获得高bins的数量,对低丰度bins的完整性和纯度有轻微改善。说明重新组装对提升组装质量作用不大。
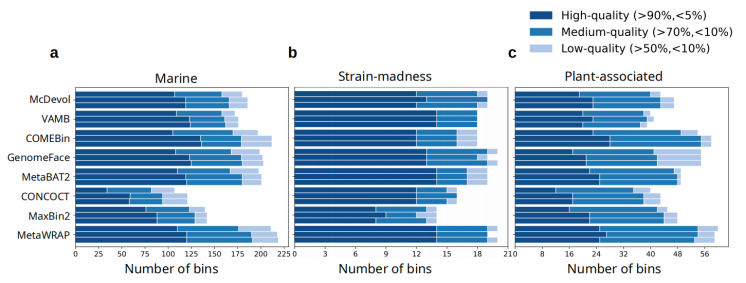
图2 重新组装组装获得的bins数量展示
(3)组装策略怎么选?
宏基因组组装策略共有三种:① Coassembly multi-sample(混合样本组装和分箱);② Multi-sample(样本组装和多样本分箱);③ Single-sample(单样本组装和分箱)。对McDevol、VAMB、COMEBin、GenomeFace和MetaBAT2进行了这三种方法的分箱性能比较,发现不同样本类型,组装策略的选择存在差异,组装结果如下图b、c、d所示,以上组装软件及策略凌恩生物都可正常开展哦,有需要随时联系 ~
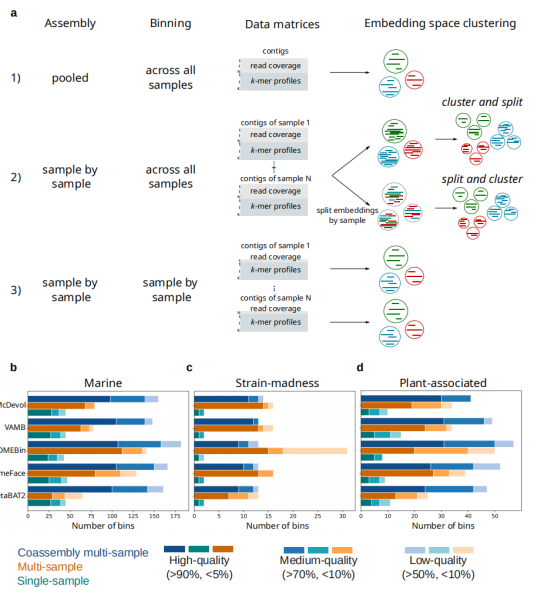
图3 不同的组装策略、软件组装效果展示
(4)运算速率及峰值内存评估结果
GenomeFace对三种数据集的运算速率比其他工具快快,但GenomeFace 的峰值内存使用量也最高。
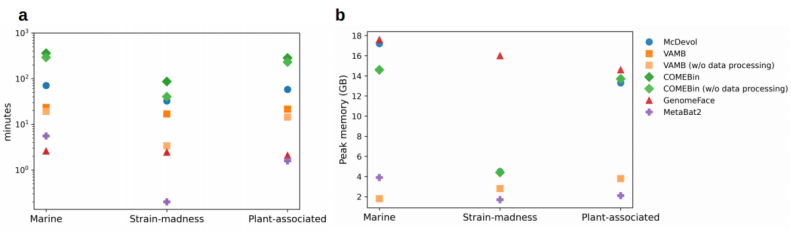
图4 运算速率
思考与总结
本研究利用CAMI2的海洋、株菌级别、植物相关的数据集,通过组装获得bins的数量、不同的组装策略、运算速率等维度对最常用到的宏基因组组装软件进行全面评估。总体而言,COMEBin 和 GenomeFace 表现最为出色,其中 MetaBAT2 和 GenomeFace 在速度方面更胜一筹。凌恩生物深耕微生态研究,为客户提供最新的组装软件满足不同的科研需求,以上6款软件、3种不同组装策略,凌恩 都可正常开展哦,期待您的咨xun。
参考文献
Evaluation of Metagenome Binning: Advances and Challenges
