AI-人工智能-多模态学习助力精准预测心脏毒性
多模态学习助力精准预测心脏毒性

目录
- 新型蛇床素衍生物对耐甲氧西林金黄色葡萄球菌 (MRSA) 展现出强大的抗菌活性,有望成为新型抗生素。
- Yuel 2 利用迁移学习提高了蛋白质 - 小分子结合亲和力预测的准确性和鲁棒性。
- MultiCTox 模型通过整合分子多模态数据,显著提高了心脏毒性预测的准确性。
- 利用可解释机器学习识别预测黑色素瘤免疫疗法疗效的关键生物标志物及阈值,并验证其临床意义。
- UniMoMo 统一生成模型,高效设计多肽、抗体和小分子结合剂,显著提升药物研发效率。
1. 抗 MRSA 新药:蛇床素衍生物展现强大潜力
耐甲氧西林金黄色葡萄球菌 (MRSA) 感染日益严重,亟需开发新型抗菌药物。本研究设计并合成了 30 种带有吡啶季铵基团的蛇床素衍生物。体外试验表明,化合物 8u 和 8ac 对金黄色葡萄球菌 ATCC 29213 和十株临床 MRSA 分离株表现出强大的抗菌活性(MIC = 0.5-1 μg/mL),同时具有低溶血活性、快速杀菌作用和最小耐药性诱导。
在 MRSA 感染的小鼠皮肤脓肿和败血症模型中,8u 和 8ac 也显示出优异的抗菌效果和安全性,与万古霉素相当。机制研究表明,8u 和 8ac 通过与磷脂酰甘油 (PG) 结合选择性靶向细菌细胞膜,增加细胞内活性氧 (ROS),诱导内容物泄漏,最终导致细菌死亡。因此,8u 和 8ac 有望成为抗 MRSA 药物研发的 promising 候选药物。
📜Paper: DOI: 10.1021/acs.jmedchem.4c03167[1]
2. 迁移学习助力蛋白质 - 小分子相互作用预测
生物分子间的相互作用,如蛋白质与小分子间的结合,构成了复杂的网络,并对生物过程的调控起着关键作用。理解这一网络对于深入研究生命活动至关重要。然而,由于生物系统中参与者的数量庞大(例如,人体细胞中约有 10⁴ 种蛋白质,潜在的相互作用数量可达 10⁸ 种),解析这些相互作用一直是一项挑战。此外,相互作用的相对强度也显著影响着生物过程。
传统上用于结合亲和力预测的数据集规模较小,通常只包含 10³ 到 10⁴ 个蛋白质 - 配体相互作用数据,这限制了模型捕捉这些相互作用全部复杂性的能力。为了克服这一挑战,研究者开发了 Yuel 2,这是一种基于神经网络的新方法,它利用迁移学习来解决小数据集的局限性。
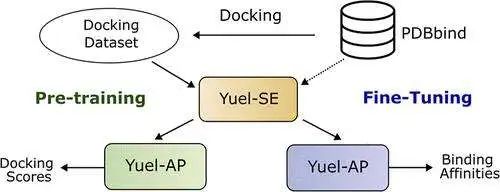
Yuel 2 首先在大规模数据集上进行预训练,学习复杂的结构特征,然后在特定数据集(如 PDBbind)上进行微调,以提高预测的准确性和鲁棒性。研究表明,Yuel 2 可以预测蛋白质与小分子之间的多种结合亲和力指标,包括 Kd、Ki 和 IC50,从而为药物设计和开发提供了对分子相互作用的全面理解。
📜Paper: https://doi.org/10.1021/acs.jcim.4c02256
3. 多模态学习助力精准预测心脏毒性
心脏毒性是指药物对心脏离子通道的抑制作用,准确预测心脏毒性对于评估心脏药物的疗效和安全性至关重要。然而,由于其复杂性,心脏毒性预测一直是一项挑战。传统的预测方法通常依赖于单一模态数据,例如仅使用分子结构或指纹信息,从而限制了预测的准确性。为了解决这一问题,研究者开发了 MultiCTox 模型,一种基于自适应多模态学习的心脏毒性预测方法。
MultiCTox 模型的关键创新在于其整合了多种分子信息,包括 SMILES 字符串、分子结构和指纹。通过设计一个融合层,模型能够将不同模态的表示统一起来。在训练过程中,模型最大化同一分子的模态内相似性,同时最小化不同分子间的相似性,确保跨模态表示的一致性。这种方法有效地利用了多模态数据的互补性,从而提高了预测的准确性。
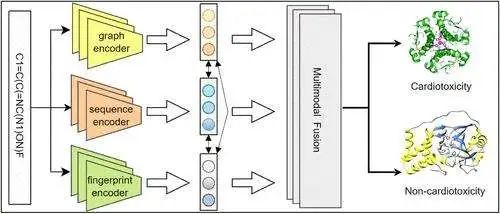
研究者使用该模型评估了候选药物对三种关键心脏离子通道(电压门控钾通道 hERG、钠通道 Nav1.5 和钙通道 Cav1.2)的抑制作用。实验结果表明,MultiCTox 模型在预测这些通道的药物抑制作用方面显著优于现有的先进方法。因此,作者认为 MultiCTox 模型将为心脏药物的开发和安全评估做出重要贡献,并降低与心脏毒性相关的风险。
📜Paper: https://doi.org/10.1021/acs.jcim.5c00022
4. 机器学习预测免疫疗法响应的生物标志物和阈值
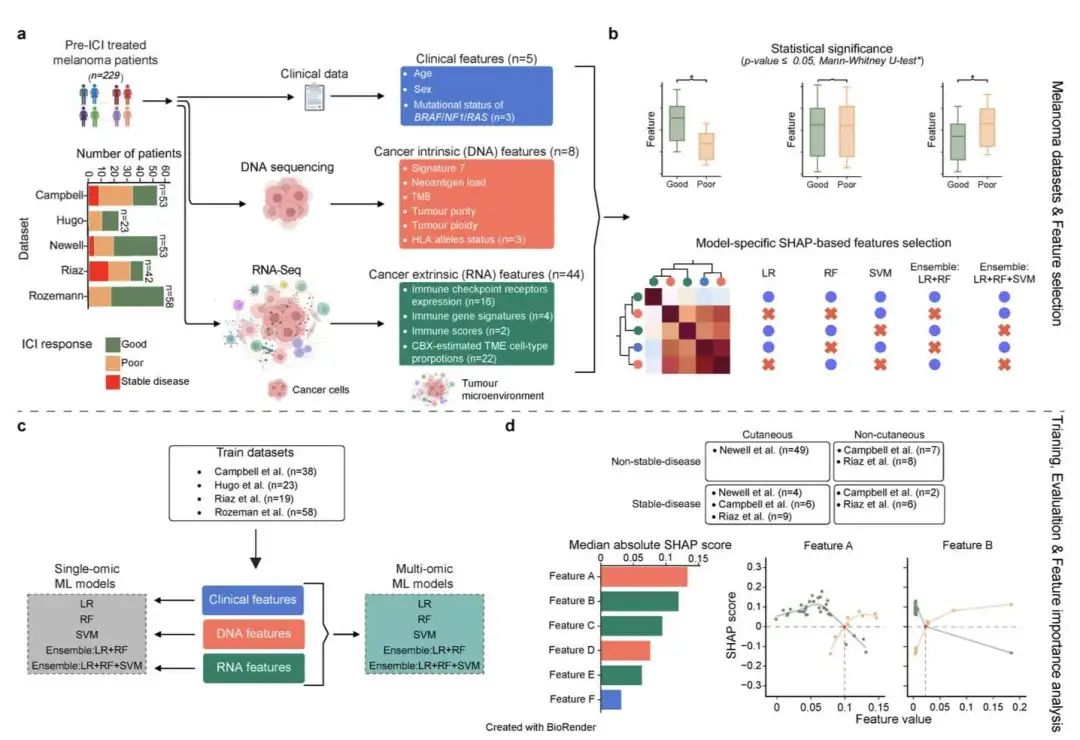
本研究探索了可解释机器学习,特别是 SHAP (Shapley Additive Explanations),在识别预测黑色素瘤患者对免疫检查点抑制剂 (ICI) 响应的关键生物标志物方面的应用。研究人员构建了一个基于临床、DNA 和 RNA 数据的多组学随机森林模型,并在独立数据集上验证其预测准确性,实现了 0.78 的 AUC-ROC。
分析表明,肿瘤突变负荷 (TMB)、新抗原负荷、免疫受体 LAG3 以及多种免疫细胞类型(如滤泡辅助性 T 细胞和 M2 巨噬细胞)是影响 ICI 疗效的最重要特征。其中,M2 巨噬细胞比例高于 30% 与较差的 ICI 响应相关。因此,作者提出将巨噬细胞清除疗法与 ICI 联合应用可能增强治疗效果。
此外,该研究强调了使用 SHAP 衍生的阈值指导治疗决策的潜力。例如,LAG3 被确定为最具预测性的基因标志物,这与 relatlimab 等 LAG3 靶向药物的临床成功相符。
未来研究方向可能包括整合其他数据模式,例如组织病理学数据,以进一步完善预测模型。
📜Paper: https://www.biorxiv.org/content/10.1101/2025.03.23.643560v1
5. UniMoMo:跨领域分子生成模型革新药物设计
UniMoMo 是一种创新性的分子结合剂生成模型框架,它将多肽、抗体和小分子统一到单个模型中,克服了传统模型仅限于单领域的局限性。这种创新方法利用几何潜扩散模型和全原子自编码器,在多个领域创造多样化的分子结构。UniMoMo 使用基于“块”的表示方法,其中块可以是标准氨基酸或分子片段,从而有效地捕捉局部几何形状和不同分子类型之间的可迁移分子相互作用。
该模型的架构包含一个在压缩的潜在空间内运行的几何潜扩散模型,显著提高了训练效率。此外,它采用迭代全原子自编码器以高保真度解码局部几何形状。通过在 PepBench、SAbDab、Crossdocked2020 等数据集上的广泛测试,UniMoMo 展现了令人印象深刻的跨领域泛化能力,其性能优于在单个数据集上训练的模型。
基准测试表明,UniMoMo 在各种指标上都优于领域特定模型,包括对子结构分布的保真度、几何合理性和结合亲和力预测。尤其是在针对 GPCR 等具有挑战性靶点的从头设计中,UniMoMo 在多肽、抗体和小分子的设计方面均取得了优异的性能。与现有最先进的模型相比,它还在恢复率、结构合理性和结合能预测方面取得了显著改进。UniMoMo 的成功突出了统一建模在从头分子设计中的可行性,并展示了利用多样化数据集增强生成性能的潜力,为药物发现和分子结合剂设计的未来发展铺平了道路。

📜Paper: https://arxiv.org/abs/2503.19300v1
参考资料
[1]
DOI: 10.1021/acs.jmedchem.4c03167: https://doi.org/10.1021/acs.jmedchem.4c03167
