【科研绘图系列】R语言绘制生存曲线图(KW plot)
禁止商业或二改转载,仅供自学使用,侵权必究,如需截取部分内容请后台联系作者!
文章目录
-
- 介绍
-
- 数据读取与处理
- 生存分析与图形绘制
- 结果保存
- 加载R包
- 数据下载
- 导入数据
- 数据预处理
- 画图
- 总结
- 系统信息
介绍
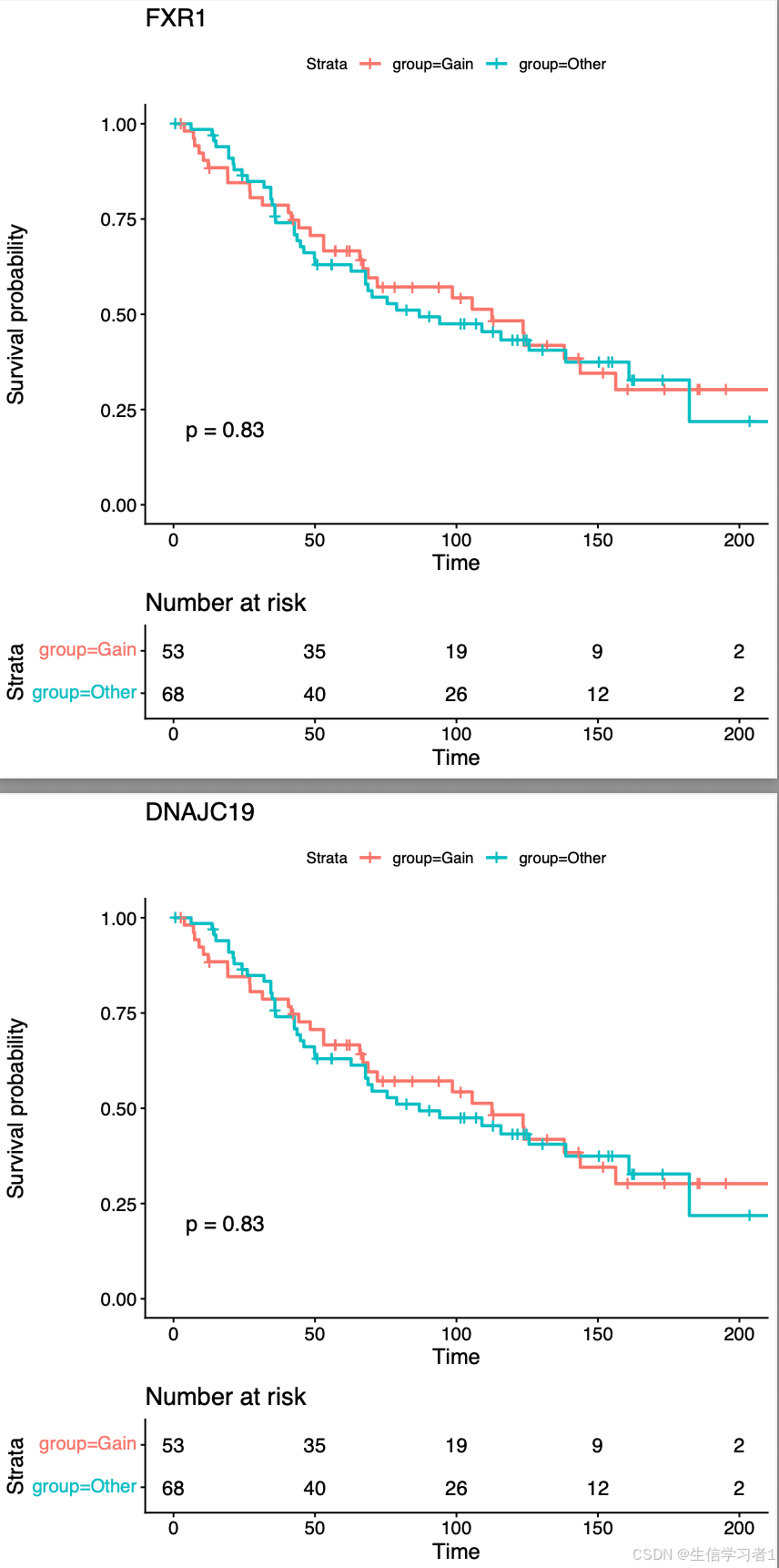
这段代码是一个用于分析基因增益和缺失对生存时间影响的R脚本。它主要利用了survival和survminer等R包,以及一些基础的R函数,对特定的数据文件进行生存分析,并生成相应的生存曲线图和p值结果。
数据读取与处理
代码首先读取了名为Gain_driven.csv和Loss_driven.csv的数据文件,这些文件可能包含了患者的基因增益和缺失信息,以及相关的生存时间数据。数据文件的前几列可能包含了患者的生存时间、生存状态、是否接受利妥昔单抗治疗等基本信息,而从第7列开始则可能包含了不同基因的增益或缺失状态。
在处理数据时,代码首先筛选出了接受利妥昔单抗治疗的患者(RITUXIMAB_ADMINISTERED_PRIMARY_REGIMEN_YES_1_NO_0 == 1),然后根据基因的增益或缺失状态将患者分为两组(“Gain”或“Loss”组,以及“Other”组)。对于基因增益,如果基因状态大于0,则归为“Gain”组;对于基因缺失,如果基因状态小于0,则归为“Loss”组。
生存分析与图形绘制
生存分析是通过survfit()函数完成的,它根据基因的增益或缺失状态对患者的生存时间进行分析。surv_pvalue()函数用于计算生存曲线之间的p值,以评估不同组之间的生存差异是否具有统计学意义。
图形绘制方面,ggsurvplo
