微网站app制作网站开发 视频播放器
今天,我们将通过实际代码演示,手把手教你完成完整的差异基因分析流程。
准备工作
安装所需R包
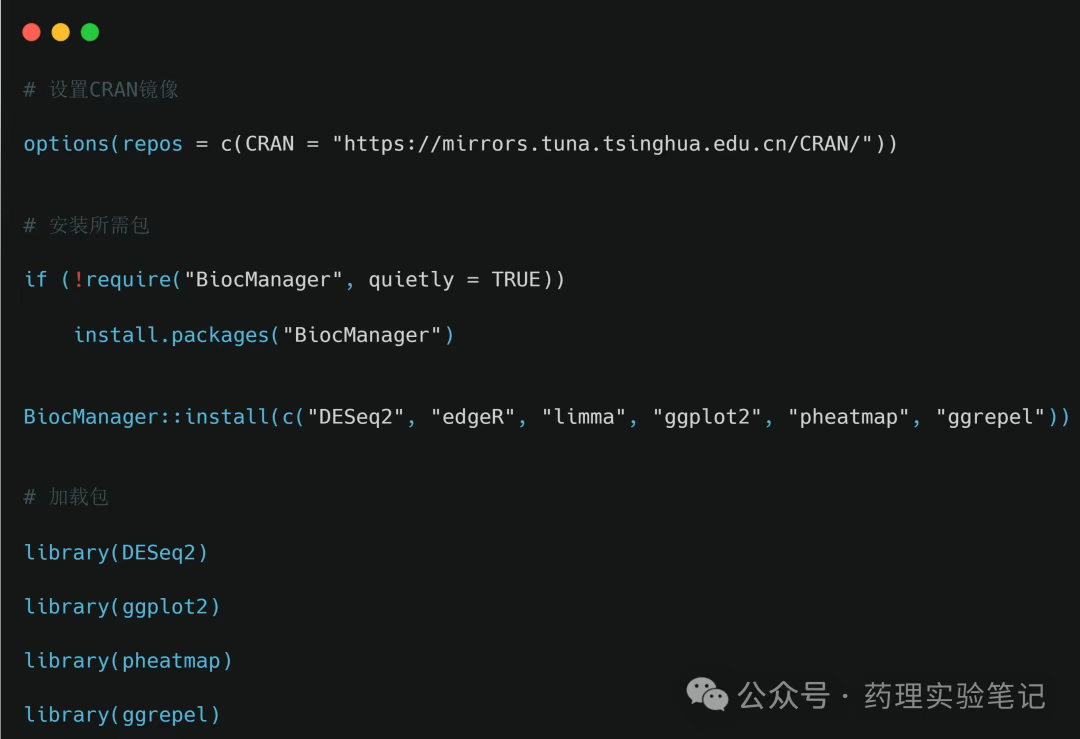
# 设置CRAN镜像
options(repos = c(CRAN = "https://mirrors.tuna.tsinghua.edu.cn/CRAN/"))# 安装所需包
if (!require("BiocManager", quietly = TRUE))install.packages("BiocManager")BiocManager::install(c("DESeq2", "edgeR", "limma", "ggplot2", "pheatmap", "ggrepel"))# 加载包
library(DESeq2)
library(ggplot2)
library(pheatmap)
library(ggrepel)
示例数据准备
我们将使用airway数据集作为示例,这是一个研究气道平滑肌细胞对糖皮质激素反应的RNA-seq数据集
# 加载示例数据
data("airway", package = "airway")# 查看数据基本信息
print(airway)
summary(assay(airway))
DESeq2差异分析完整流程
1. 数据预处理与DESeqDataSet创建
# 提取表达矩阵和样本信息 count_data <- assay(airway) col_data <- colData(airway)# 确保样本信息一致 stopifnot(all(colnames(count_data) == rownames(col_data)))# 创建DESeqDataSet对象 dds <- DESeqDataSetFromMatrix(countD
