Windows下安装Linux子系统Ubuntu
Windows下安装Linux子系统Ubuntu
作者:曾美尹 中国农科院深圳基因组研究所
审核:刘永鑫 中国农科院深圳基因组研究所
更新时间:2025/11/10
Ubuntu是一种免费、开源的Linux系统发行版,它和Windows、macOS一样,都是操作系统,它的编程环境强大稳定,更符合科研需求,在Windows系统中下载了Ubuntu子系统后,可以实现Linux环境和Windows环境的无缝切换。
本文介绍了Windows安装Linux子系统Ubuntu的步骤:启用子系统 —— 微软商店下载安装 —— 设置用户名和密码,随后即可使用;此外,还可在RStudio中将Terminal设置为Bash(Windows Subsystem for Linux),从而可视化地运行子系统。
视频教程
B站链接:https://www.bilibili.com/video/BV1VgCwBiEK7/
Ubuntu安装
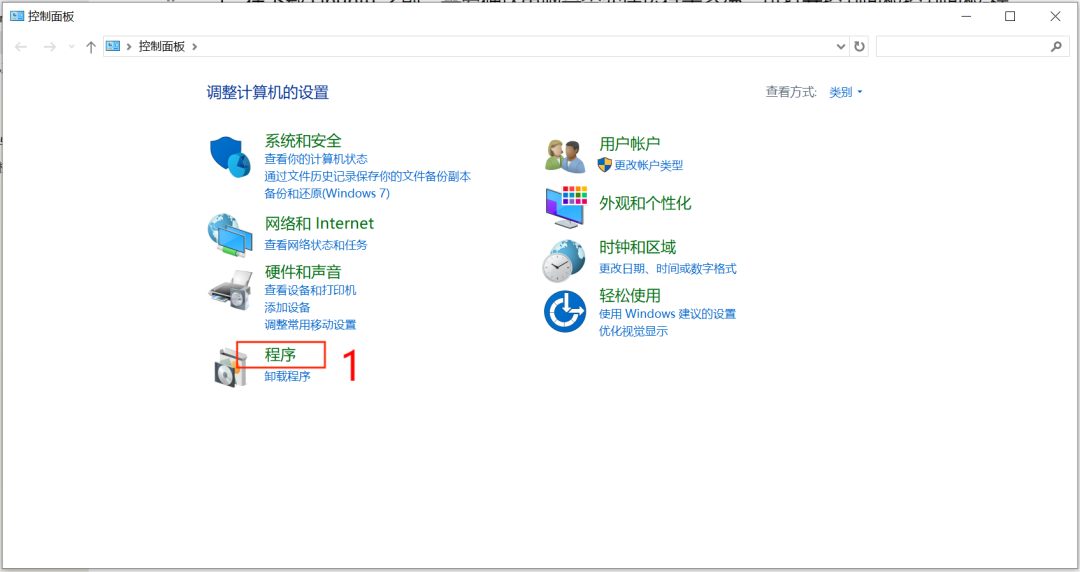
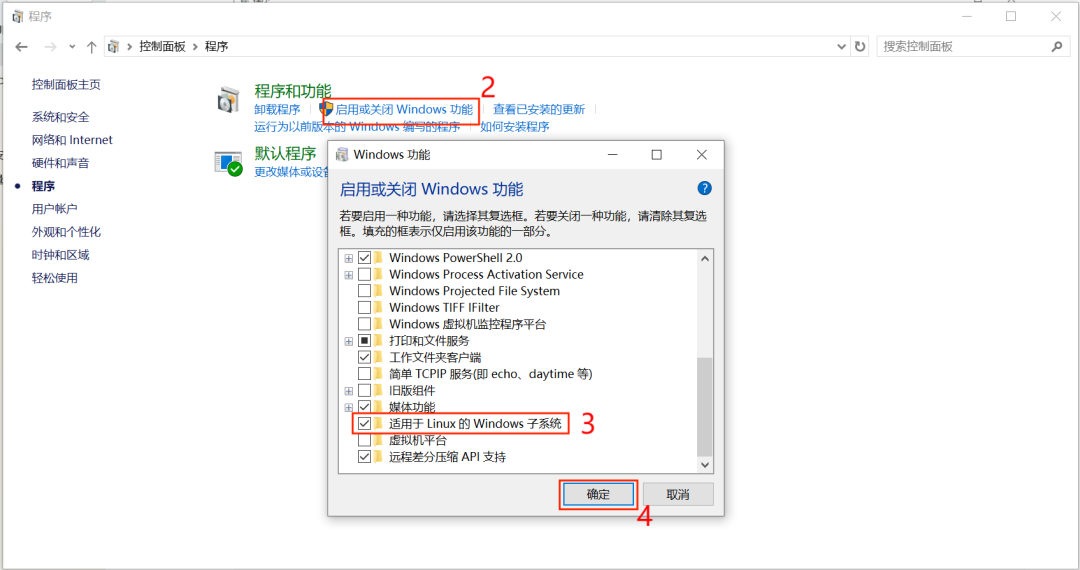
1.设置中启用子系统:在下载Ubuntu之前,需要确认电脑是否能够启用子系统。可打开 控制面板-程序-启用或关闭Windows功能,勾选“适用于Linux的Windows子系统”,然后点击确定,随后重启电脑:
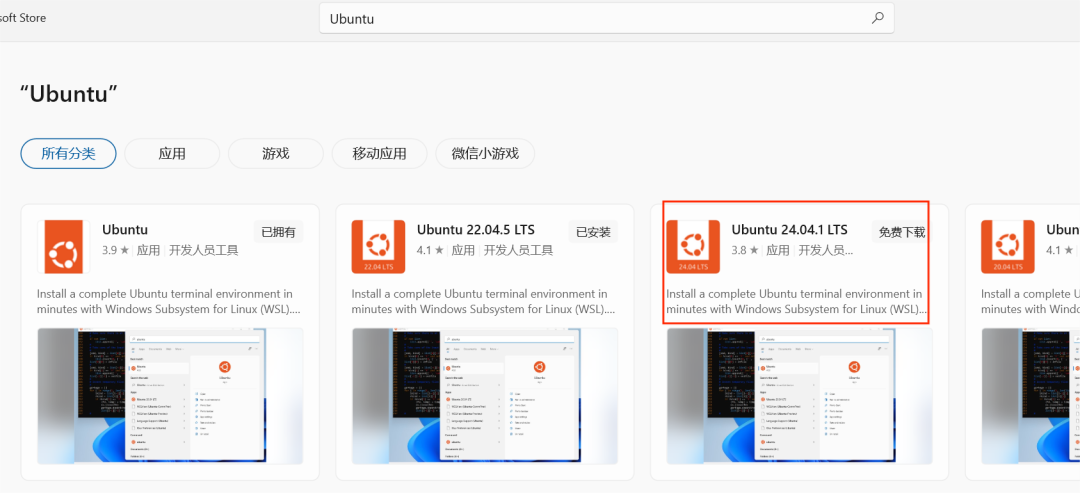
2. Ubuntu下载安装:随后就可以进行Ubuntu的下载了,它可以直接在电脑自带的微软商店进行下载安装,直接在搜索栏搜索ubuntu,选择任意版本均可(这里我选择24.04):
3. root用户设置:安装完成后在开始菜单点击图标即可跳转到Ubuntu中,或者也可以直接在搜索栏搜索Ubuntu打开。打开Ubuntu后按照指示等待配置文件下载完成,再设置好自己的用户名和密码(不要用中文!输密码的时候光标不显示是正常的):
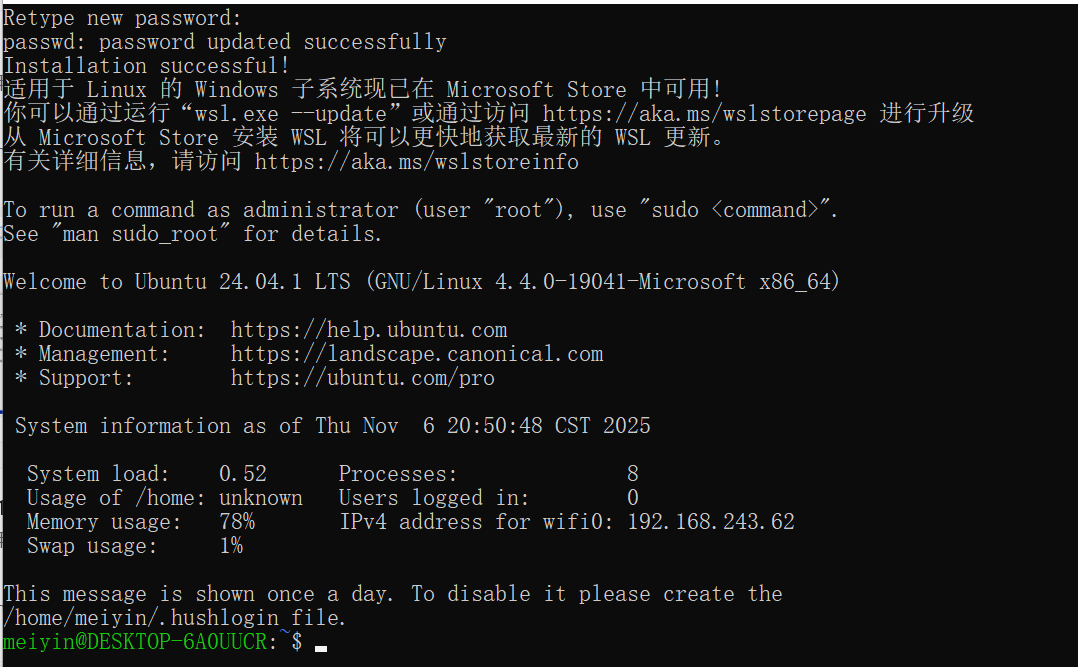
出现这段绿色的字符串就说明成功创建了用户并且已经进入了Linux环境,随后就可以在Ubuntu中运行Linux命令了。
在RStudio中使用Ubuntu
1. 在RStudio中操作Ubuntu:平常操作子系统时,我们除了可以直接在Ubuntu界面运行指令,也可以通过RStudio进行操作。如果你的电脑中刚好安装了RStudio,那么只需要将Tools —— Global Options —— Terminal处的New terminals open with切换为Bash(Windows Subsystem for Linux),随后在操作台打开一个New terminal就好了;
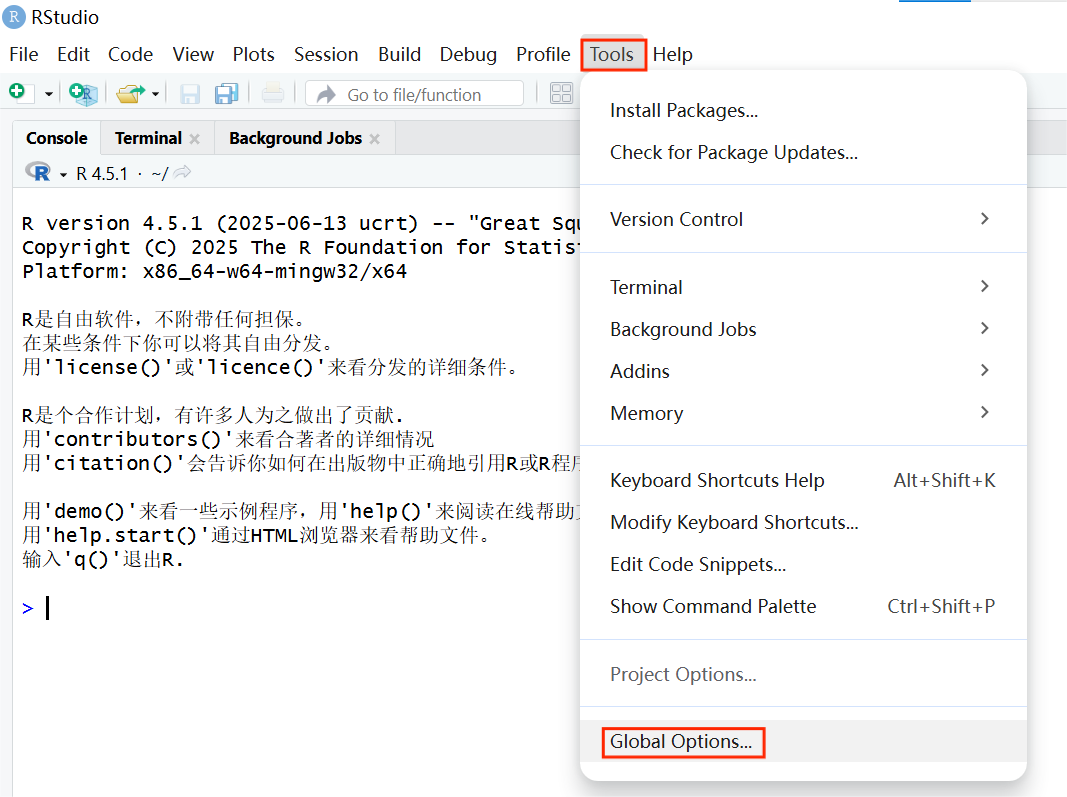
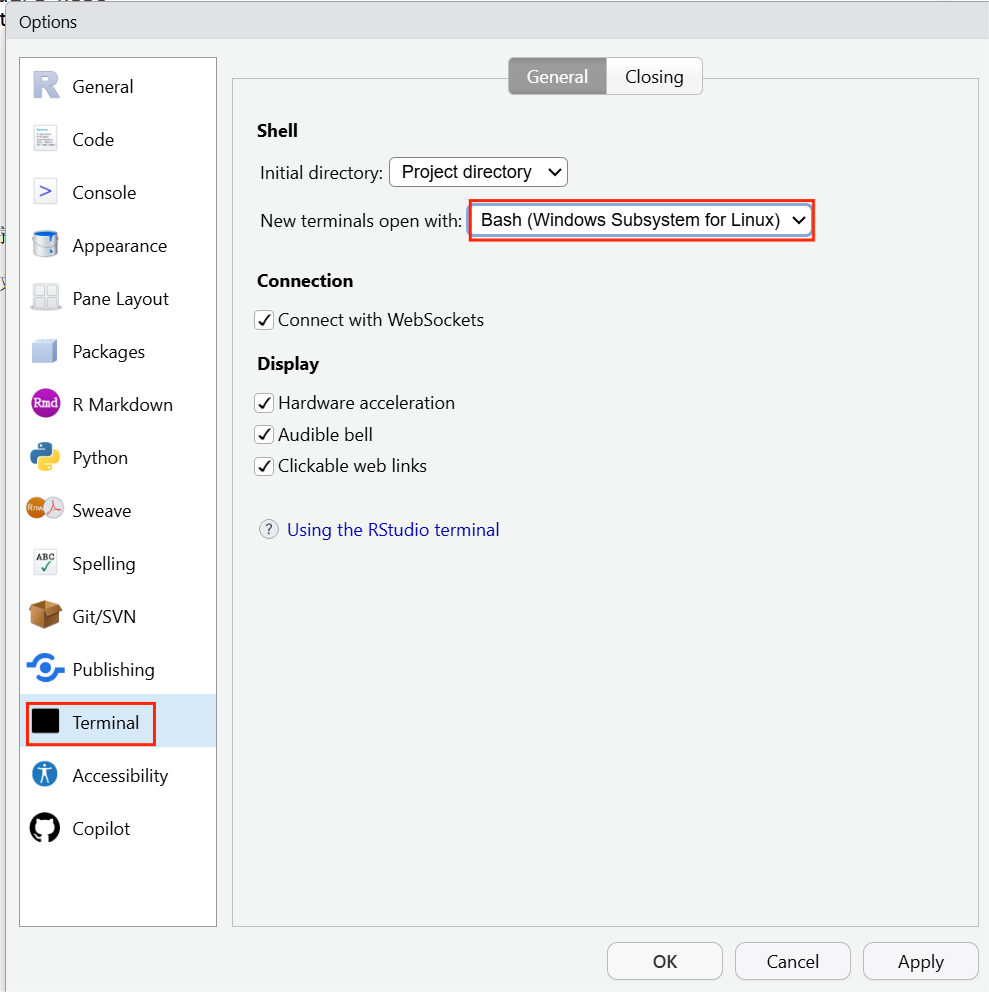
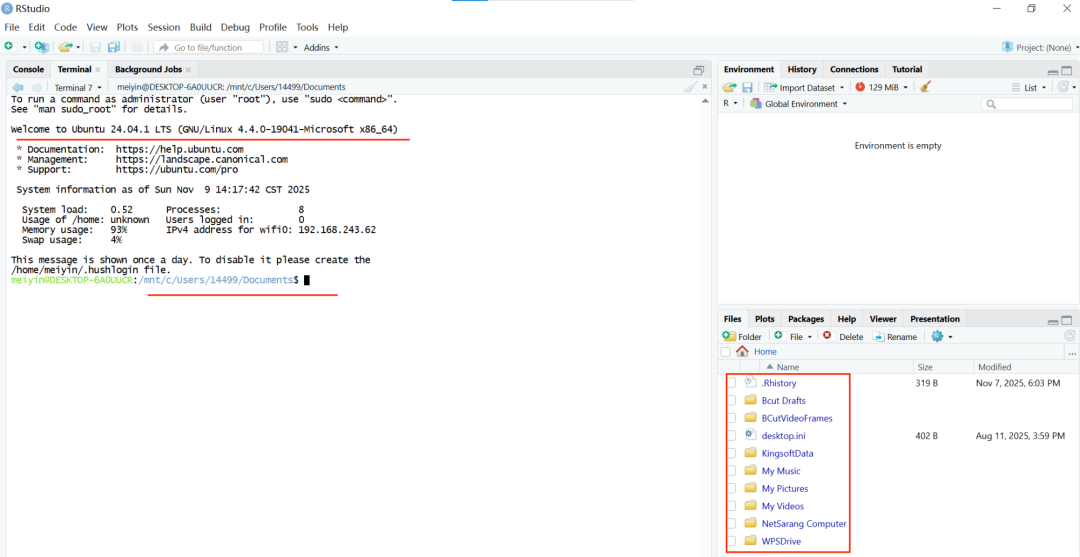
切换New Terminal后可以直接在该terminal下输入Linux命令,与直接使用Ubuntu没有区别,只是RStudio提供了更加可视化的界面,可在Files栏直接观察文件状态
宏基因组推荐
9月12-14日,高级转录组分析和R语言数据可视化
10月18-19日,微生物组-扩增子16S分析
11月15-16日,微生物组-宏基因组分析
本公众号现全面开放投稿,希望文章作者讲出自己的科研故事,分享论文的精华与亮点。投稿请联系小编(微信号:yongxinliu 或 meta-genomics)
iMeta高引 fastp PhyloSuite ImageGP2 iNAP2 ggClusterNet2
iMeta工具 SangerBox2 美吉2024 OmicStudio Wekemo OmicShare
iMeta综述 高脂饮食菌群 发酵中药 口腔菌群 微塑料 癌症 宿主代谢
10000+:扩增子EasyAmplicon 比较基因组JCVI 序列分析SeqKit2 维恩图EVenn
iMetaOmics高引 猪微生物组 16S扩增子综述 易扩增子(EasyAmplicon)
系列教程:微生物组入门 Biostar 微生物组 宏基因组
专业技能:学术图表 高分文章 生信宝典 不可或缺的人
